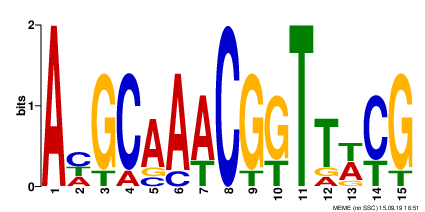
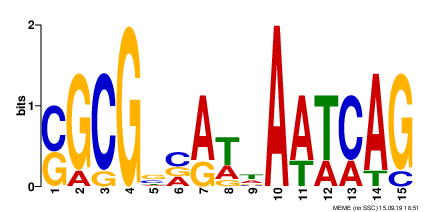
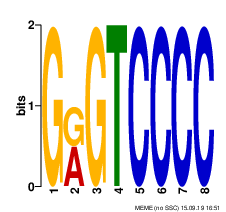
Для работы мною был выбран организм Escherichia coli (strain ECO57).
На сайте Uniprot был произведен поиск с помощью Keyword: "Purine biosynthesis"
по нужному нам организму.
Затем из 12 аннотированных белков были выбраны 9 (Таблица 1).
| Идентификатор белка | Мнемоника белка | Название белка | Название гена | Координаты |
| Q8XCJ9 | PURT_ECO57 | Formate-dependent phosphoribosylglycinamide formyltransferase | purT | 2532849..2534027 |
| P0ADG8 | IMDH_ECO57 | Inosine-5'-monophosphate dehydrogenase, IMP dehydrogenase, IMPD, IMPDH, EC 1.1.1.205 | guaB | complement(3348881..3350347) |
| P0A7D6 | PURA_ECO57 | Adenylosuccinate synthetase, AMPSase, AdSS, EC 6.3.4.4 | purA | 5258540..5259838 |
| Q8XA46 | PUR4_ECO57 | Phosphoribosylformylglycinamidine synthase, FGAM synthase, FGAMS, EC 6.3.5.3 | purL | complement(3412175..3416062) |
| Q8XCT6 | FOLD_ECO57 | Bifunctional protein FolD [Includes: Methylenetetrahydrofolate dehydrogenase, EC 1.5.1.5; Methenyltetrahydrofolate cyclohydrolase, EC 3.5.4.9] | folD | complement(649328..650194) |
| Q8X612 | PUR2_ECO57 | Phosphoribosylamine--glycine ligase, EC 6.3.4.13 | purD | complement(5013573..5014862) |
| P64295 | GUAA_ECO57 | GMP synthase [glutamine-hydrolyzing], EC 6.3.5.2 | guaA | complement(3347235..3348812) |
| Q8X611 | PUR9_ECO57 | Bifunctional purine biosynthesis protein PurH [Includes: Phosphoribosylaminoimidazolecarboxamide formyltransferase, EC 2.1.2.3 | purH | complement(5014874..5016463) |
| Q8XAC5 | PUR5_ECO57 | Phosphoribosylformylglycinamidine cyclo-ligase, EC 6.3.3.1 | purM | 3337475..3338512 |
Далее, с помощью базы данных EMBL была найдена запись полного генома
Escherichia coli O157:H7 str. Sakai
(BA000007.3).
Для всех генов выбранных белков были найдены координаты Upstream-региона из 100 нуклеотидов.
С помощью команд из пакета EMBOSS (extractfeat, descseq, extractseq, seqret) были получены
последовательности нужных Upstream-регионов и записаны в единый fasta файл
(result.fasta).
Для поиска мотивов была использована команда ememe программы MEME.
Параметры: -revcomp, -nmotifs 3 (-revcomp проводит поиск на прямой и обратной цепях ДНК;
-nmotifs задает число мотивов для поиска).
Результат выдачи программы: meme.html.
Информация о найденных мотивах для удобства была собрана в Таблицу 2.
| № | Мотив 1. | Мотив 2. | Мотив 3. |
| Информативность | 19.1 | 16.6 | 15.0 |
| Энтропия | 19.1 | 16.7 | 15.6 |
| Длина мотива | 15 | 15 | 8 |
| Число белков, в которых встретился мотив | 7 | 7 | 2 |
| E-value | 2.5e-001 | 1.0e+003 | 2.2e+004 |
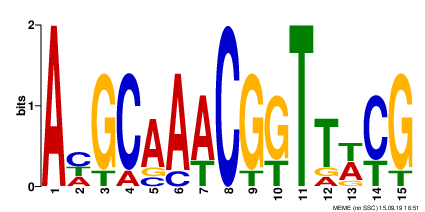 |
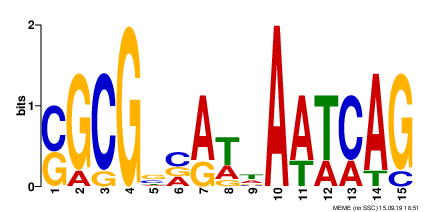 |
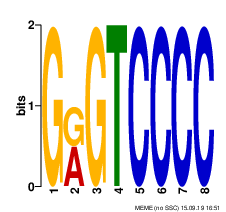 |
| Мотив 1. | Мотив 2. | Мотив 3. |
По итогу можно сказать, что ни один из найденных мотивов не является хорошим (нужно E-value < 0.001). Однако лучшим из представленных можно назвать 1 мотив (так как значение E-value для него минимальное, а значения информативности и энтропии максимальные).
© Наумова Юлия, 2019