UniProt
(S)-1-Phenylethanol dehydrogenase
Введение
Фермент (S)-1-фенилэтанол дегидрогеназа был найден в организме Aromatoleum aromaticum Azoarcus sp. (strain EbN1), бактерии имеющей только один штамм. Штамм EbN1-это разлагающая ароматические соединения бактерия, она встречается в пресноводных и почвенных средах обитания. Уникальным свойством штамма EbN1 является его способность разлагать толуол и этилбензол двумя различными анаэробными способами.[1]
(S)-1-фенилэтанол дегидрогеназа из денитрифицирующего штамма бактерий EbN1 катализирует реакцию окисления (S)-1-фенилэтанола до ацетофенона и биотехнологически интересную обратную реакцию. Фермент состоит в семействе алкогольдегидрогеназ. Кодирующий ген был экспрессирован, и очищенный белок был кристаллизован. Рентгеновские структуры апо-формы и НАД+-связанной формы были проанализированы с разрешением 2,1 и 2,4 А соответственно, что показало, что фермент представляет собой тетрамер с двумя типами гидрофобных интерфейсов димеризации, аналогичными бета-оксоацил-[ацил-несущий белок] редуктазе.[2]
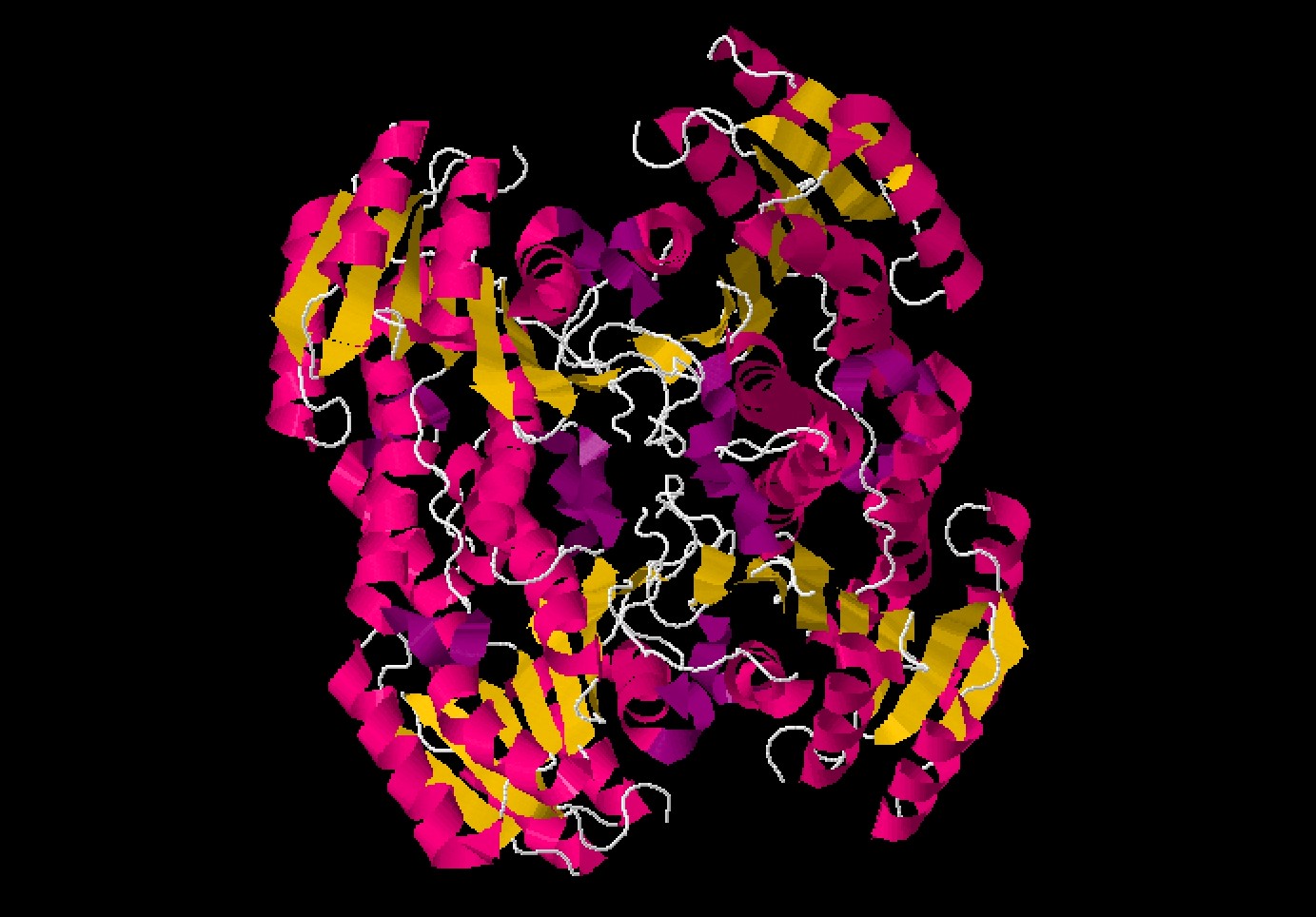
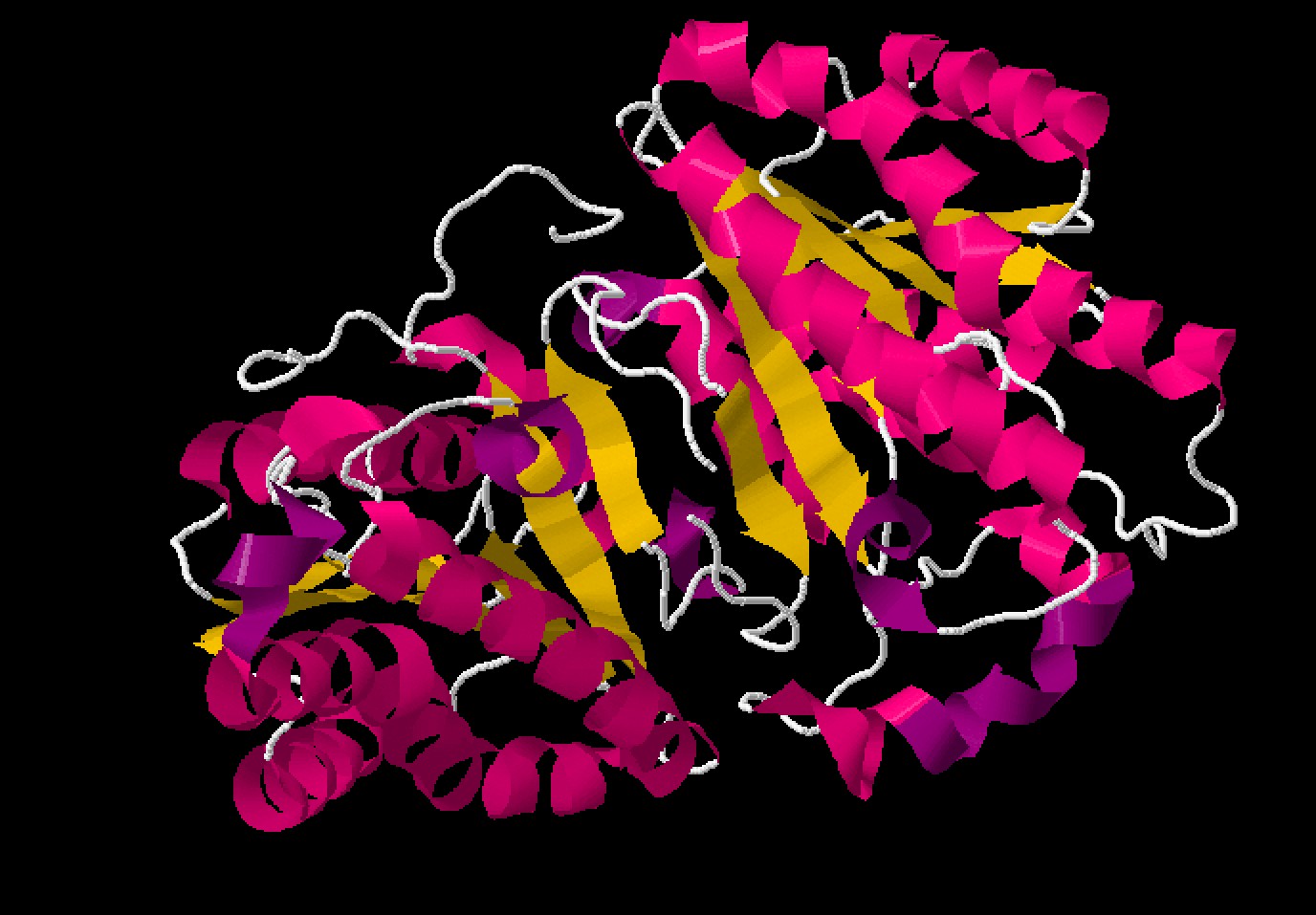
Моделирование введения субстрата ацетофенона в активный центр выявило структурные предпосылки сильной энантиоселективности фермента и каталитического механизма, то есть была выявлена совместимость для реакции.[3]
Характерестики (S)-1-фенилэтанол дегидрогеназы
| Тип характеристики | Характерестика |
| UniProt ID | PED_AROAE |
| UniPrit AC | Q5P5I4 |
| RecName | (S)-1-Phenylethanol dehydrogenase |
| PDB ID | 2EW8,2EWM |
| EMBL | CR555306 |
| Длина в аминокислотных остатках | 249 AA/249 аминокислот |
| Молекулярная масса(MW) | 26662 MW |
В UnoProt для всего белка известна структура. Строки "DR PDB; 2EW8; X-ray; 2.10 A; A/B/C/D=1-249." и "SQ SEQUENCE 249 AA" показывают, что изучены с 1-ой по 249-ую аминокислоты.
Поиск
| Поле поиска | Количество результатов |
| name:"s -1-phenylethanol dehydrogenase" | 294 |
| name:"s -1-phenylethanol dehydrogenase" taxonomy:rhodocyclales | 2 |
| name:"s -1-phenylethanol dehydrogenase" taxonomy:rhodocyclales organism:"aromatoleum aromaticum strain ebn1" | 2 |
| name:"s -1-phenylethanol dehydrogenase" length:[249 TO 249] | 11 |
| name:"s -1-phenylethanol dehydrogenase" organism:human | 0 |
| name:"s -1-phenylethanol dehydrogenase" organism:mouse | 0 |
Изменения вносимые в описание со временем
2012-09-05 был обнаружен инициирующий биосинтез метеонин.
Полную последовательность удалось описать при самой первой записи.
Особенность последовательности
В структуре будет 12 a-спиралей(helix). b-тяжей(strand) 8.
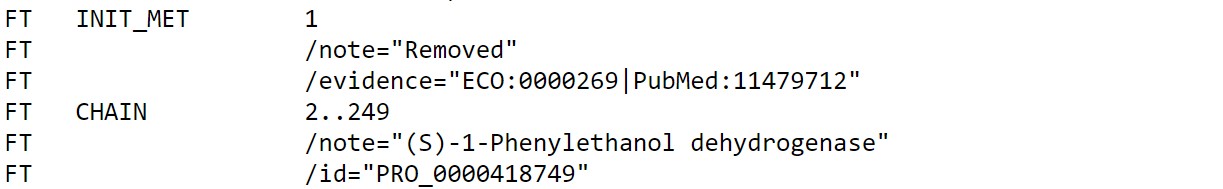
С инициирующего метеонина "INIT_MET" начинается биосинтез фермента.
Далее метеонин отщипляется, это показывается в строке "CHAIN" диапазоном 2-249.
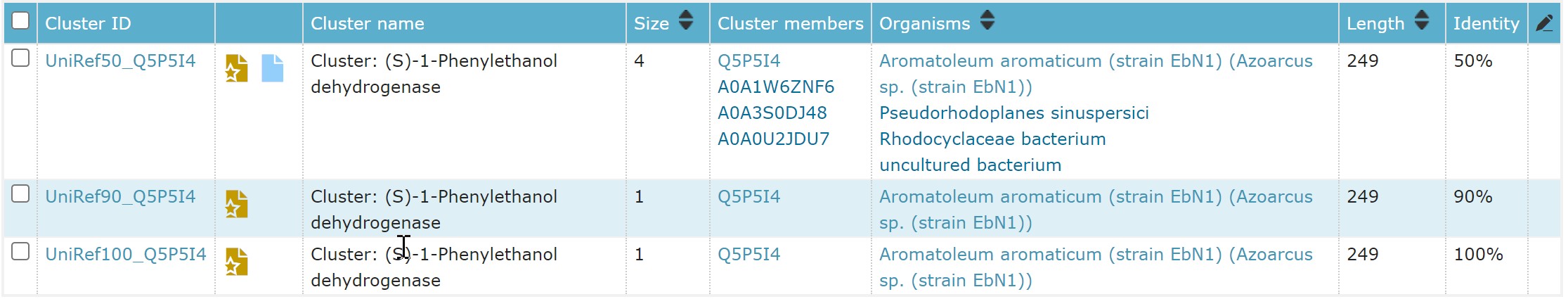
Кластеры UniRef бактерии Aromatoleum aromaticum (strain EbN1)
Кластеры UniRef
Есть кластеры UniRef50_Q5P5I4(в кластере таких 4 белка); UniRef90_Q5P5I4(в кластере только один белок);
UniRef100_Q5P5I4(в кластере один белок). По размеру кластеров можно сделать вывод, что белок не распространён.
Если взять 50% идентичности, то получим только 3 схожих белка.
При 90% идентичности нет схожих белков.
Сравнение Aromatoleum aromaticum (strain EbN1) и Azospira oryzae (strain ATCC BAA-33 / DSM 13638 / PS)
Протеом бактерия Aromatoleum aromaticum (strain EbN1) сам является референсным. В роде Ароматолеум известен только этот штамм, поэтому можно взять для сравнения референсный протеом близкой бактерии из семейства Rhodocyclaceae. Была взята бактерия Azospira oryzae (strain ATCC BAA-33 / DSM 13638 / PS). В обоих протеомах изучено 413 белков, в бактерии Aromatoleum aromaticum Azoarcus sp. (strain EbN1) изученность протеома составляет 9,21%, а в бактерии Azospira oryzae (strain ATCC BAA-33 / DSM 13638 / PS) 12,03%.
| Функциональная группа | Aromatoleum aromaticum Azoarcus sp. (strain EbN1) | Azospira oryzae (strain ATCC BAA-33 / DSM 13638 / PS) |
| Proteom ID | UP000006552 | UP000005633 |
| Количество белков | 4483 | 3432 |
| Количество белков в Swiss-Prot | 413(9,21%) | 413(12,03%) |
| Трансмембранный белок | 698 | 698 |
| Фермент | 891 | 714 |
| Азотофиксация | 0 | 18 |
Отличием единственного изученного штамма бактерии Aromatoleum aromaticum Azoarcus sp. (strain EbN1) является отсутствие азотофиксации, отделяющее EBN1 от остальных близкородственных азотфиксирующих растительных симбионтов кластера Azoarcus. Это подтверждается результатами запросов. У бактерии Aromatoleum aromaticum Azoarcus sp. (strain EbN1) нет белков отвечающих за фиксацию азота(Запрос показал один белок, но он не отвечает за фиксацию азота, хоть и связан с азотом). У бактерии Azospira oryzae (strain ATCC BAA-33 / DSM 13638 / PS) было получено 18 белков.
Использовавшиеся команды
- Трансмембранные белки
- annotation:(type:transmem) AND organism:"Aromatoleum aromaticum (strain EbN1) (Azoarcus sp. (strain EbN1)) [76114]" AND proteome:up000006552
- annotation:(type:transmem) AND organism:"Azospira oryzae (strain ATCC BAA-33 / DSM 13638 / PS) (Dechlorosoma suillum) [640081]" AND proteome:up000005633
- Ферменты
- ec:* AND organism:"Aromatoleum aromaticum (strain EbN1) (Azoarcus sp. (strain EbN1)) [76114]" AND proteome:up000006552
- ec:* AND organism:"Azospira oryzae (strain ATCC BAA-33 / DSM 13638 / PS) (Dechlorosoma suillum) [640081]" AND proteome:up000005633
- Азотофиксация
- "nitrogen fixation" AND organism:"Aromatoleum aromaticum (strain EbN1) (Azoarcus sp. (strain EbN1)) [76114]" AND proteome:up000006552
- "nitrogen fixation" AND organism:"Azospira oryzae (strain ATCC BAA-33 / DSM 13638 / PS) (Dechlorosoma suillum) [640081]" AND proteome:up000005633
- Swiss-Prot
- reviewed:yes AND organism:"Aromatoleum aromaticum (strain EbN1) (Azoarcus sp. (strain EbN1)) [76114]" AND proteome:up000006552
- "nitrogen fixation" AND organism:"Azospira oryzae (strain ATCC BAA-33 / DSM 13638 / PS) (Dechlorosoma suillum) [640081]" AND proteome:up000005633