Сборка de novo
Подготовка чтений программой trimmomatic
Скачивание архива с чтениями в рабочую директорию было сделано с помощью команды:
wget ftp://ftp.sra.ebi.ac.uk/vol1/fastq/SRR424/008/SRR4240378/SRR4240378.fastq.gz.
Адаптеры были собраны командой: cat /mnt/scratch/NGS/adapters/* >> adapters.fasta.
Адаптеры были удалены командой: java -jar /usr/share/java/trimmomatic.jar SE SRR4240378.fastq.gz trimmed.fastq.gz -trimlog trim.log ILLUMINACLIP:adapters.fasta:2:7:7.
Было удалено 1.85% чтений.
После этого были удалены с правых концов чтений нуклеотиды с качеством ниже 20 и чтения длиной меньше 32: java -jar /usr/share/java/trimmomatic.jar SE trimmed.fastq.gz SRR4240378_trimmed2.fastq.gz -trimlog trim_2.log TRAILING:20 MINLEN:32.
Это удалило 4.24% чтений, сохранились 95.76% чтений. Исходный файл размером 91Mб стал весить после очистки 84Мб.
Получение k-меров
Командой velveth мы создаем k-меры указанной длины: velveth k_mers_31 31 -fastq -short SRR4240378_trimmed2.fastq.gz &> velveth.log.
Командой velvetg мы собираем контиги на основе k-меров, созданных командой velveth: velvetg k_mers_31 &> velvetg.log. N50 составил 7028.
Самые длинные контиги были выделены командой: sort -n -k 2 -r k_mers_31/stats.txt | head.
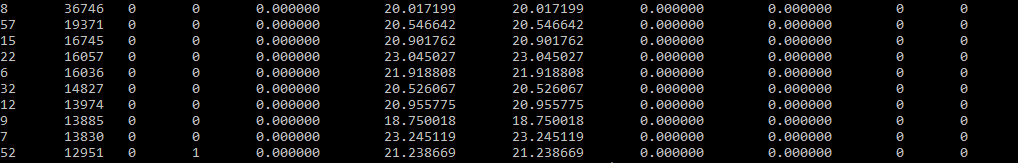
Контиги с аномально большим покрытием получены командой: sort -n -k 6 -r k_mers_31/stats.txt | head -n 3.

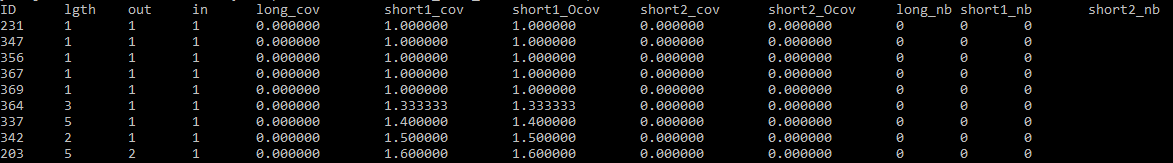
Контиги с аномально маленьким покрытием получены командой: sort -n -k 6 k_mers_31/stats.txt | head -n 10.
С помощью команды seqret достаем последовательности наибольших контигов:
seqret k_mers_31/contigs.fa:'*length_36746*' contig8.fasta
seqret k_mers_31/contigs.fa:'*length_19371*' contig57.fasta
seqret k_mers_31/contigs.fa:'*length_16745*' contig15.fasta
Анализ контигов
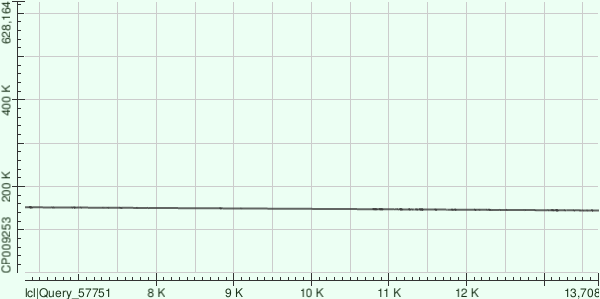
Контиг 8
Общий вес выравнивания составляет 13826. Контиг ложится на участок хромосомы с 500370 по 508806. При проценте покрытия 73%, процент идентичности 75.62%. Есть шесть разрывов. Ссылка на выдачу.

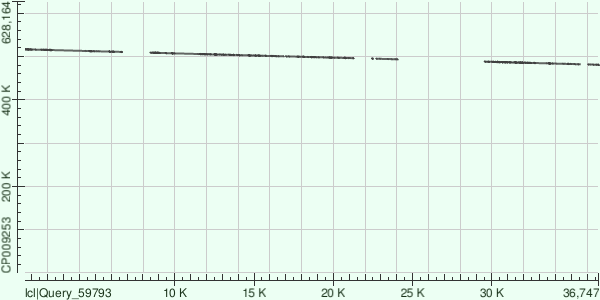
Контиг 57
Общий вес выравнивания составляет 4532. Контиг ложится на участок хромосомы с 573092 по 582686. При проценте покрытия 63%, процент идентичности 73.43%. Есть один разрыв. Ссылка на выдачу.

Контиг 15
Общий вес выравнивания составляет 4423. Контиг ложится на участок хромосомы с 144368 по 151796. При проценте покрытия 44%, процент идентичности 77.80%. Разрывов нет. Ссылка на выдачу.