Трансмембранные белки
База данных OPM
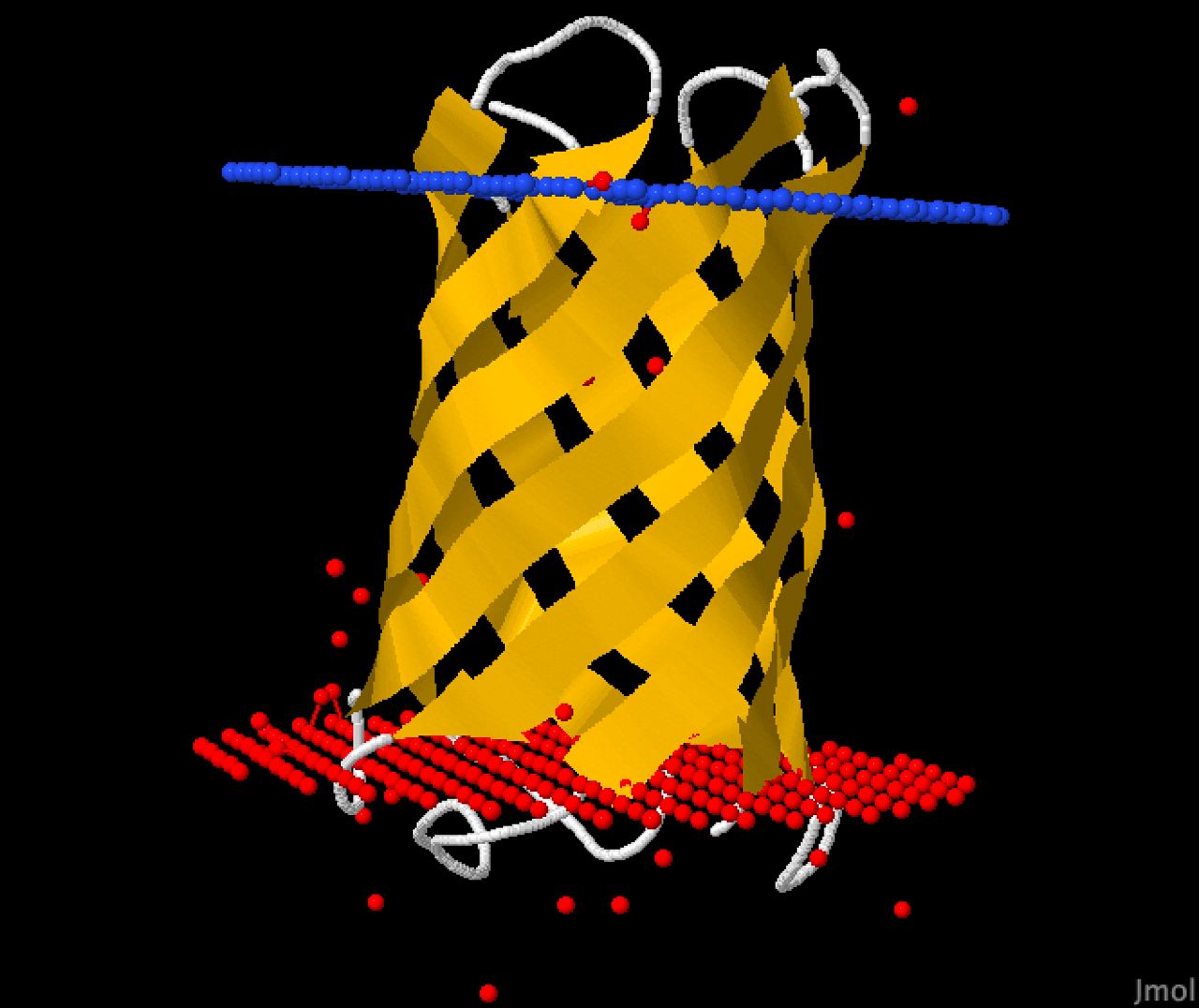
В базе данных OMP был выбран 3qra - Attachment invasion locus protein(Белок внешней мембраны).
AC в UniProt: Q8D0Z7; идентификатор PDB 3qra.
| Тип белка | Трансмембранный белок, бета-бочонок |
| Суперсемейство | OmpA-OmpF porin |
| Семейсво | Enterobacterial Ail/Lom protein |
| Организм | Yersinia pestis(Чумная палочка) |
| Локализация | Внешняя мембрана Грам-отрицательной бактерии |
| Uniprot ID | Q8D0Z7_YERPE |
| Толщина гидрофобного слоя | 25.2 Å |
| Число трансмембранных структур | 8 |
| Средняя длина трансмембранных структур | 9 |
DeepTMHMM: Предсказание трансмембранных элементов по последовательности белка
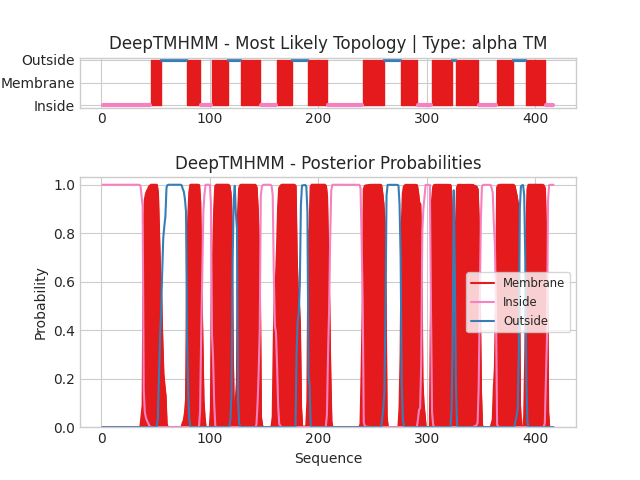
С помощью сервиса DeepTMHMM был проведен анализ выданного белка P43531 и выбранного для первого задания белка Q8D0Z7.
Сервис DeepTMHMM позволяет предсказывать трансмемранные участки белка по первичной структуре.
На схемах показано какие участки белка находятся внутри мембраны (membrae), с внутренней стороны мембраны (inside) и с внешней стороны мембраны (outside). По вертикали на нижней схеме рисунка отмечено предсказание положения участков белка, по горизонтали - координаты остатков белка, а на нижней схеме - вероятность с которой остаток принадлежит той или иной топологии. Программа предсказала 12 трансмембранных участков. N и C концы находятся с внутренней стороны мембраны.
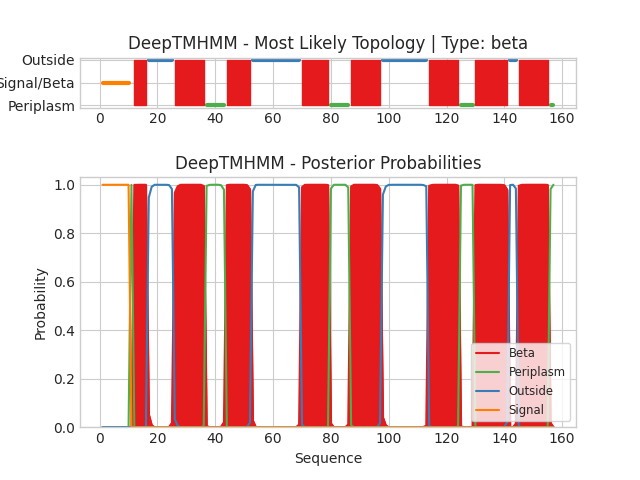
На схеме цвета обозначают где находится участок: Beta - бета-слой, рeriplasm - периплазматический слой, Outside - внеклеточный, Signal - сигнальный пептид. По вертикали на нижней схеме рисунка отмечено предсказание положения участков белка, по горизонтали - координаты остатков белка, а на нижней схеме - вероятность с которой остаток принадлежит той или иной топологии. Для этого белка предсказано 8 трансмембранных участков. N-конец является сигнальным. С-конец находится в периплазматическом слое.
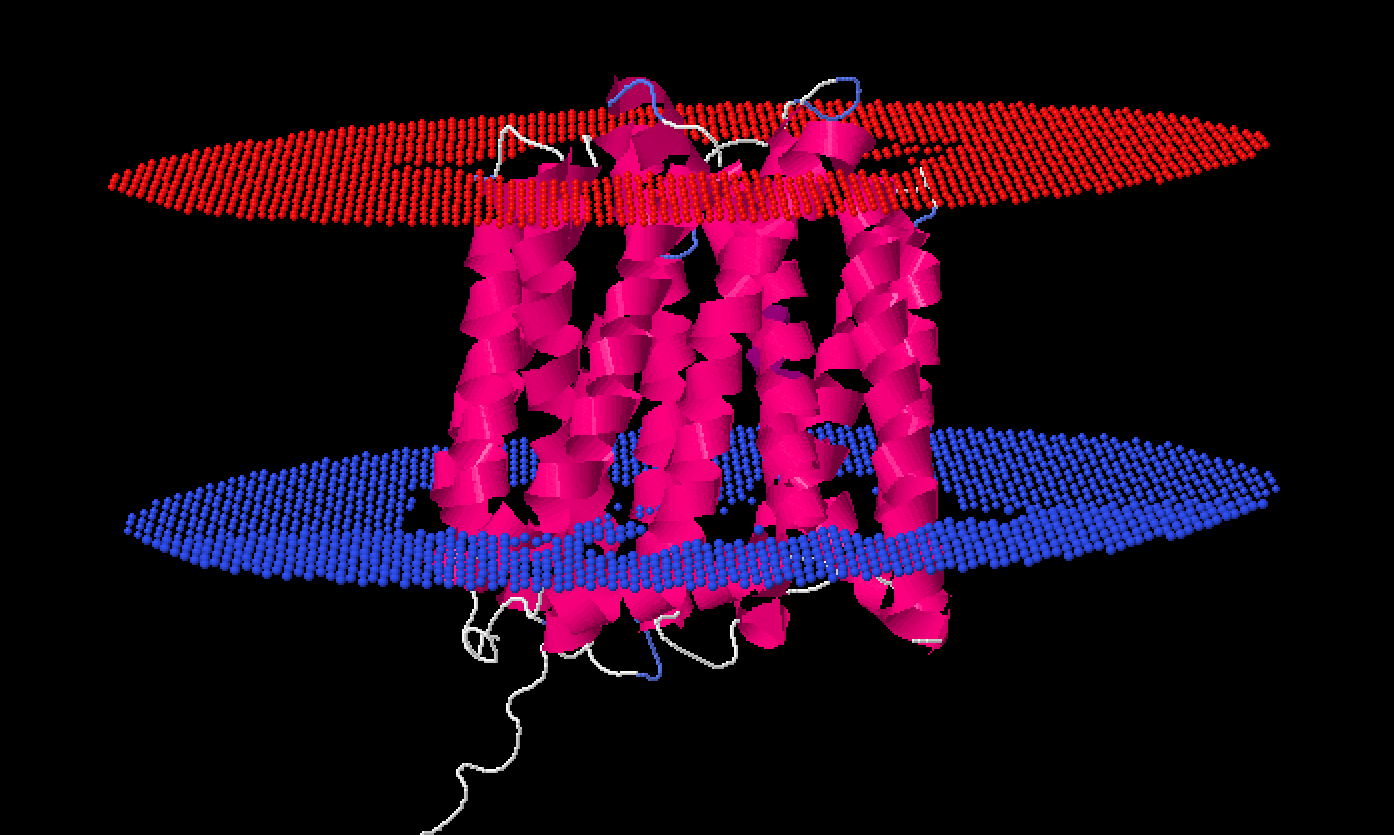
PPM: Предсказание положения выданного белка в мембране
Был использован выданный мне белок с AC в UniProt P43531. В соответствии с данными, полученными в предыдущем задании параметр Topology (N-ter) был установлен in.
| ΔGtransfer | -84.0 kcal/mol |
| Толщина гидрофобного слоя | 29.6 ± 2.1 Å |
| Угол изгиба структур | 13 ± 1° |
| Число трансмембранных структур | 13 |
| Трансмембранные сегменты | 1( 39- 60), 2( 70- 92), 3( 101- 120), 4( 125- 144),
5( 165- 188), 6( 189- 210), 7( 241- 265), 8( 274- 291), 9( 307- 322),10( 327- 345),11( 364- 385),12( 390- 410) |
| Средняя длина трансмембранного сегмента | 20 |
Сравнение алгоритмов предсказания трансмембранных спиралей
Предсказания для YNFM_ECOLI схожи за исключением нескольких участков. Алгоритмы одинаково оценили количество трансмембранных участков. DeepTMHMM и PPM определили почти одни и те же участки.
Изходя из AlphaFold структура белка предсказана достаточно точно.
DeepTMHMM
1(39-52), 2(79-91), 3(102-120), 4(126-146), 5(159-179), 6(191-210), 7(242-262), 8(277-295), 9(304-324), 10(327-349), 11(365-385), 12(392-411)
PPM
1(39-60), 2(70-92), 3(101-120), 4(125-144), 5(165-188), 6(189-210), 7(241-265), 8(274-291), 9(307-322), 10(327-345), 11(364-385), 12(390-410)
Гораздо больше разхождений в предсказании трансмембранных спиралей Q8D0Z7. DeepTMHMM справился заметно хуже. Ни один участок не был предсказан верно.
DeepTMHMM
1(12-16), 2(26-36), 3(44-52), 4(70-79), 5(87-97), 6(114-124), 7(130-141), 8(145-155)
PPM
1(29-37), 2(54-62), 3(69-74), 4(98-106), 5(113-119), 6(141-150), 7(157-162), 8(173-180)

Результаты DeepTMHMM и данные PPM 3.0 отличаются, т.к. DeepTMHMM и PPM 3.0 имеют разное начало отсчета, т.е. данные из PPM начинают отсчет с первого а. о. первого бета-листа, в то время как DeepTMHMM учитывает начальную сигнальную последовательность в 25 а.о.
DeepTMHMM делает предсказание только на основе свойств а. о. и не имеет никаких "представлений" об особенностях различных мембран, а PPM предсказывает трансмембранные участки а основе "встраивания" белка в определенную мембрану (в данном случае мембрана грамположительной бактерии). Этим можно объяснить небольшие различия в границах трансмембранных участков, предсказанных этими методами.
База данных TCDB
В базе данных TCDB выбранного мной белка АС: Q8D0Z7 не оказалось.
Найденный P43531 . Код: 2.A.1.36.1
2 - электрохимический потенциалзависимый транспортер
А - транспортный белок
1 - Фасилитаторы
36.1 - код белка