|
|
|
|
|
|
|
|
|
|
|
 |
|
||||||||
|
|
|
|
|
|
|
|
|
||
|
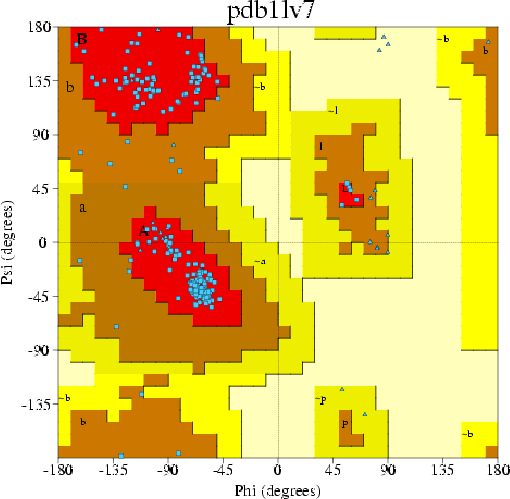
Качество структуры: PROCHECK, EDS, PDB_REDO Рассмотрим pdb файл заданного белка 1LV7 Проанализируем выдачу программы PROCHECK для своей записи PDB. Для этого воспользуйтесь базой данных PDBSum. В выдаче программы предствалена карта Рамачандрана
Предпочитаемые области [A,B,L] --- 93.3% Кончевые остатки (excl. Gly and Pro) 4 Остатки глицина 26
Основываясь на данные карты Рамачандрана, можно сделать вывод,
что данная модель хорошая, так как больше 90% остатков, отличных от глицина и пролина, находятся
в предпочитаемой области.
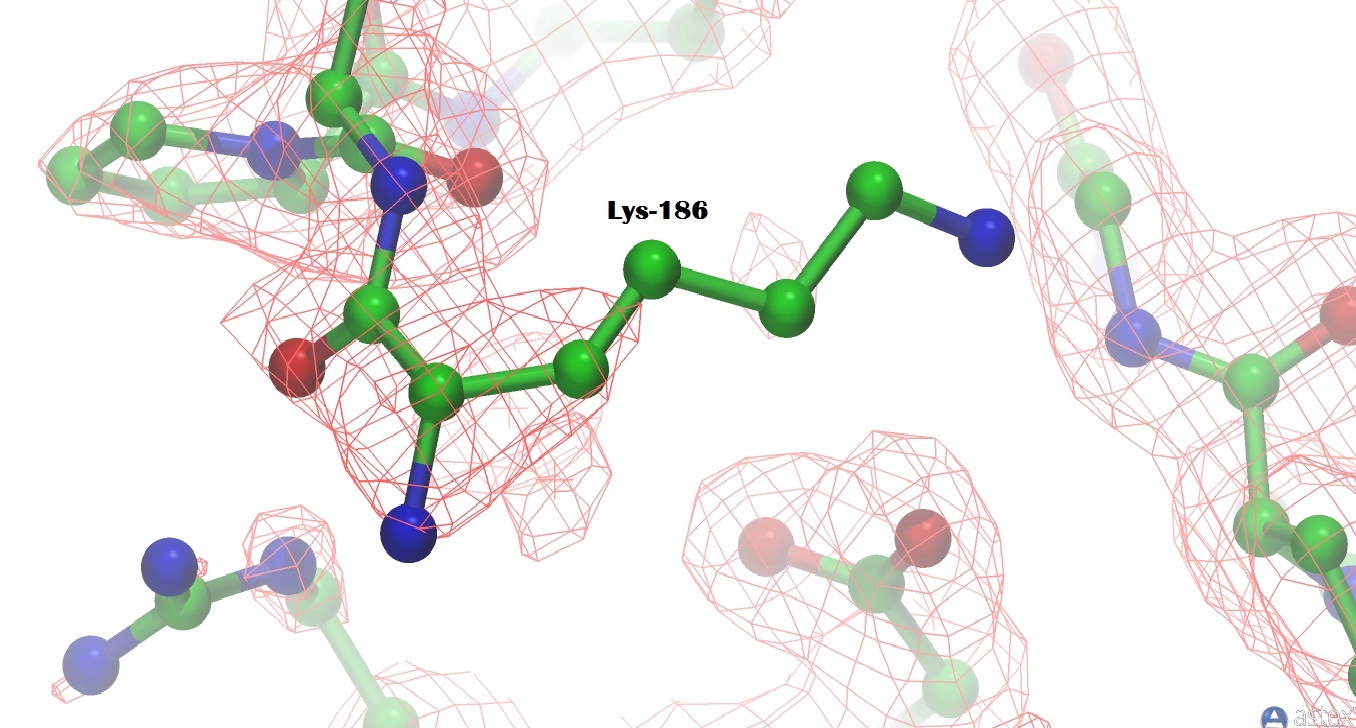
На сервере EDS были найдены 10 (4%) остатков имеют большой Z-score по RSR. Для дальнейшего анализа возьмем один из них, например, Lys -186.
пространственного R-фактора (RSR) = 0,261 Z-score пространственного R-фактора = 2,455 коэффициента корреляции для электронной плотности ("real-space correlation coefficient") = 0,825 Пространственный R-фактор >0,1 , Z-score >0. Значит данный остаток плохо расположен, и как следствие он плохо вписался в электронную плотность, что видно из картинки.
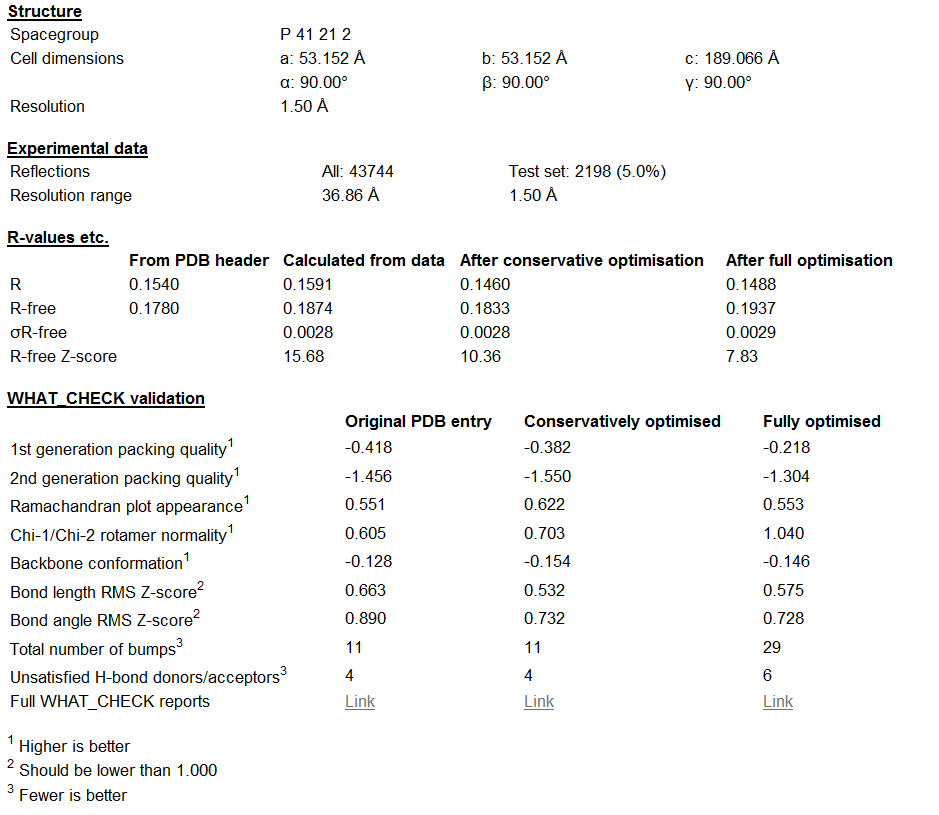
Улучшились все показатели за исключением total numer of bumps, unsatisfied H-bond donor/acceptor, backbone conformation, R-free, в целом качество структуры после оптимизации улучшилось.
|
| ||||||||
|
|
|||||||||
|
© Замараев Алексей |
|
||||||||