|
|
|
|
|
|
|
|
|
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
 |
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
|
|
|
|
|
|
|
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Ферменты и метаболические пути
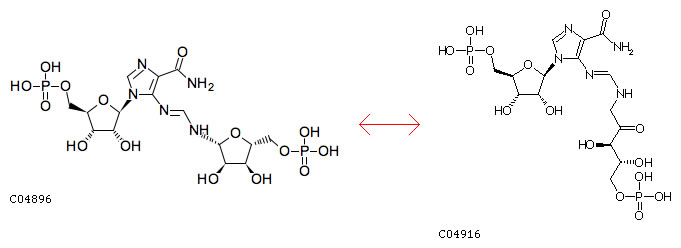
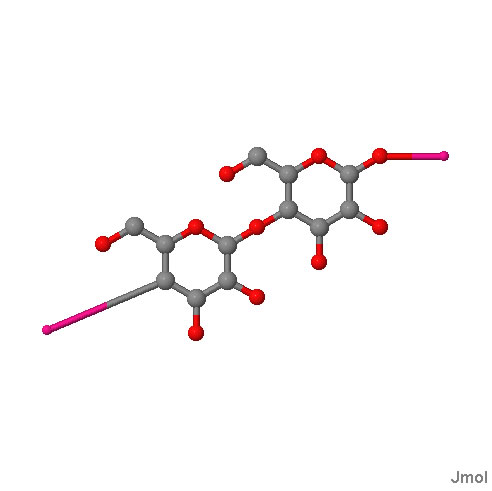
Заданный белок HIS4_ECOLI EC=5.3.1.16 5-Isomerases-изомеразы (класс ферментов, катализирующих р-ции изомеризации) 3-Intramolecular Oxidoreductases-внутримолекулярные оксидоредуктазы (класс ферментов, катализирующих обратимые окислит.-восстановит. р-ции) 1-Interconverting Aldoses and Ketoses-превращает альдозы в кетозы, и наоборот 16- 1-(5-phosphoribosyl)-5-[(5-phosphoribosylamino)methylideneamino] imidazole-4-carboxamide isomerase- 1-(5-фосфорибозил)-5-[(5-фосфорибозаиламино)метилденеамино] имидазол-4-карбоксамид изомераза Реакция 1-(5-phosphoribosyl)-5-[(5-phosphoribosylamino)methylideneamino]imidazole-4-carboxamide = 5-[(5-phospho-1-deoxyribulos-1-ylamino)methylideneamino]-1-(5-phosphoribosyl)imidazole-4-carboxamide
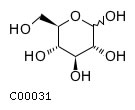
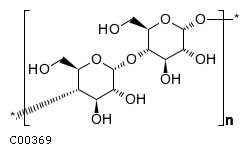
Идентификатор eco:b2024 Имя гена hisA Метаболические пути eco00340 Histidine metabolism (метаболизм гистидина) KEGG структурные формулы заданных соединений D-глюкоза(D-Glucose) Идентификатор KEGG C00031 Крахмал(Starch) Идентификатор KEGG C00369 Метаболический путь от одного заданного вещества к другому Выбранная цепочка ферментативных реакций: на карте оранжевым отмечена цепочка, красным - крахмал (C00369), зеленым - D-глюкоза(C00031), желтым - промежуточный продукт мальтоза (С00208) Сравнение метаболических путей у разных организмов
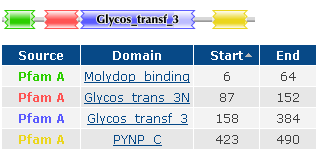
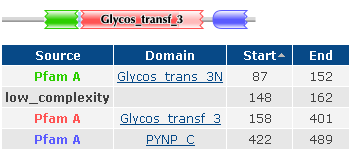
Карта метаболизма сахарозы и крахмала свойственна данным организмам, и имеет схожие пути метаболизма. Сравнение ферментов из далеких организмов На страничке SRS составляем запрос([uniprot-ECNumber:2.5.1.46] & ([uniprot-ID:*_human] | [uniprot-ID:*_ARCFU])). У Archaeoglobus fulgidus было также найдено 2 белка TYPH1_ARCFU и TYPH2_ARCFU. Были установлены доменные структуры белков.
слева- TYPH1_ARCFU справа- TYPH2_ARCFU У человека было найдено 2 белка TYPH_HUMAN и B3KQ24_HUMAN
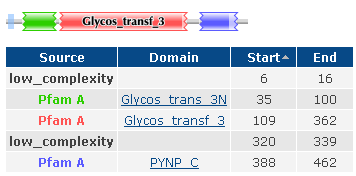
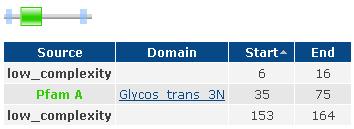
слева- TYPH_HUMA справа- B3KQ24_HUMAN Белки TYPH_HUMAN и B3KQ24_HUMAN имеют один похожий домен Glycos_trans_3N
Было произведено сравнение домена Glycos_trans_3N у Typh2_arcfu и typh_human с помощью локального выравнивания water. Процент идентичности: 77/244 (31.6%) Схожесть: 125/244 (51.2%) Счет: 300.0 В Kegg были найдены белки Typh2_arcfu (AF1342) и Typh_human(1890) и выбраны для человеческого белка лучшего ортолога из архей, а для архейного –- лучшего ортолога у эукариот.
Из данной таблицы видно, что ортолог чеовеческого белка, обладает большей идентичностью, счетом и длинной выравнивания. А у белка археи был найден ортолог только предсказанного белка.
|
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
© Замараев Алексей |
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||