|
|
|
|
|
|
|
|
|
|
|
 |
|
||||||||
|
|
|
|
|
|
|
|
|
||
Поиск белка с заданной функциональной специфичностью
В качестве белка-прототипа задан белок B5Y088_KLEP3. Репрессор сахарозного оперона организма Klebsiella pneumoniae (strain 342) 334 аминокислотных остатка
Для поиска данного белка в протеоме Marinomonas, был создан профиль ДНК-связывающего домена, для этого получили все последовательности ДНК-связывающих доменов семейства LACI. На главной страничке БД SMART проведел поиск по имени домена, HTH_LACI. Затем получил представительское выравнивание доменов в формате FASTA, сохраните его в файле SMART.fasta. Далее последовательности всех бактериальных доменов данного семейства сохраняем в отдельный файл и спомощью программы ClustalW2 выравниванием последовательности scrr ДНК-связывающих доменов под профиль представительского выравнивания SMART.fasta. Получаем выравнивание доменов заданной группы специфичности scrr.fasta, в данной работе это SCRR (катаболизма сахарозы). Просмотр лого(scrr) Затем были получены выравнивания последовательности ДНК-связывающих доменов других групп специфичности (all.rar), после чего проводим множественное выравнивание muscle(muscle.fasta) и импортирем в Genedoc, группы специфичности были раскрашены по цветам, а консервативны позиции отмечены черным (Alig.jpg) Просмотр лого(все группы специфичности) Следующим шагом стало создание профиля заданной группы специфичности для этого добавляем веса в выравнивание с помощью pwf из пакета PFTOOLs: Профиль строится следующим образом:: И производим поиск по профилю в протеоме Marinomonas В результате на выходе мы видим 13 предполагаемых белков, которые соответствуют профилю днк-связыващих доменов нашей группы специфичности. Затем проводим множественное выравнивание предсказанных белков (predict.txt) с представительскими доменами (smart.fasta) и импортируем в GeneDoc (alig2.jpg), можно заметить точно консервативные позиции, это аланин и валин, которые располагаются на расстоянии 4 аминокислотных остатков. В файле alig2.jpg они отмечены красным цветом. Затем PDBsum было найдено лого соответствующее днк-связывающему домену LacI. Просмотр лого (LacI) Как видно из лого, на 6 и 11 позиции находятся аланин и валин, также как и в наших пресказанных белках. Если посмотреть доменную струкуру нашего белка:
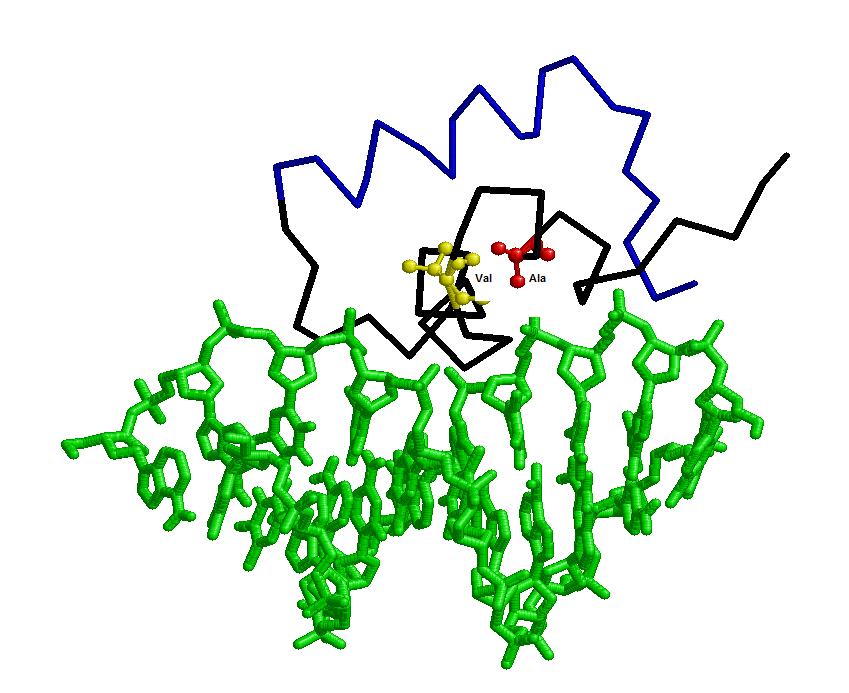
Днк-связывающему домену LacI приведена структура: На картинке структуры домена LacI видно, что 10 и 15 позиции, соответсвуют аланину и валину, а красные черточки внизу данных аминокислотных остатков говорят о высокой консервативности. Это все говорит о том, что предсказынне нами белки в геноме Marinomonas имеют Днк-связывающий домен LacI и относятся к заданной группе специфичности. С помощью Rasmol было создано изображение, на котором зеленая спираль это ДНК, черным выделен Днк-связывающий домен LacI, а красным и желтым консервативные позиции, аланин и валин соответственно.
|
| ||||||||
|
|
|||||||||
|
© Замараев Алексей |
|
||||||||
 .
.