|
|
|
|
|
|
|
|
|
|
|
 |
|
||||||||
|
|
|
|
|
|
|
|
|
||
Молекулярная динамика биологических молекул в GROMACSНам даны файлы:
С помощью программы fiber из пакета 3DNA построим небольшой дуплекс с последовательностью GATCTA. Для этого зададим путь и переменную среды: export X3DNA=/home/preps/golovin/progs/X3DNA
export PATH=/home/preps/golovin/progs/X3DNA/bin:${PATH}
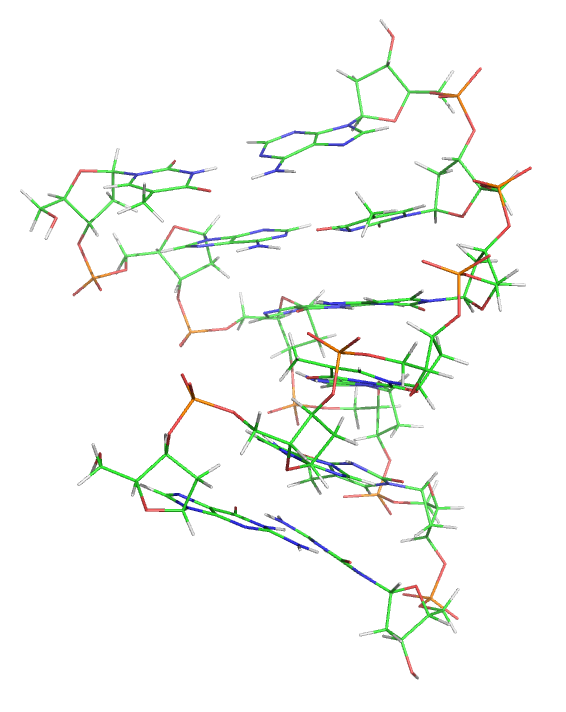
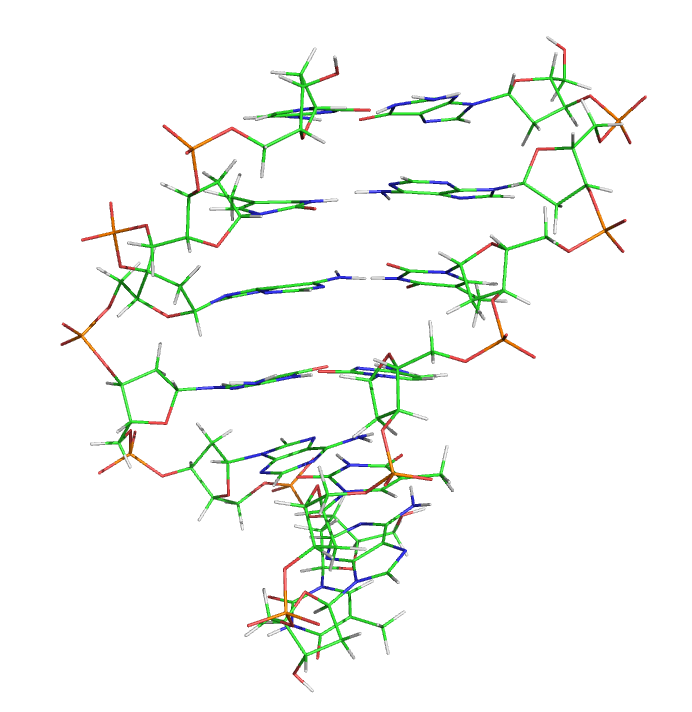
Теперь построим файл топологии системы в силовом поле amber99sb и файл с координатами в формате Gromacs. Предполагается, что структура дуплекса находится в файле dna.pdb. pdb2gmx -f dna.pdb -o dna -p dna -ff amber99sb -water tip3p При этом в pdb удалим 5' фосфаты из структуры дуплекса. Их два на 5' конце кажой цепи. Также надо изменить имена нуклеотидов добвавив D к названию нуклеотида," Т"->"DT" (пример для vim: :%s/ \([GATC]\) \([AB]\)/ D\1 \2/). И замените имя атома "С5М" на " С7". Сделаем небольшой отступ в ячейке от ДНК. editconf -f dna.gro -o dna_ec -d 1.5 Проведём оптимизацию геометрии системы, что бы удалить "плохие" контакты в молекуле. grompp -f em -c dna_ec -p dna -o dna_em -maxwarn 1 mdrun -deffnm dna_em -v Добавим в ячейку молекулы воды. genbox -cp dna_em -p dna -cs -o dna_s Нейтрализуем заряд системы. Это делаем в два шага: строим tpr и запускаем genion. grompp -f em -p dna -c dna_s -o dna_s genion -s dna_s -o dna_si -p dna -np X где Х=10 это количество положительных ионов необходимых для нейтрализации заряда системы, т к заряд системы -10. Проведём "утряску" воды: grompp -f pr -c dna_si -p dna -o dna_pr -maxwarn 1 mdrun -deffnm dna_pr -v Теперь скопируем наши файлы на суперкомпьтер. Запустим тестовое моделирование на суперкомпьтере. grompp -f md -c dna_pr -p dna -o dna_md -maxwarn 1 mpirun -np 16 -q test -maxtime 5 /home/golovin/progs/bin/mdrun_mpi -deffnm dna_md -v Запускаим основное моделирование на суперкомпьтере. mpirun -np 16 -maxtime 1200 /home/golovin/progs/bin/mdrun_mpi -deffnm dna_md -v Номер Вашей задачи -240955 Анализ результатовЛюбой анализ начинают с визуального анализа движений молекул. Поэтому получим pdb файл с анимацией движения молекулы. В данной анимации видно, что происходит переход из ДНК из А формы в В. Сначала ДНК находилась в А форме, это похоже на картинку ниже, если посмотреть сбоку на ДНК
t= 0.00000 пс Затем она стала похоже на В форму.
t= 9600.00000 пс
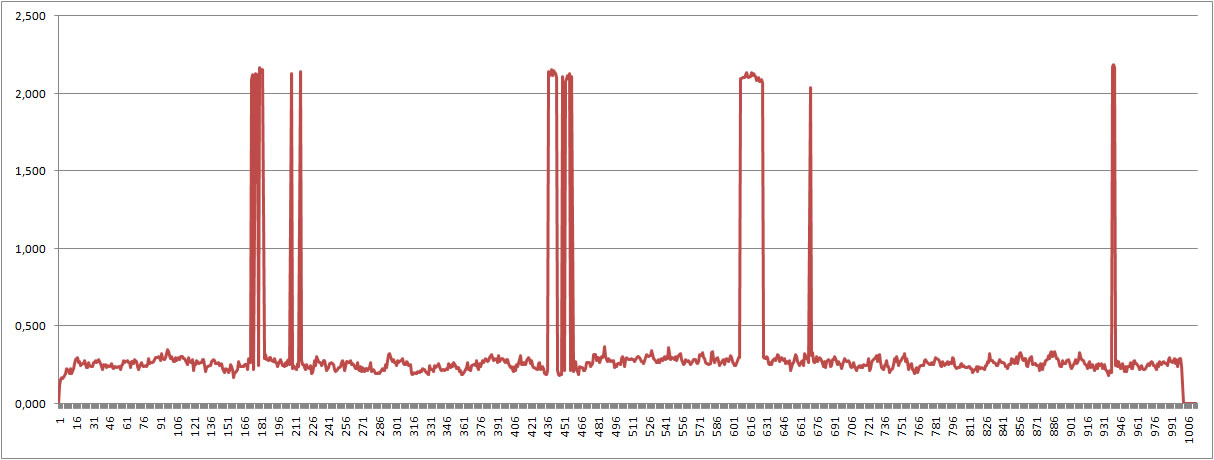
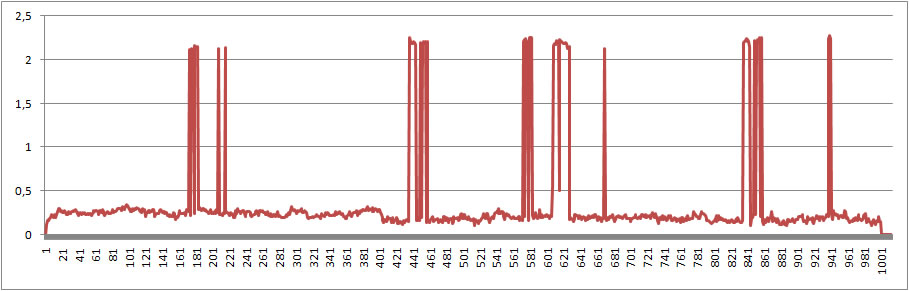
Как видно на графике сердне-квадратичное отклонение в ходе симуляции остаяется постоянным за исключением нескольких точек, в принципе это можно назвать артефактами моделирования.
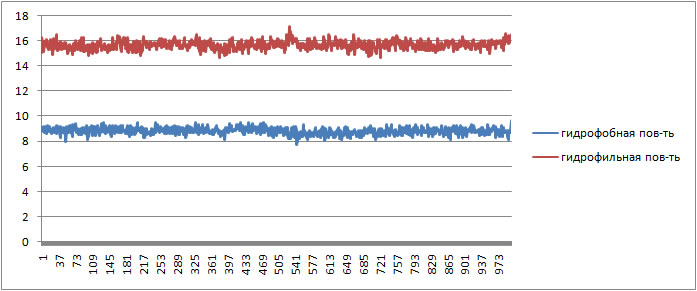
Как видно из графика выше гидрофобная и гидрофильная пов-ть доступных растворителю, в нашем случае это вода, не изменяются
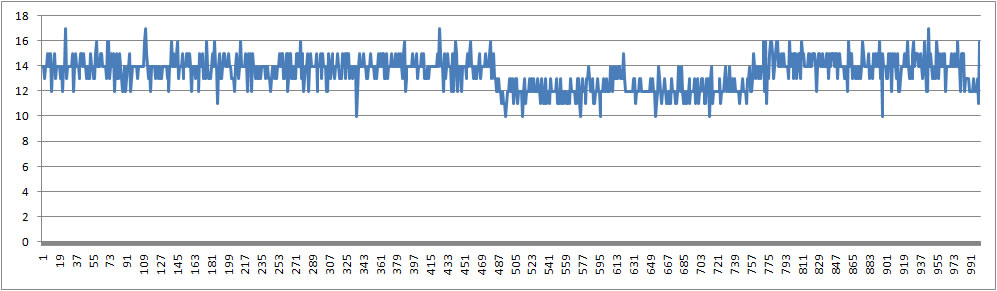
На графике выще представлено количество водородных связей в дуплексе ДНК, для дуплекса GATCTA должно в норме быть 14 водородных связей, на графикн видно, что в процессе перехода происходят какие-то конформационные изменения, которые приводят к увеличение расстоянию между одной парой нуклеотидова АТ, скорее всего это с 3' конца, где произошло подплавление, поэтому этот конец менее стабилен. Не менее интересно будет изучить количество вдородных связей ДНК-Вода
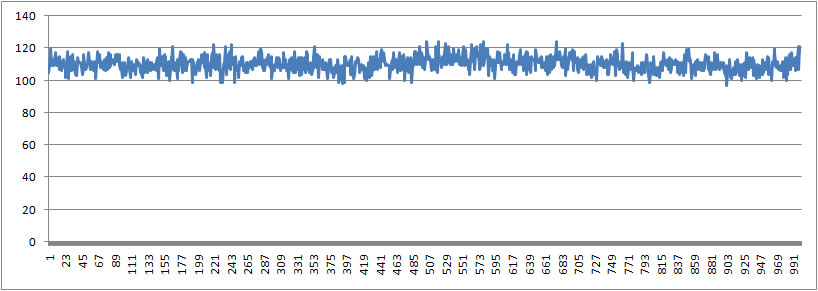
Как видно на графике количество водородных свзязей вода-ДНК не изменяется в процессе перехода ДНК из формы А в В, что скорее всего говорит, что в структуре ДНК не произходит сильных конформационных измений, которые могут повлиять на количество водородных связей ДНК-вода. |
|
||||||||
|
|
|||||||||
|
© Замараев Алексей |
|
||||||||