|
|
|
|
|
|
|
|
|
|
|||||||||||||||||||||||||
 |
|
||||||||||||||||||||||||||||||||
|
|
|
|
|
|
|
|
|
||||||||||||||||||||||||||
Мембранные белки, транспортные белки Открыл домашнюю страничку БД PDBsum, предоставляющей краткое схематическое отображение информации о структуре. Нашел документ с идентификатором PDB 3H90. Получиk окно с выравниванием последовательности из UniProt и последовательности из PDB. Нумерация двух баз данных отличается, начало и конец базы данных Uniprot длинее на 7 а.о и 10 а.о соответственно.
Было проведено глобальное выравнивание FIEF_PECCP и FIEF_ECOLI Последовательности заданных белков: FIEF_PECCP и FIEF_ECOLI Для этого применял программу needle пакета EMBOSS needle rec.fasta ec.fasta virovn.msf -aformat msf Файл, выданный программой needle скачать Полученное выравнивание cкачать
Открыл домашнюю страничку БД ОРМ, по PDB ID нашел описание ТМ-сегментов в белке-прототипе 3H90.
Сохранил все в файле mark.msf
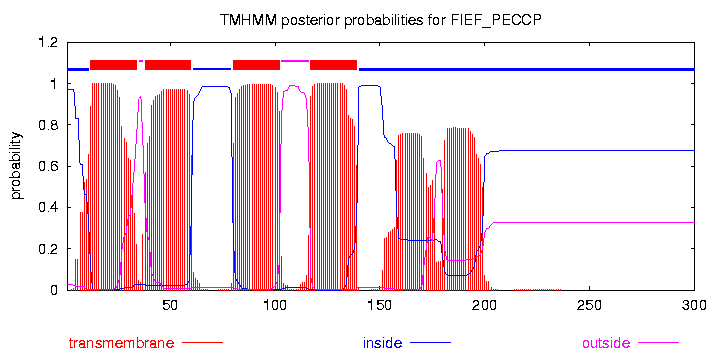
Предсказания топологии FIEF_PECCP с помощью сервера TMHMM.
Добавил к последовательностям в файле mark.msf еще одну искусственную последовательность с разметкой трансмембранных спиралей в соответствии с результатами предсказания.
Резюмируя, можно сказать, что качество предсказание методом TMHMM высокое, т к велика специфичность и чувствительность, а процент недопрдсказаний 15%. Однако этод метод не предсказал два трансмембранных сегмента 5(149-158), 6(179-198). НО, если посмотреть каринку полученную методом TMHMM, то можно заметить два пика, которые скорее всего соответствуют 5 и 6 сегменту, а так как их вероятность приблизительно равно 80%, программа TMHMM не внесла их в список трансмембранных сегментов.
|
| ||||||||||||||||||||||||||||||||
|
|
|||||||||||||||||||||||||||||||||
|
© Замараев Алексей |
|
||||||||||||||||||||||||||||||||