Результаты поиска при длине слова 2 и максимальном числе находок 500:
| Все находки | Находки с E-value < 0.001 | max E-value |
| 319 | 295 | 6.7 |
3. Изменение объёма поиска.
Выбрала находку с минимальным ненулевым E-value 7e-169 и весом 475 бит. Потом повторила поиск, ограничив его таксоном Firmicutes, и E-value изменилась до 7e-170. То есть, если N - размер всей базы данных (суммарная длина всех последовательностей), а n - суммарная длина всех последовательностей из таксона Firmicutes, то N = 10n. N ~ 198311666, средняя длина последовательностей в UniProtKB / Swiss-Prot ~ 358 аминокислот (числа отсюда) , поэтому примерное число последовательностей из таксона Firmicutes равно 55394. На сайте Uniprot указано, что 68654 последовательности из Firmicutes относятся к Swiss-Prot. Расчеты очень примерные, но порядок совпал. При вычислении веса (обычного или в битах) размер базы данных никак не фигурирует, так что из-за изменения объема поиска вес измениться не может.
4. Другие веб-интерфейсы BLASTP.Uniprot
Можно открыть результат поиска в новом окне, быстро работает, на вход принимает только последовательность либо идентификаторы Uniprot. Доступна база данных UniprotKB, прямо в базе данных можно ограничить поиск таксоном или кластером похожих белков.
BLAST
Медленнее, принимает на вход последовательность или разные идентификаторы, можно загрузить файл. Показывает все выравнивания прямо на странице с результатами. В параметрах можно указать длину слова для поиска, установить лимит на число совпадений с query последовательностью. Можно установить штрафы за начало и продолжение гэпов. Доступны максимально разнообразные базы данных, в том числе Non-redundant protein sequences (nr).
EMBL - EBI
Есть графа Functional predictions, где показана доменная структура каждой находки (в BLAST тоже такое есть, но не для каждой находки) (рисунок 1).
Рисунок 1. Доменная структура находки.
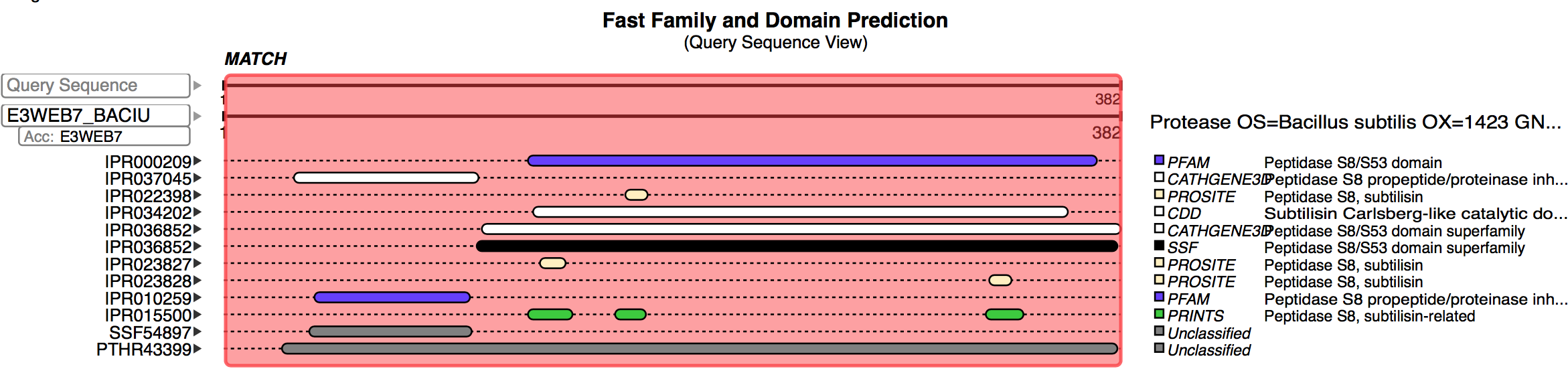
На вход можно загрузить файл либо ввести последовательность, идентификаторы не принимает. В параметрах можно установить штрафы за открытие и продолжение гэпа, порог не только по E-value и числу находок, но и по весу; указать начало и конец query последовательности, выбрать статистические данные для выравниваний на выходе. Доступные базы данных: UniProt Knowledgebase, UniProtKB/Swiss-Prot, UniProtKB/Swiss-Prot isoforms, UniProtKB/TrEMBL. Также, как в Uniprot прямо при выборе базы данных можно выбрать таксон, кластер похожих белков, некоторые специальные базы данных.
Во всех трех интерфейсах есть борьба с регионами низкой сложности, можно поменять матрицу аминокислотных замен, порог E-value, порог числа находок, есть графическое представление выравниваний (рисунки 2, 3, 4):
Рисунок 2. В Uniprot:
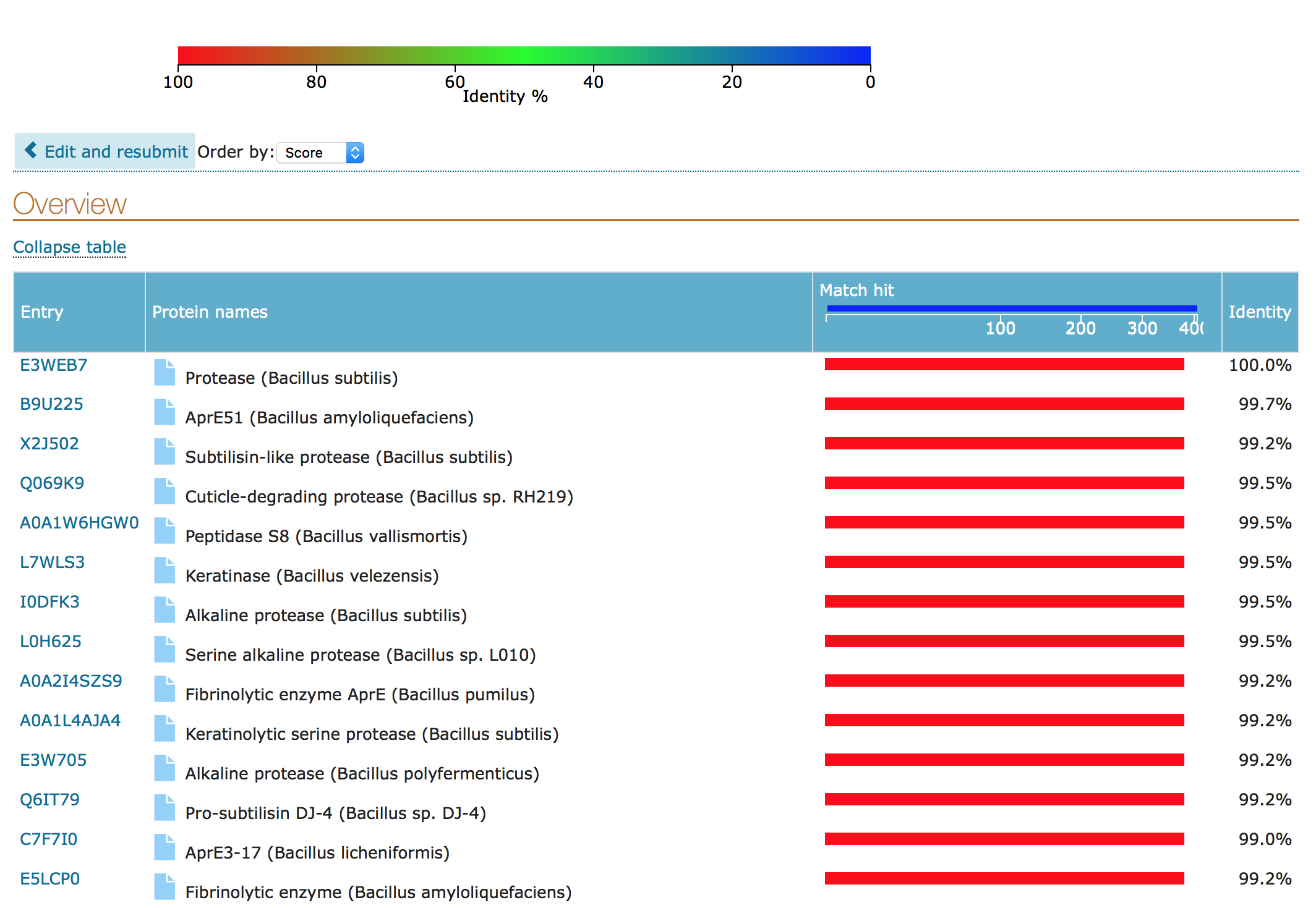
Рисунок 3. В BLAST:
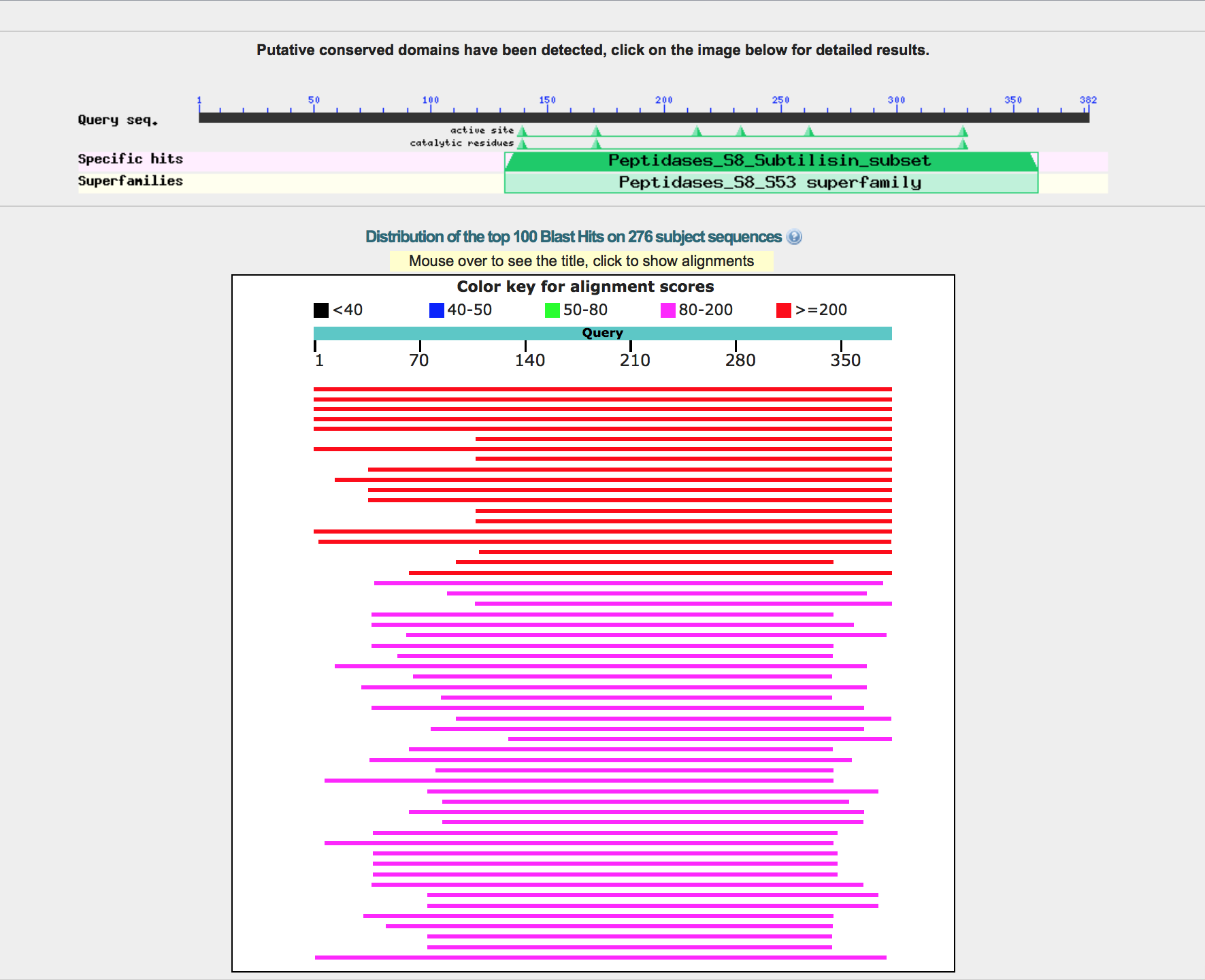
Рисунок 4. В EMBL - EBI:
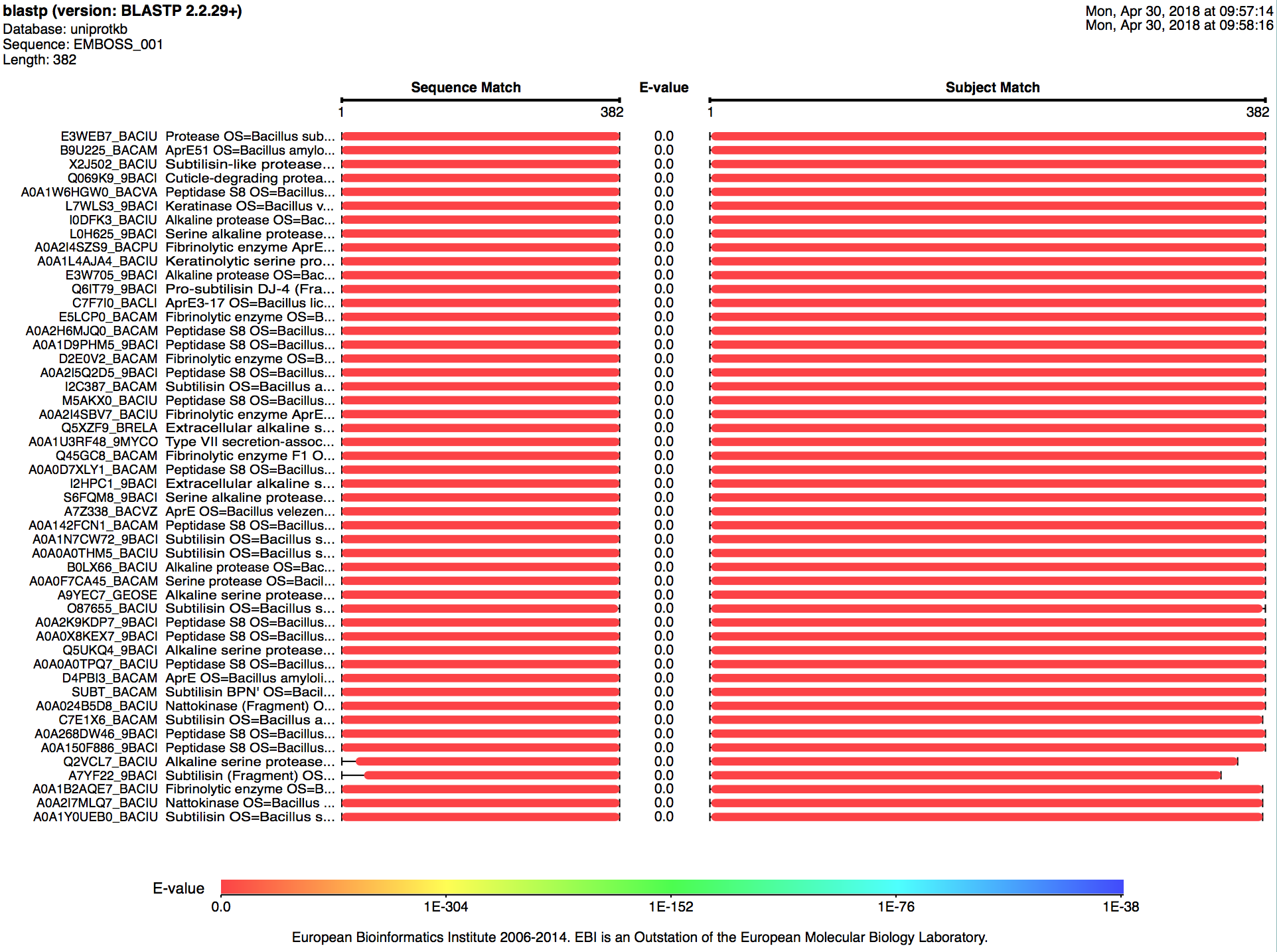
Таким образом, если нужно очень быстро пробластовать без особых параметров, то лучше использовать Uniprot BLAST; если, например, интересно посмотреть доменную организацию каждой находки или провести поиск в какой-то специальной базе даннных по белкам (например, в базе по полиморфизмам антител клеток-киллеров), то удобнее EMBL-EBI BLAST, а если мы не хотим упустить максимальное число находок, пусть даже плохо аннотированных и хотим видеть выравнивания со всеми характеристиками сразу на странице с результатом, то используем просто BLAST.
5. Плохая матрица.При использовании матрицы BLOSUM62 программа выдает 276 находок, а при PAM250 - 179 находок. На рисунках показаны находки с наилучшими E-value:
|
Рисунок 5. С BLOSUM62: 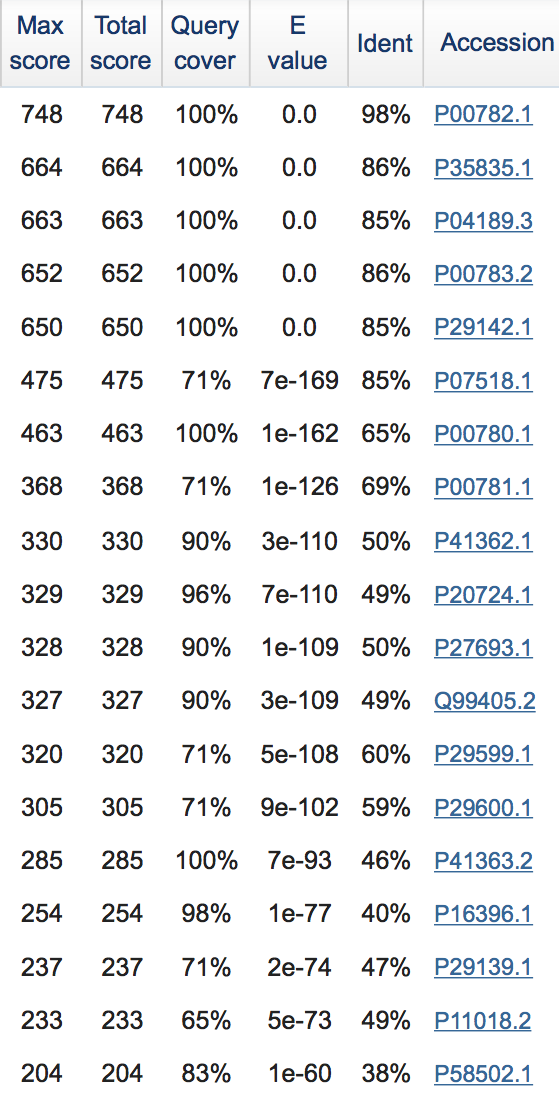 |
Рисунок 6. С PAM250: 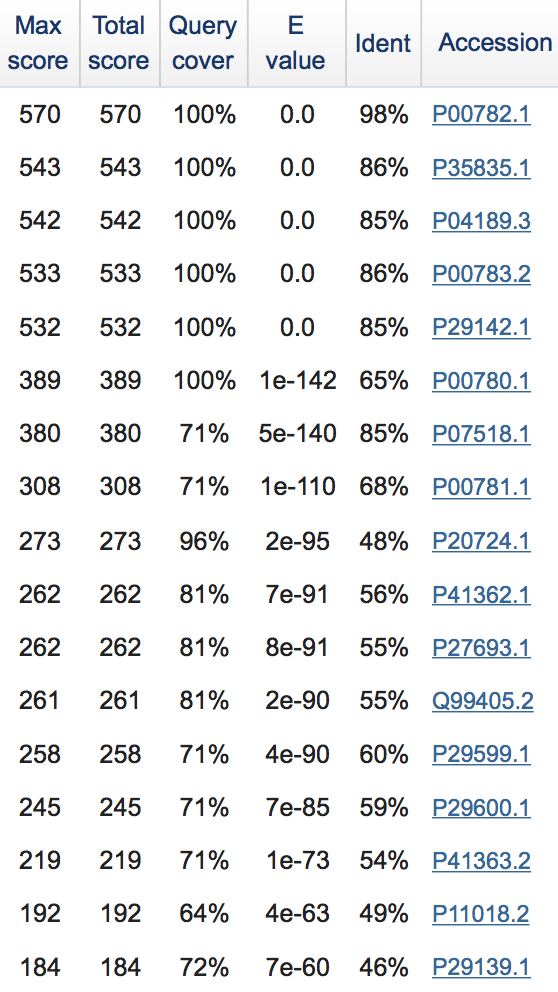 |
PAM250 хуже, спорить трудно.