1.Таблица с общей информацией.
| UniProt AC | UniProt ID | RefSeq ID | PDB ID | Длина | Молекулярная масса, Да | Рекомендуемое UniProt название |
| Q46G04 | ACDA1_METBF | WP_011305243.1 | 3CF4 | 806 AA | 88775 MW | Full=Acetyl-CoA decarbonylase/synthase complex subunit alpha 1 Short=ACDS complex subunit alpha 1 |
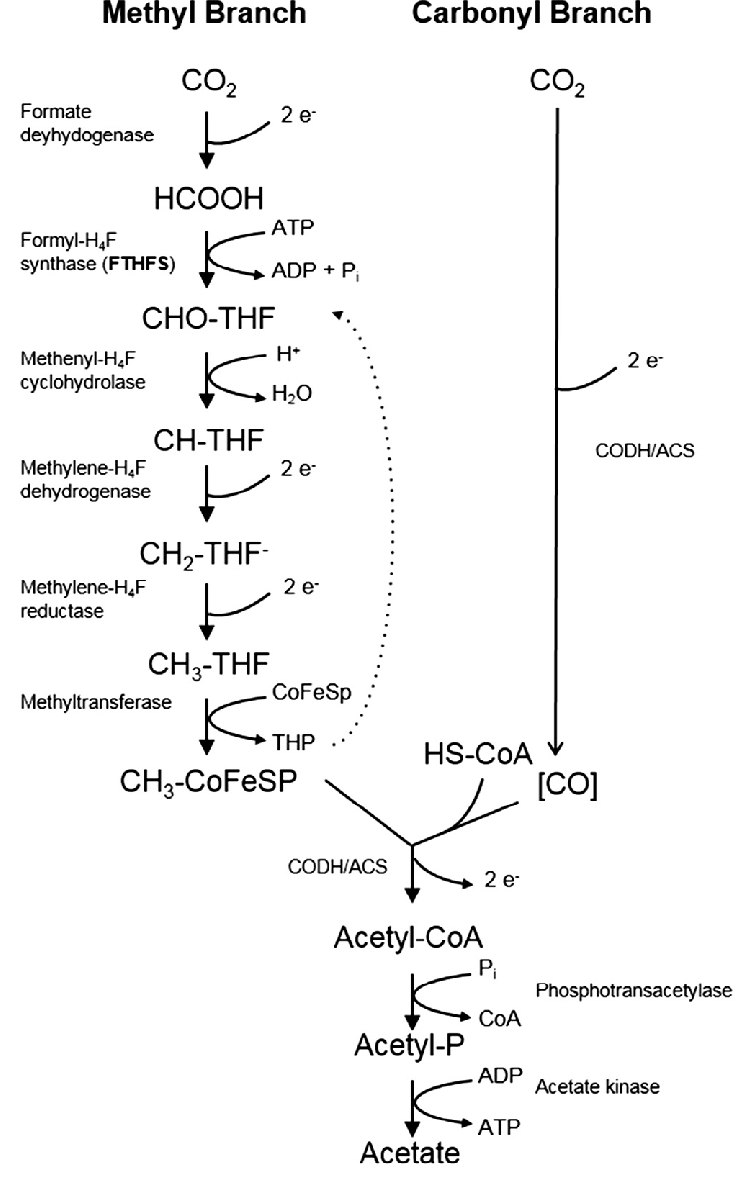
Семейство белков, непосредственно производящих бОльшую часть биогенного метана на Земле – это Ni-зависимые дегидрогеназы моноксида углерода (Ni-CODH), состоящие из альфа, бета, гамма, дельта, эпсилон субъединиц (по 8 каждой). Ni-CODH катализирует, например,
обратимое окисление CO до CO2 в ацетогенах, метаногенах и в других микроорганизмах, использующих CO. Эти реакции являются ключевыми в фиксации углерода (рисунок 1). PDB ID 3CF4 соответствует
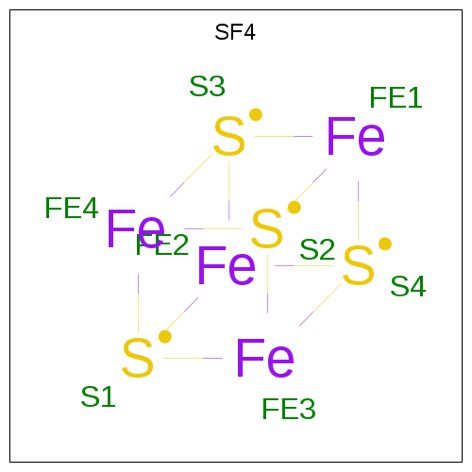
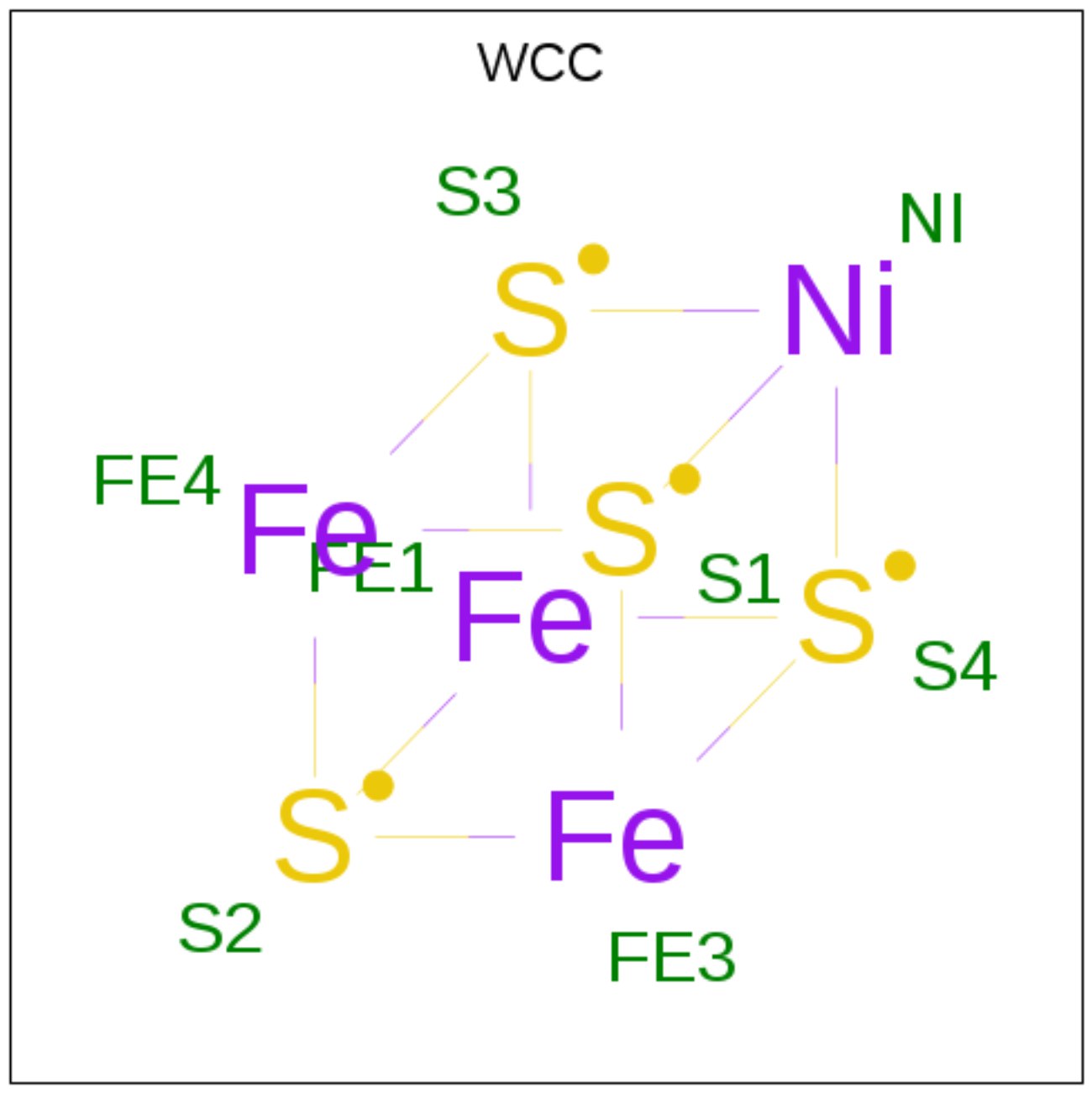
альфа(2)эпсилон(2) субъединице этого комплекса, в которой много лигандов: FeS-кластер (рисунок 2), NiFeS-кластер (рисунок 3) (данный кластер
представлен в ацетил-КоА декарбоксилазах исключительно метаногенов и, по-видимому, является ключевым в реакциях метаногенеза), Fe (III), моноксид углерода, уксусная кислота, дигидроксиэтил,
глицерин https://www.rcsb.org/structure/3cf4.
Однако под UniProt AC Q46G04 находится лишь альфа(1) субъединица (цепь А) ацетил-КоА декарбонилазы, состоящая из 806 аминокислот,
экспериментально выявленная в Methanosarcina barkeri
(штамм Fusaro/ DSM 804).
2. Кластеры
| ID кластера | Количество белков в кластере | Длина сравниваемой последовательности (аминокислоты) | Примечания |
| UniRef100_Q46G04 | 2 | 806 | Последовательность исследуемого белка абсолютно идентична последовательности только одного белка: того же самого фермента из другого штамма того же вида (Methanosarcina barkeri str. Wiesmoor) |
| UniRef90_Q46G04 | 28 | 806 | У гомологов из других штаммов M. barkeri и из других видов. |
| UniRef50_Q46G04 | 95 | 806 | У гомологов из других штаммов M. barkeri и из других видов. |
3. Результаты сеансов поиска
| Тип поиска | Текст запроса | Сколько всего белков | Сколько белков из Swiss-Prot | Примечания |
| 1. Поиск по полному рекомендуемому названию. | name:"acetyl coa decarbonylase synthase complex subunit alpha 1" | 1132 | 59 | Находятся многочисленные субъединицы Ni-зависимой дегидрогеназы из различных метаногенов (Methanobacterium, Methanococcus, Methanothermococcus, Methanotorris и др.) |
| 2. Поиск по краткому рекомендуемому названию. | name:"acds complex subunit alpha 1" | 889 | 59 | То же |
| 3. Поиск среди белков своего организма. | name:"acds complex subunit alpha 1" organism:"methanosarcina barkeri (strain fusaro dsm 804)" | 9 | 2 | Нашлись все (альфа, бета, гамма, дельта, эпсилон, по одной или по две) субъединицы Ni-зависимой дегидрогеназы. |
| 4. Поиск по тому же названию среди белков из организмов семейства Methanosarcinaceae. | name:"acds complex subunit alpha 1" taxonomy:methanosarcinaceae | 415 | 33 | |
| 5. Поиск по тому же названию среди белков из организмов отдела Euryarchaeota. | name:"acds complex subunit alpha 1" taxonomy:euryarchaeota | 817 | 59 | |
| 6. Поиск по названию «гомеобокс» | name:homeobox | 44012 | 1396 | Для большого числа найденных полипептидов длина либо равна 60 аминокислот. Это согласуется с литературными данными о том, что длина гомеобоксного участка гомеозисного гена часто близка к 180 п. о. https://ru.wikipedia.org/wiki/Гомеобокс#Гомеодомен https://www.uniprot.org/keywords/KW-0371 |
| 7. Поиск по названию «гомеобокс» среди членистоногих | name:homeobox taxonomy:arthropoda | 4496 | 57 | Очень много гомеобоксных белков для насекомых, больше всего, конечно, для дрозофилы. Тоже с характерной длиной 60. |
| 8. Поиск по названию «гомеобокс» среди инфузорий | name:homeobox taxonomy:ciliophora | 2 | 0 | Оба белка имеют Annotation score всего 1, по видимому, являются транскрипционными регуляторами, причем один из них имеет типичную для гомеобоксных доменов длину, близкую к 60 аминокислотам, а второй белок имеет несколько нетипичных доменов. |
| 9. Поиск по названию «трипсин» | name:trypsin | 18951 | 311 | |
| 10. Поиск «трипсин» без «ингибитор» | name:trypsin NOT name:inhibitor | 14973 | 95 | То есть для ингибиторов трипсина было 3978 записей. |
4a. Сравнение записей Uniprot и RefSeq
| UniProt | RefSeq |
| Приведена катализируемая реакция, сказано, к какому классу ферментов относится. | Четко описана роль в биохимическом пути, в состав какого ферментного комплекса входит. |
| Все возможные идентификаторы для других баз данных. | Мало идентификаторов для других баз данных. |
| Указаны все лиганды, места их контактов с белком. | Не указаны, можно только искать в публикациях. |
| Описаны основные физико-химические свойства (максимальная скорокть реакции, константа Михаэлиса , регуляция активности фермента, молекулярная масса) со ссылками на публикации. | Только ссылки на публикации про данный белок в принципе. |
| Есть информация о вторичной структуре. | Нет. |
| Нет. | Есть интерактивный fasta-формат. |
| Показывает кластеры 100, 90, 50 % идентичности. | Показывает несколько (4) идентичных белка. |
| Запись соотносит белок только с 1 видом и конкретным штаммом. | Предупреждают, что данный белок может быть аннотирован и в других геномах RefSeq этого же или других видов. |
4b. Изучение истории записи моего белка.
Белок был аннотирован TrEMBL 13 сентября 2015 года, когда создали первую запись, в которую внесли последовательность белка, которая с тех пор больше не менялась. Тогда же у белка появился UniProt AC Q46G04_METBF. До 6 июля 2016 года в запись успели внести 89 поправок. Видимо, удалось экспериментально доказать существование Ацетил-КоА декарбонилазы на уровне белка (с annotation score = 5), поэтому 6 июля 2016 года запись была аннотирована куратором Swiss-Prot и, таким образом, получила статус Reviewed и UniProt ID ACDA1_METBF, который расшифровывается как Acetyl-CoA Decarbonylase из Methanosarcina Barkeri Fusaro. С тех пор запись правили еще 11 раз, последний – 28 февраля 2018 года.
4c. Примеры нестандартных модификаций белков в записи.
Альтернативный сплайсинг в Q62141.
В разделе CC -!- ALTERNATIVE PRODUCTS указано число идентификаторы изоформ. Фраза "No experimental confirmation available" означает, что эта изоформа была только предсказана, но не получена. Если этой пометки нет, значит данная изоформа была аннотирована хотя бы в двух разных базах данных, либо была обнаружена у близких видов.| CC | -!- ALTERNATIVE PRODUCTS: |
| CC | Event=Alternative splicing; Named isoforms=4; |
| CC | Name=4; |
| CC | IsoId=Q62141-4; Sequence=Displayed; |
| CC | Note=No experimental confirmation available.; |
| CC | Name=1; |
| CC | IsoId=Q62141-1; Sequence=VSP_014187; |
| CC | Name=2; |
| CC | IsoId=Q62141-2; Sequence=VSP_008225, VSP_008226, VSP_014187; |
| CC | Name=3; |
| CC | IsoId=Q62141-3; Sequence=VSP_008227, VSP_008228, VSP_014187; |
| CC | Note=No experimental confirmation available.; |
В разделе FT VAR_SEQ описаны конкретные различия изофрм:
| FT | VAR_SEQ | 275 | 302 | KKMKLRGTKDLSIAAVGKYGTLQEFSFF -> VLVHVWVLP |
| FT | AHGRSGVEAQAAGEPEARA (in isoform 3). | |||
| FT | {ECO:0000303|PubMed:15489334}. | |||
| FT | /FTId=VSP_008227. | |||
| FT | VAR_SEQ | 275 | 293 | KKMKLRGTKDLSIAAVGKY -> VGLQLKCAVVWFGYCTAE |
| FT | E (in isoform 2). | |||
| FT | {ECO:0000303|PubMed:10620510, | |||
| FT | ECO:0000303|PubMed:15489334}. | |||
| FT | /FTId=VSP_008225. | |||
| FT | VAR_SEQ | 294 | 954 | Missing (in isoform 2). |
| FT | {ECO:0000303|PubMed:10620510, | |||
| FT | ECO:0000303|PubMed:15489334}. | |||
| FT | /FTId=VSP_008226. | |||
| FT | VAR_SEQ | 303 | 954 | Missing (in isoform 3). |
| FT | {ECO:0000303|PubMed:15489334}. | |||
| FT | /FTId=VSP_008228. | |||
| FT | VAR_SEQ | 955 | 1098 | Missing (in isoform 1, isoform 2 and |
| FT | isoform 3). {ECO:0000303|PubMed:10620510, | |||
| FT | ECO:0000303|PubMed:15489334, | |||
| FT | ECO:0000303|PubMed:7889570}. | |||
| FT | /FTId=VSP_014187. |
Фосфорилированные остатки в Q15334.
В разделе CC -!- PTM – доказанная модификация со ссылкой на публикацию:
| CC | -!- PTM: Phosphorylated at least at Ser-663 by PRKCI. |
| CC | {ECO:0000269|PubMed:12725730}. |
В разделе FT MOD_RES перечислены все предполагаемые модификации:
| FT | MOD_RES | 663 | 663 | Phosphoserine. |
| FT | {ECO:0000269|PubMed:12725730}. | |||
| FT | MOD_RES | 958 | 958 | Phosphothreonine. |
| FT | {ECO:0000250|UniProtKB:Q80Y17}. | |||
| FT | MOD_RES | 967 | 967 | Phosphoserine. |
| FT | {ECO:0000250|UniProtKB:Q80Y17}. | |||
| FT | MOD_RES | 985 | 985 | Phosphoserine. |
| FT | {ECO:0000250|UniProtKB:Q80Y17}. |
В последовательности аминокислот модифицированные аминокислоты никак не отличаются, указаны просто однобуквенным кодом.
© Belousova Evgenia, 2017