Обязательная часть
Команды:
| /P/y14/term3/block4/SNP/bedtools2/bin/bedtools bamtobed -i pr12/chr6_aligned_sorted.bam > chr6.bed | Конвертирует выровненные риды из .bam формата в .bed, записывает результат в файл chr6.bed. |
| /P/y14/term3/block4/SNP/bedtools2/bin/bedtools intersect -a /P/y14/term3/block4/SNP/rnaseq_reads/gencode.genes.bed -b chr6.bed -u > chr6.intersect | Пересекает координаты выравниваний в файле chr6.bed с координатами разметки генов в файле gencode.genes.bed (причем разметка по генам именно в опции -а, потому что мы хотим увидеть, сколько ридов легло на каждый ген, а не на сколько генов откартировался каждый рид) и записывает в файл chr6.intersect названия генов, на которые откартировался хотя бы 1 рид (за счет опции -u). То есть просто убирает из файла gencode.genes.bed все гены, на которые не откартировались риды. |
| /P/y14/term3/block4/SNP/bedtools2/bin/bedtools intersect -a chr6.intersect -b chr6.bed -c > chr6_count.intersect | Пересекает координаты генов, на которые откартировался хотя бы один рид, из файла chr6.intersect с координатами всех ридов из файла chr6.bed и опция -с считает, сколько ридов откартировалось на каждый фрагмент гена, результат записывает в chr6_count.intersect |
Все риды откартировались на 2 белок-кодирующих гена: EEF1A1 из предыдущего практикума, на него откартировалось большинство ридов, и на ген MTO1,
на который откартировался всего 1 рид. Наверное, это случайный рид, и отсеквенировать хотели транскрипты EEF1A1. Еще по одному риду
откартировалось на гены miRNA AL603910.1, miscRNA Metazoa_SRP, antisense RP11-505P4.6, из которых последние два гена в Ensemble не нашлись.
Информация о генах:
| ген | EEF1A1 | MTO1 | AL603910.1 |
| полное название | eukaryotic translation elongation factor 1 alpha 1 | mitochondrial tRNA translation optimization 1 | novel transcript |
| размер | 8046 bp | 47658 bp | 370 bp |
| координаты | 73,515,750-73,523,797 | 73,461,578-73,509,236 | 73,492,025-73,492,742 |
| цепь | reverse | forward | reverse |
| функция | Фактор элонгации трансляции | фактор оптимизации трансляции в митохондриях | miRNA |
| количество экзонов | в транскрипте ENST00000316292.13, на который откартировались все риды - 7 | в транскрипте ENST00000498286.5 - 12 | |
| количество транскриптов | 11 | 19 | 1 |
Дополнительные задания
| Задание | Команда | Что делает |
| 4. Объединить чтения в кластеры | /P/y14/term3/block4/SNP/bedtools2/bin/bedtools cluster -i chr6.bed > chr6_clusters.bed | Команда кластеризует все риды и записывает результата в chr6_clusters.bed. Даже без параметра -d все риды объединяются в один кластер, то есть находятся очень близко. |
| 5. Набрать из хромосомы 1000 случайных фрагментов по 200 нуклеотидов | /P/y14/term3/block4/SNP/bedtools2/bin/bedtools random -g chr6.genome -l 200 -n 1000 > chr6_random.bed | Команда генерирует координаты 1000 случайных фрагментов 6 хромосомы длиной 200 bp и записывает их в файл chr6_random.bed |
| 7. Получить координаты одного из покрытых чтениями генов, расширенные на 1000 нуклеотидов в обе стороны | /P/y14/term3/block4/SNP/bedtools2/bin/bedtools flank -b 1000 -i chr6.intersect -g chr6.genome > chr6_flank.bed | Команда расширяет на 1000 bp в обе стороны координаты всех транскриптов, записанных в файле chr6.intersect (там только те, на которые откартировался хотя бы один рид). На рисунках изначальный транскрипт и тот же фланкированный транскрипт обведены в рамку. |
| До | После |
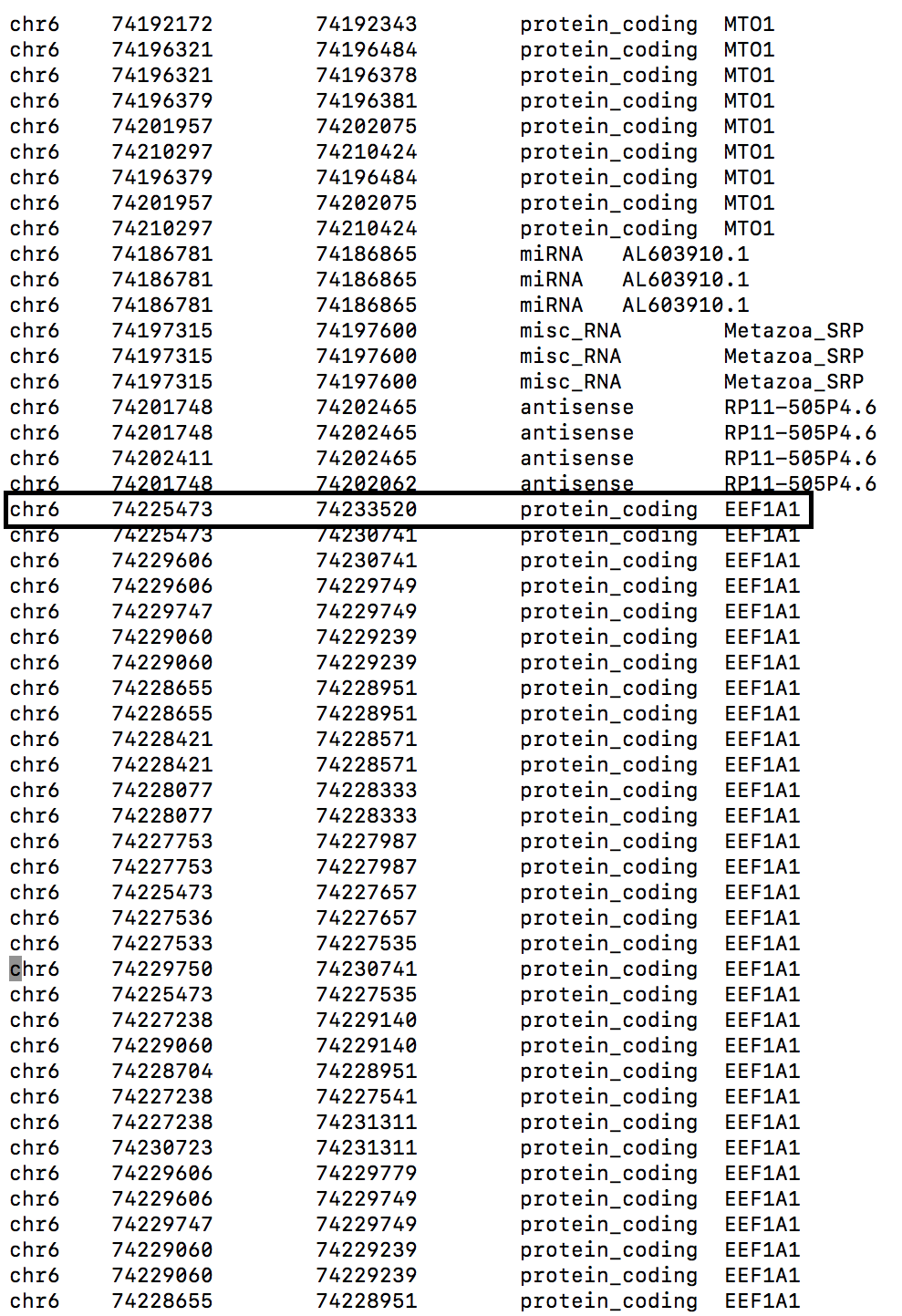 |
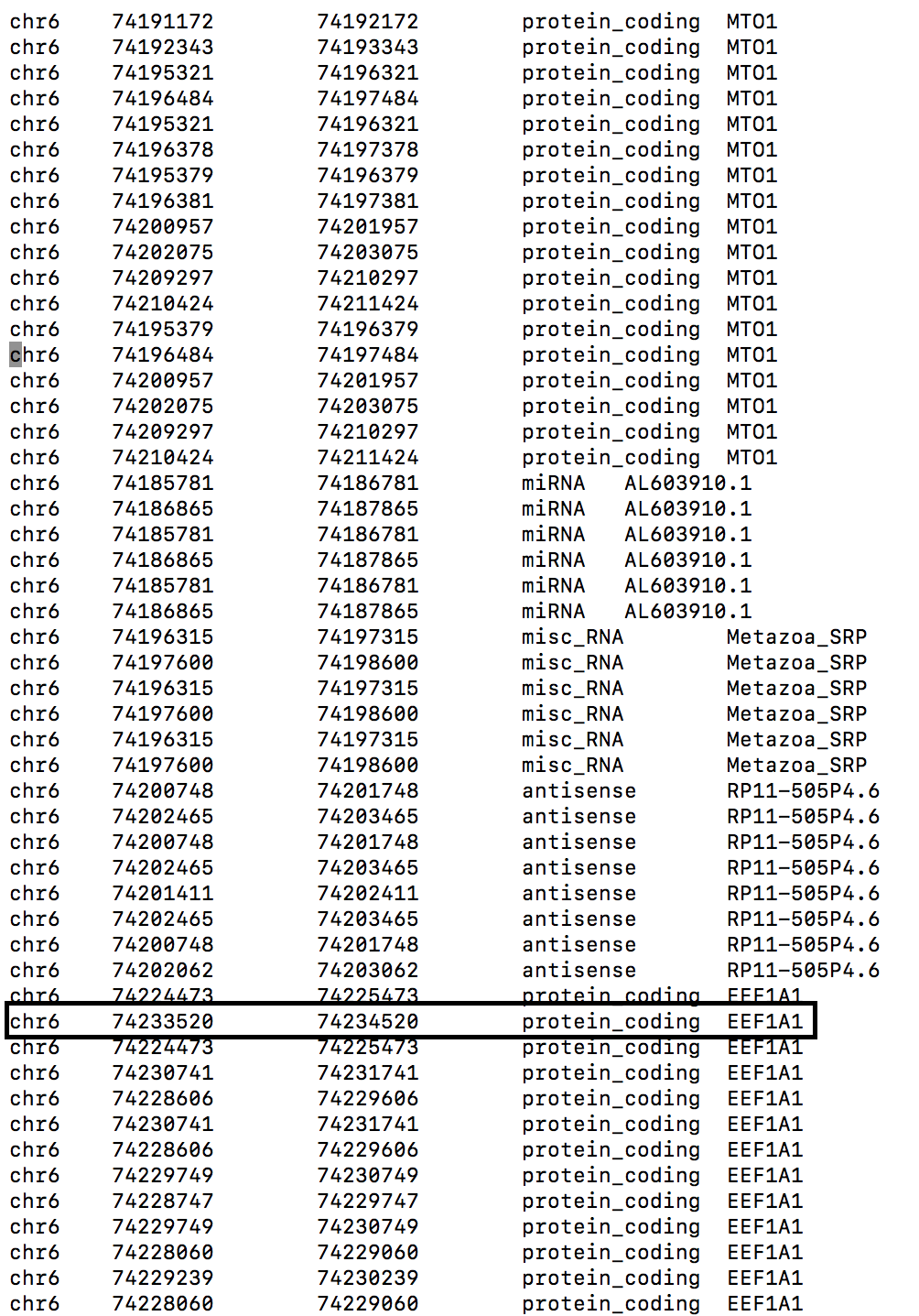 |