Задание 1. PoseView.
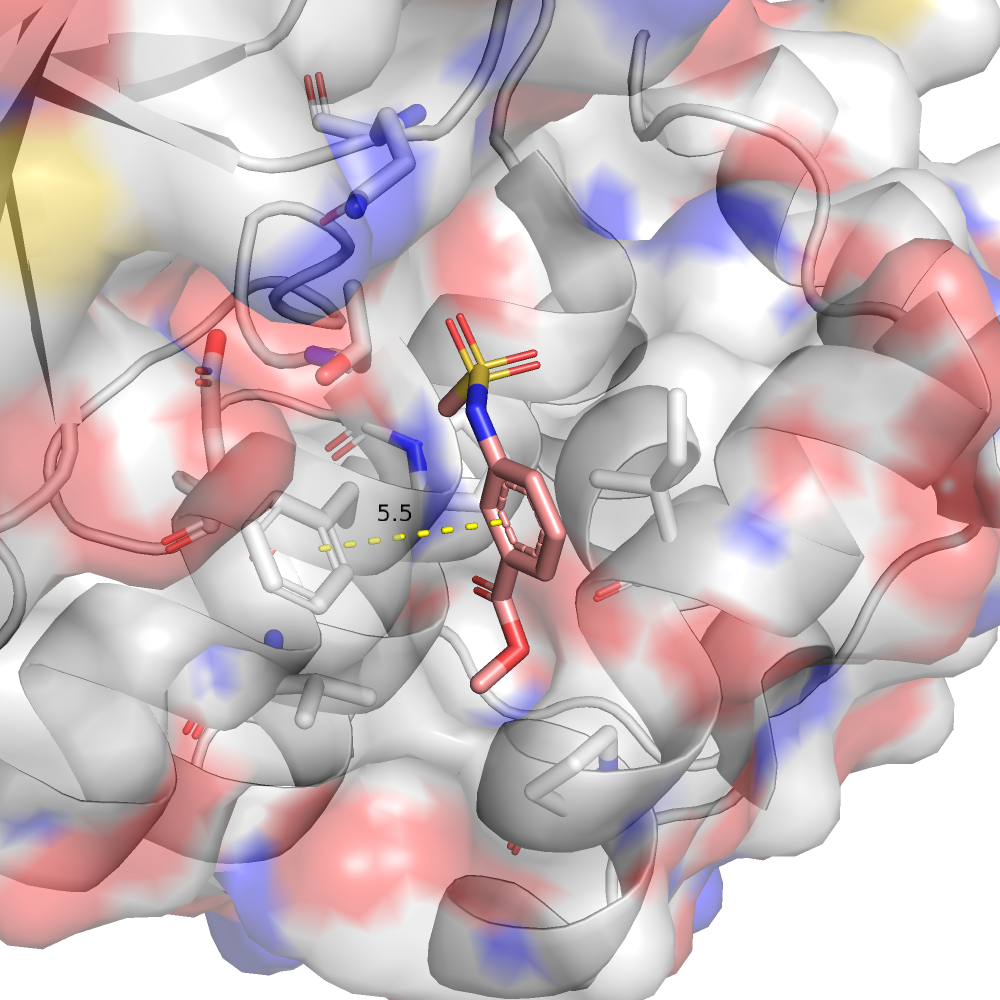
К сожалению, ни Plex View, ни Proteins.Plus не показали взаимодействий с лигандом на 2D диаграмме для моей структуры 5REF, что довольно обидно, потому что как минимум одно стекинговое взаимодействие c PHE 294 там есть. Сам этот остаток в окружении лиганда программа видит, а взаимодействие - нет. Наверное, расстояние в 5.5 ангстрем она расценивает, как слишком большое.
 |
 |
Задание 2. PyMol mutagenesis.
Сначал рассмотрим структуру AB1 (код 0025, цепь P, позиция 5). Все остатки описаны в таблице ниже, варьируемый остаток покрашен в розовый. Здесь при мутагенезе получилось много вроде бы подходящих аминокислот, с небольшой стерической напряженностью и хорошо представленным ротамером. Поэтому одного кандидата на место глицина выбрать оказалось затруднительно. Но, все же, рассуждаем. Мне кажется, что представленность ротамера около 30% - это совсем не плохо (особенно, если ротамеров вообще много), поэтому я рассмотрела MET и ASN и нашла у них по одному предполагаемому взаимодействию с окружением, и, учитывая, что у них малая напряженность, это очень неплохие кандидаты. Также неплохой кандидат THR, а вот LEU мне нравится меньше, потому что несмотря на небольшую напряженность и хорошую представленность, окружение кажется не очень подходящим для гидрофобной аминокислоты. Это же касается VAL. Для PHE и PRO нашлось по одному взаимодействию, у них очень хорошая представленность таких ротамеров, но сравнительно высокая напряженность. Ну и, наконец, SER и CYS выходят в лидеры, потому что для них нашлось по два взаимодействия, у таких ротамеров отличная представленность и малая стерическая напряженность. Выбрать из них лучшего кандидата сложно (они, в конце концов, похожи!), у CYS меньше напряженность, а у SER чуть лучше одна из водородных связей... Все же, я думаю, исходным остатком был CYS.
| Аминокислота | Номер ротамера | Стерическая напряженность (strain) | Представленность ротамера | Изображение |
| CYS | 1/3 | 13.29 | 81.7% | 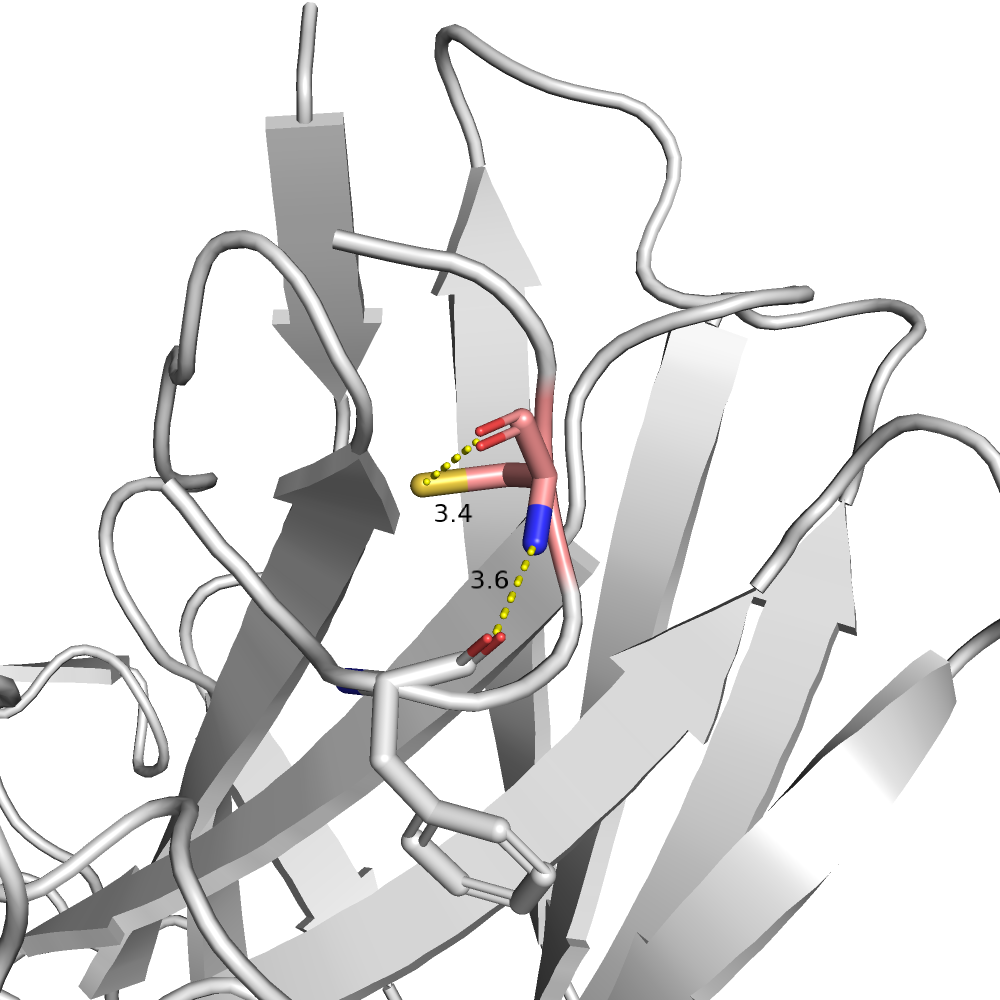 |
| SER | 1/3 | 17.04 | 87.0% | 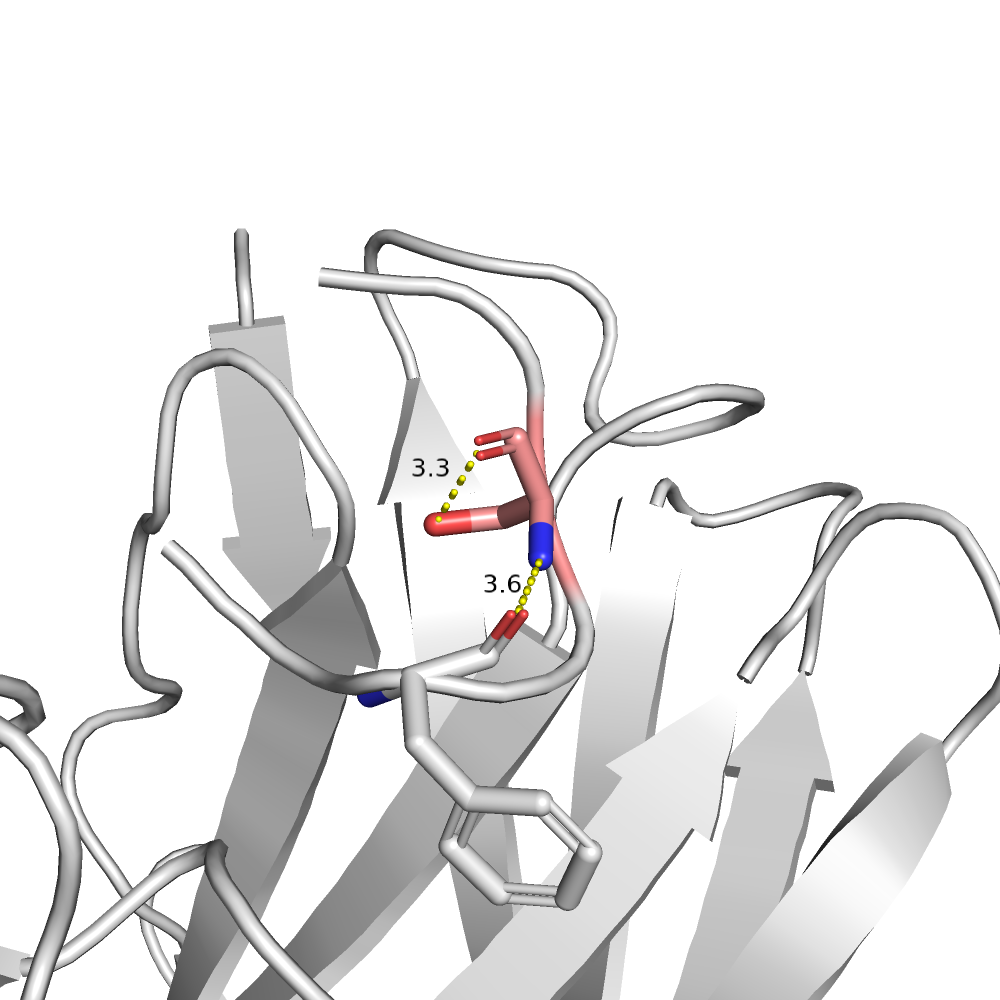 |
| VAL | 1/3 | 20.75 | 73.8% |  |
| PHE | 1/2 | 61.73 | 91.6% | 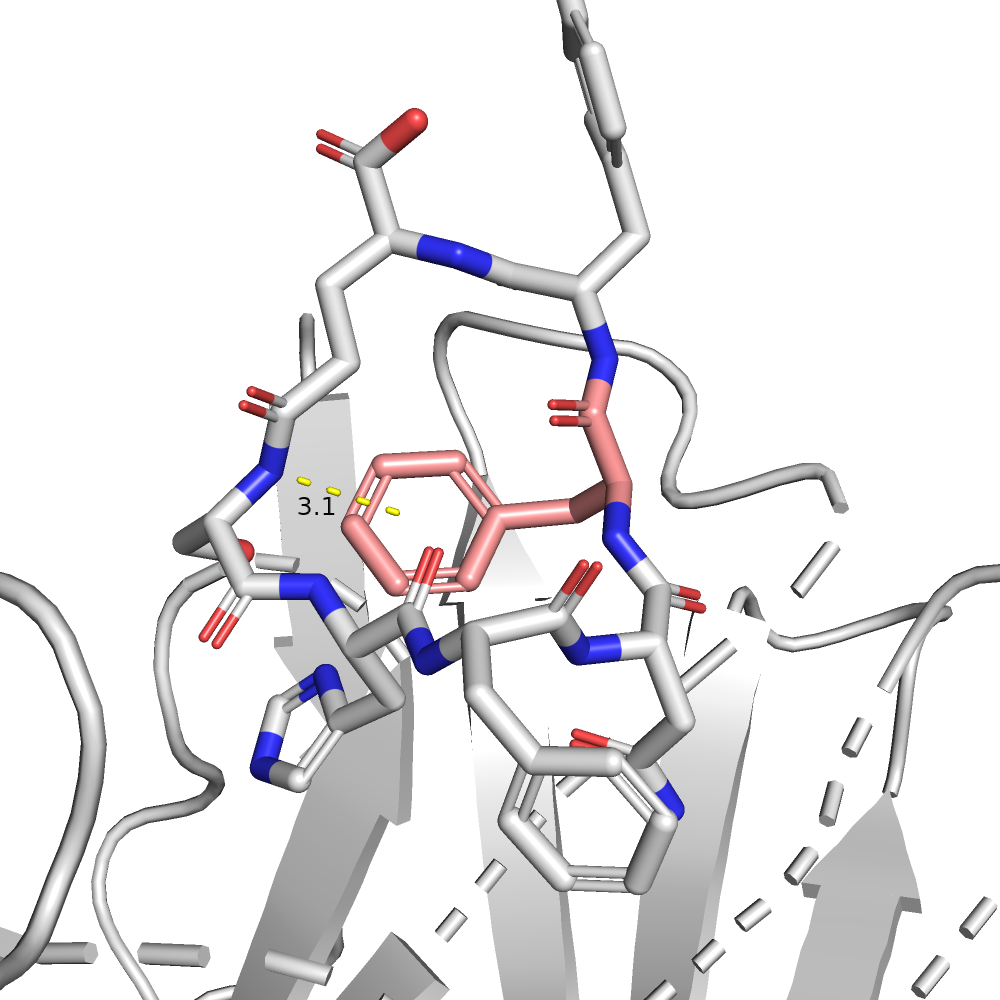 |
| PRO | 1/2 | 41.75 | 65.8% | 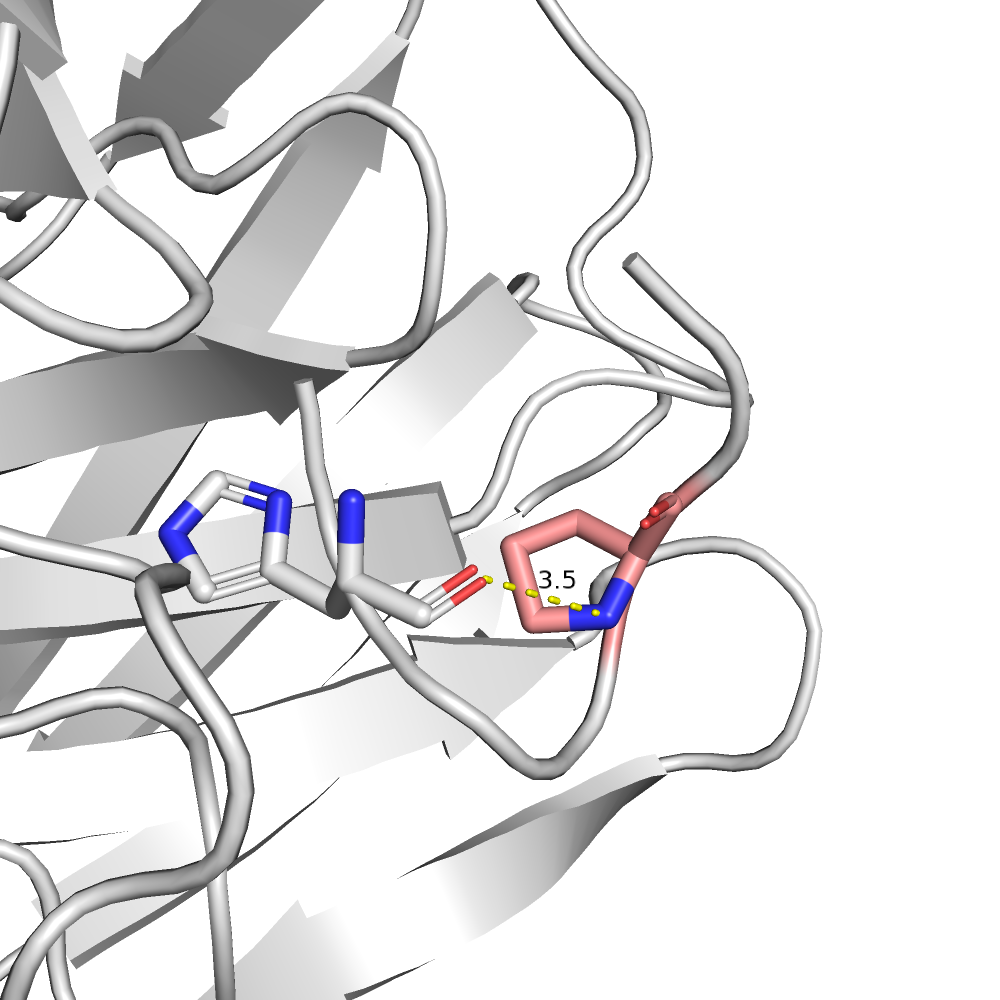 |
| LEU | 1/5 | 15.82 | 49.8% |  |
| THR | 2/2 | 14.74 | 46.9% | 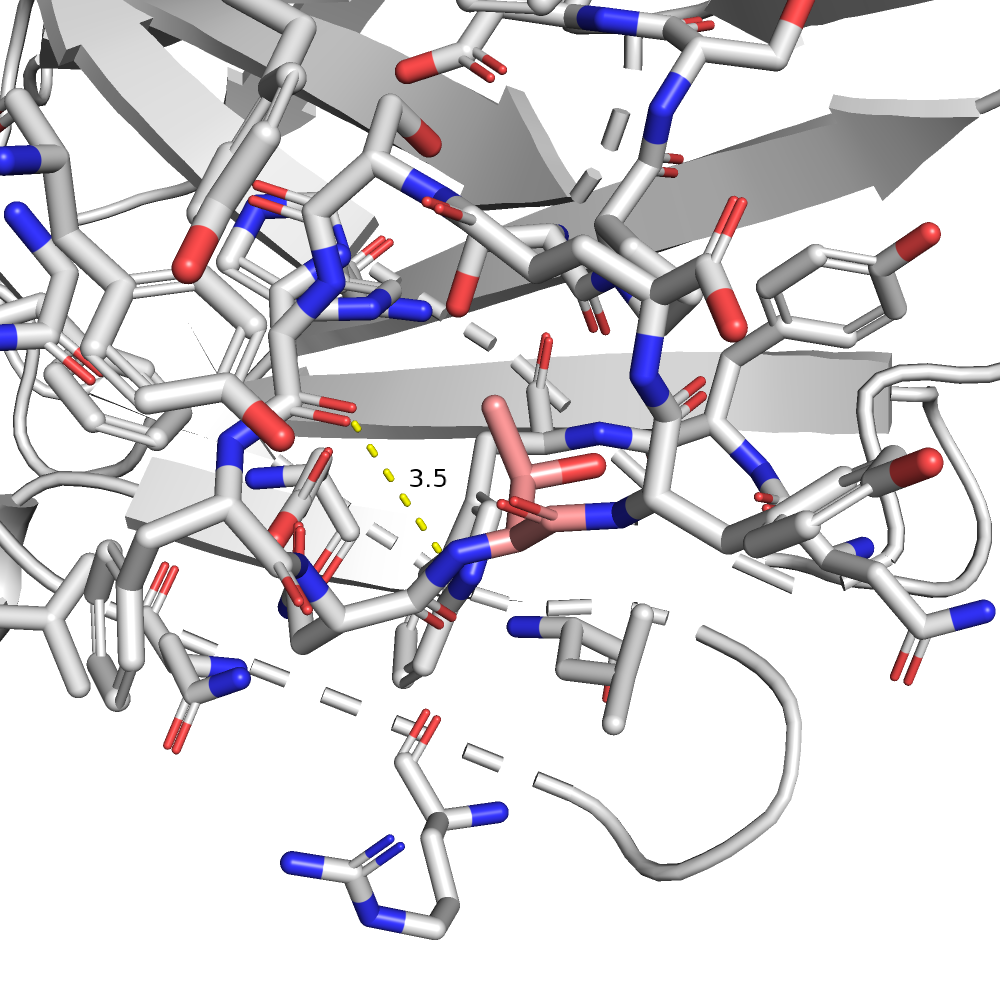 |
| ASN | 1/15 | 14.34 | 30.4% | 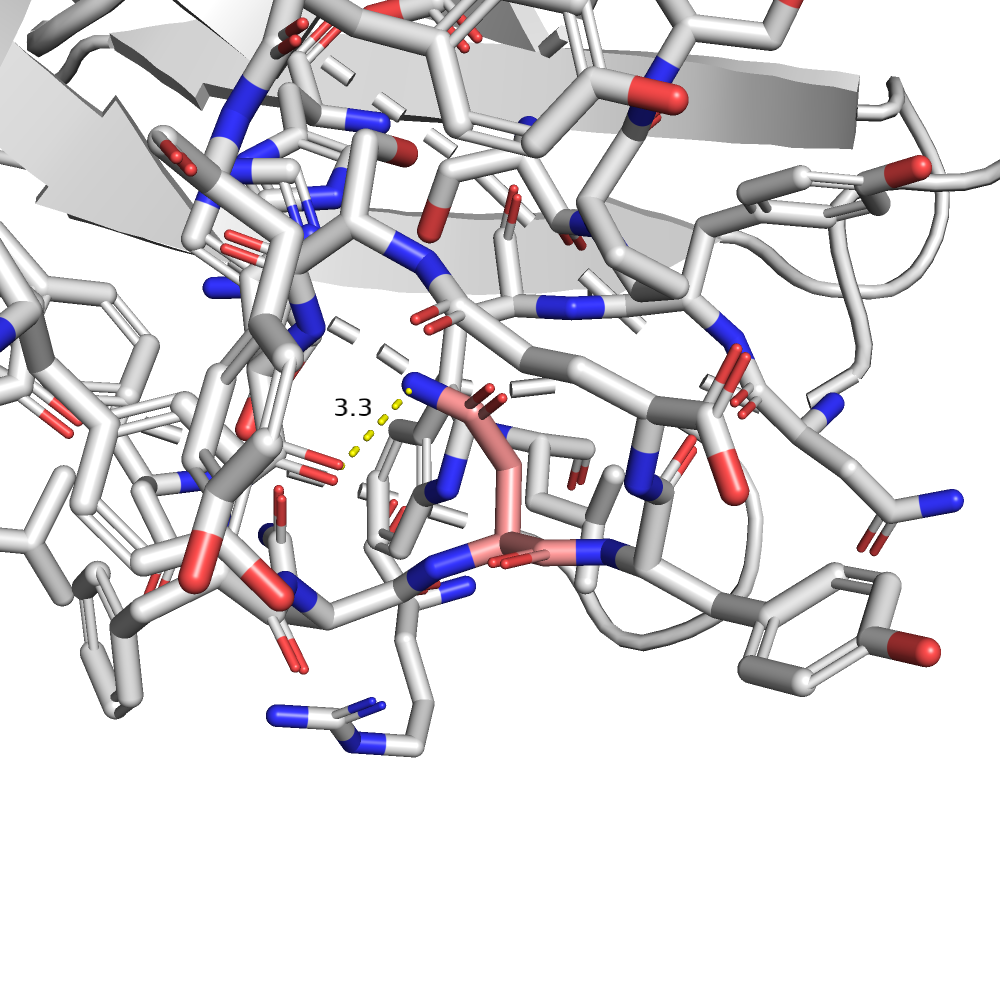 |
| MET | 2/10 | 13.78 | 27.8% | 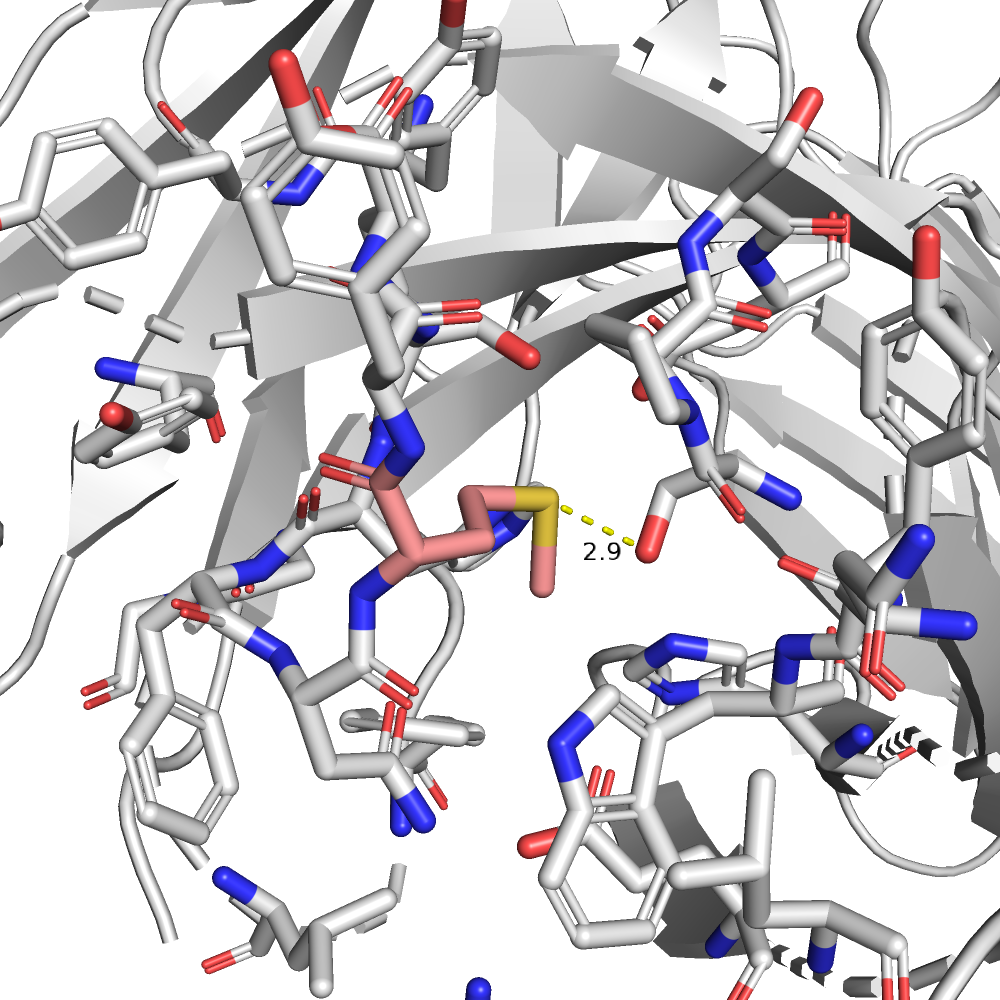 |
Работа над ошибками
Моя ошибка состояла в том, что я не сообразила, что нужно переключаться между ротамерами, и смотрела на первый попавшйся ротамер каждой аминокислоты. Первый попавшийся ротамер для глутамина и глутамата (про которые мне подсказали в ведомости) был плохим, поэтому я их не рассмотрела. Теперь я выбрала их наилучшие ротамеры и рассмотрела их.
| Аминокислота | Номер ротамера | Стерическая напряженность (strain) | Представленность ротамера | Изображение |
| GLU | 1/17 | 15.10 | 30.1% | 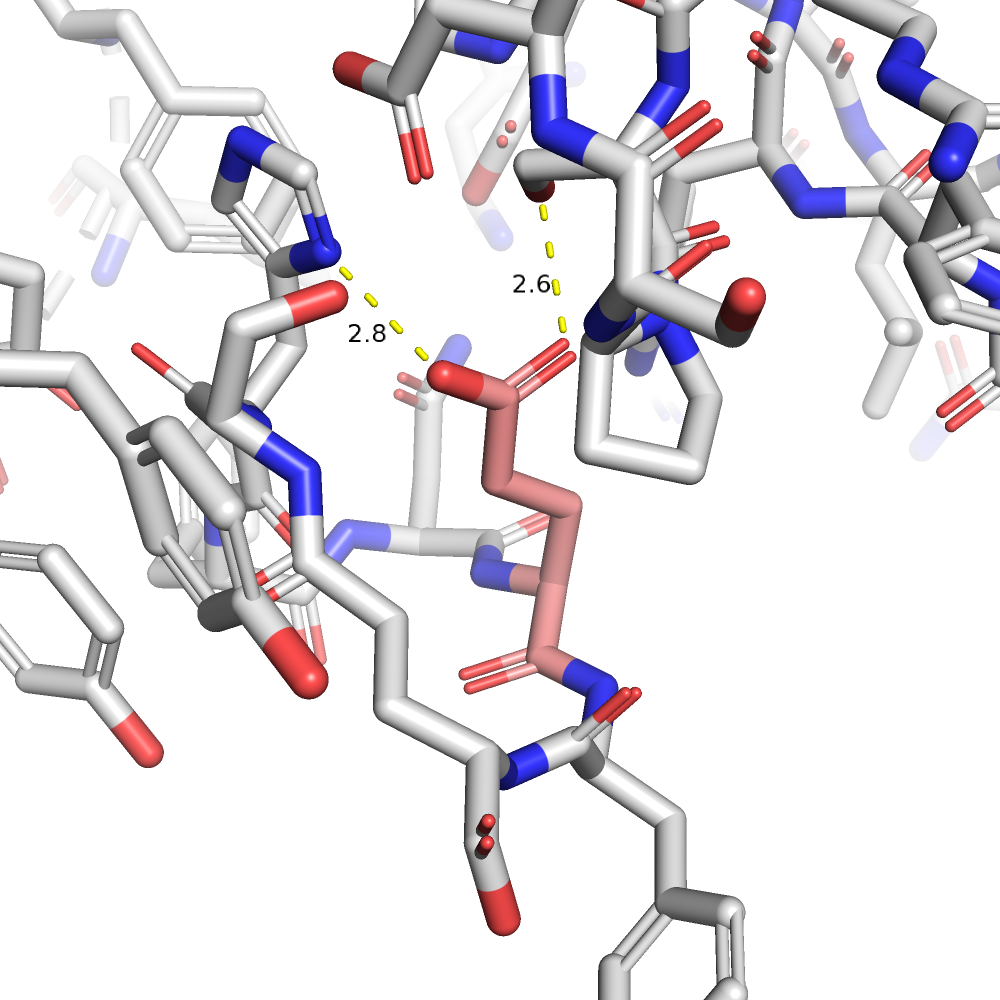 |
| GLN | 1/14 | 16.00 | 26.4% | 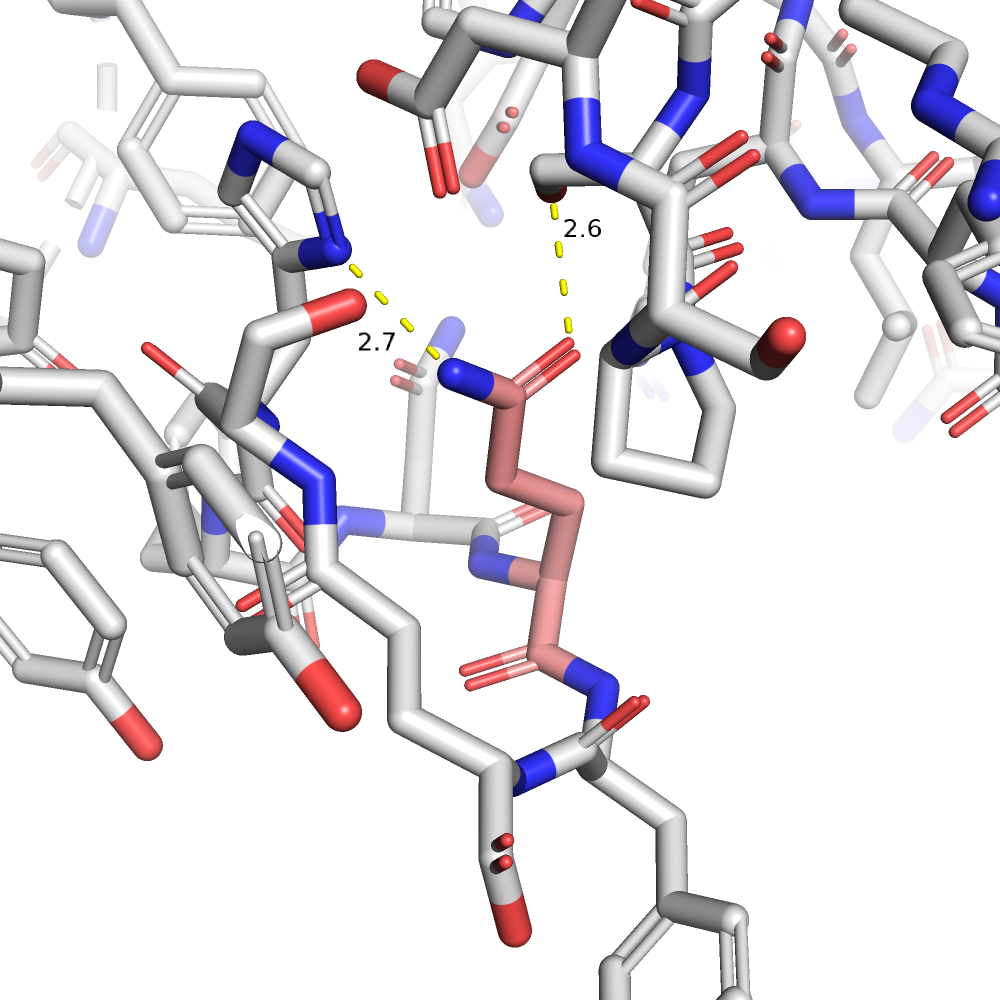 |
Здесь, конечно, картина получается интереснее, чем для цистеина, например, потому что и глутамин, и глутамат образут по две хорошие водородные связи с боковыми радикалами соседей. Для глутамата показатели напряженности и представленности ротамера чуть лучше, поэтому я остановлю выбор на нем.
Теперь рассмотрим структуру AB2 (код 0055, цепь С, позиция 3). Здесь хороших кандидатов оказалось гораздо меньше. Из трех наилучших, показаных в таблице ниже, я бы выбрала SER на роль исходного остатка, потому что для него и взаимодействий больше всего нашлось, и напряженность в сочетании с представленностью ротамера у него самые оптимальные.
| Аминокислота | Номер ротамера | Стерическая напряженность (strain) | Представленность ротамера | Изображение |
| PRO | 1/2 | 34.58 | 62.3% | 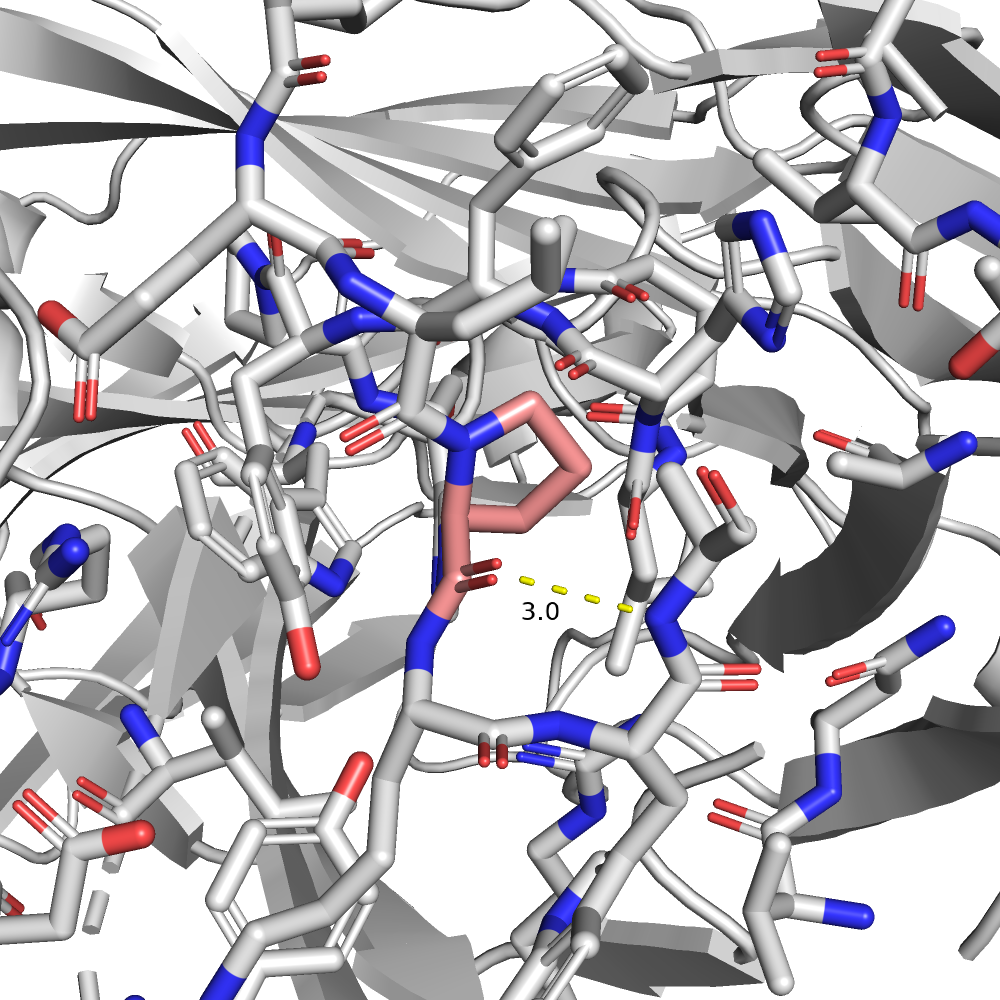 |
| SER | 1/3 | 16.69 | 41.9% | 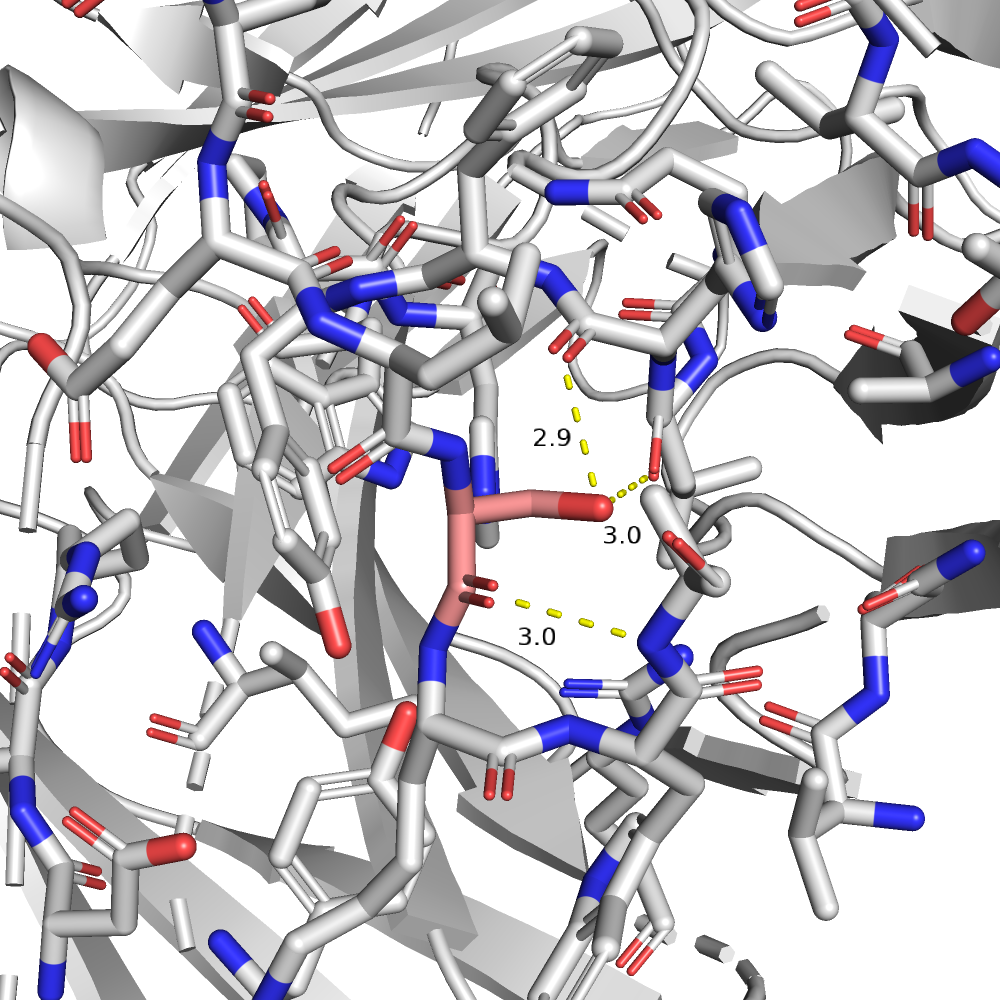 |
| VAL | 3/3 | 18.66 | 23.2% | 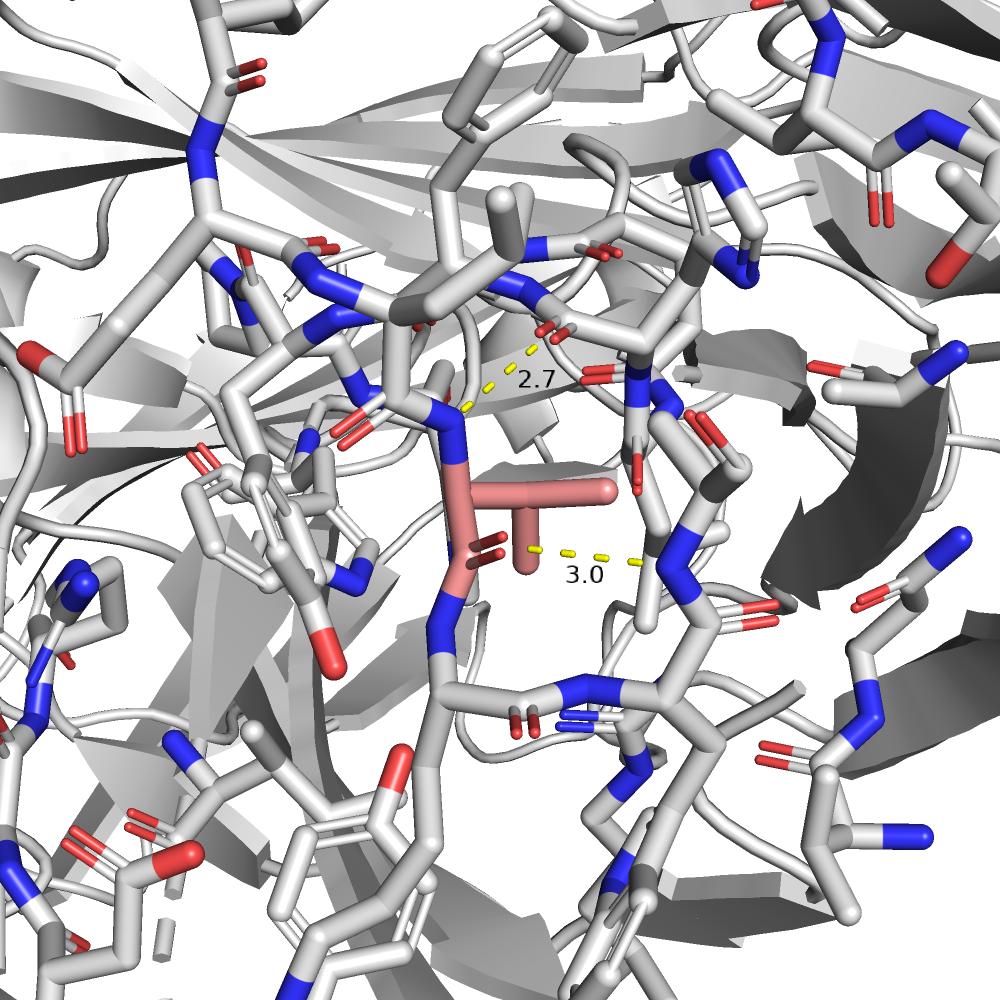 |
Работа над ошибками
Действительно, я упустила подсказку окружения: гистидин и аргинин предполагают в данной позиции отрицательно заряженную аминокислоту. Я попробовала подставить глутамат, но он оказался слишком большим и накладывался на радикалы соседей. Поэтому я рассмотрела аспартат, выбрала ротамер с наименьшим напряжением, и, конечно, получился вариант гораздо лучше, чем все, что я рассматривала до этого. Мой ответ: аспартат.
| Аминокислота | Номер ротамера | Стерическая напряженность (strain) | Представленность ротамера | Изображение |
| ASP | 3/9 | 14.63 | 8.5% | 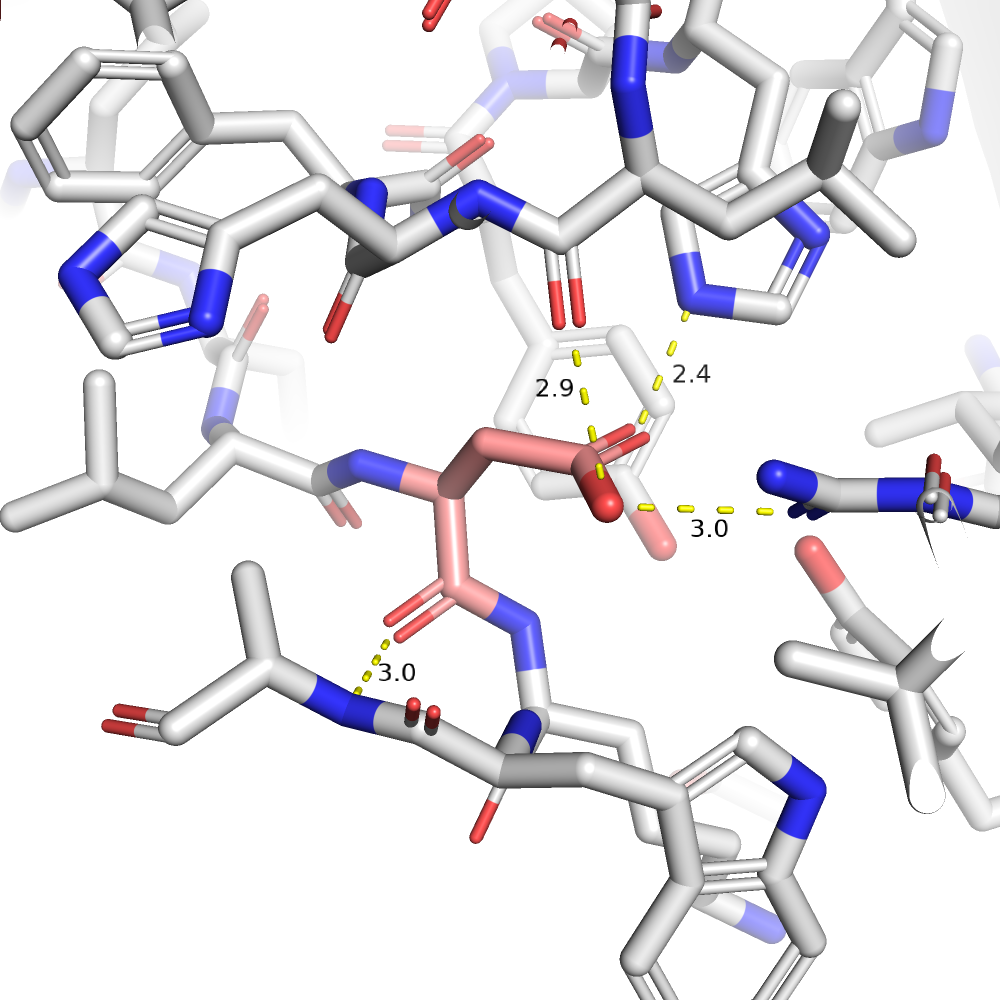 |