Практикум 2.Химическое строениа нуклеиновых кислот
Задание 1.
Были построены модели структур A-, B- и Z-формы ДНК с помощью инструментов пакета 3DNA. Структуры были построены на пятикратном повторении
нуклеотидов GATC
Далее представлены ссылки:
Задание2.
В программе Jmol была визуализированна структура В-формы ДНК. И рассмотрена большие и малые бороздки. Также, более подробно было рассмотрено
азотистое основание тимин. Ниже приведены имена атомов, смотрящих в направлении большой и малой бороздки.
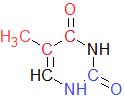
В сторону большой бороздки обращены атомы: C7,C5,C4,O4
В сторону малой бороздки обращены атомы:O2,N1,C2
Таблица. Спиральные параметры разных форм ДНК.
| |
A-форма |
И-форма |
*Z-форма |
|
| Тип спирали |
правая |
правая |
левая |
| Шаг спирали (A) |
28.02 |
40.68 |
43.5 |
| Число оснований на виток |
11 |
10 |
12 |
|
| Ширина большой бороздки(фосфоры азотистых оснований) |
17.1 (A-A) |
17.75 (A-T) |
15.17 (A-T) |
| Ширина малой бороздки |
7.98 (A-A) |
11.69 (A-T) |
7.19 (A-A) |
|
Задание 3.
В таблице ниже приведены средние значения торсионных углов для тРНК (PDB ID:1g59) и ДНК (PDB ID:1ddn)
| НК |
α |
β |
γ |
δ |
ε |
ζ |
χ |
| тРНК |
-53,18 |
42,49 |
51,81 |
95,51 |
-135,76 |
-61,16 |
-141,21 |
| Наиболее деформированный нуклеотид тРНК |
126,3 |
-151 |
-166,9 |
104,6 |
-119,8 |
-27,5 |
179,1 |
ДНК |
1,98 |
-79,92 |
-26,51 |
151,16 |
-108,02 |
-101,78 |
-113,72 |
| Наиболее деформированный нуклеотид ДНК |
90,3 |
-142,6 |
173,7 |
160,2 |
-80,1 |
-169 |
-115,8 |
Из полученных данных видно, что тРНК больше похожа на А форму ДНК
Задание 3.
Были определены водородны связи, образующие стебли во вторичной структуре тРНК цепи D.
Stem 1
Stem 1
30 (0.011) ....>B:.501_:[..G]G-----C[..C]:.572_:D<.... (0.010) |
31 (0.010) ....>D:.502_:[..G]G-**--U[..U]:.571_:D<.... (0.016) |
32 (0.010) ....>D:.503_:[..C]C-----G[..G]:.570_:D<.... (0.008) |
33 (0.009) ....>D:.504_:[..C]C-----G[..G]:.569_:D<.... (0.006) |
34 (0.009) ....>D:.505_:[..C]C-----G[..G]:.568_:D<.... (0.009) |
35 (0.008) ....>D:.506_:[..C]C-----G[..G]:.567_:D<.... (0.008) |
36 (0.009) ....>D:.507_:[..A]A-----U[..U]:.566_:D<.... (0.015) |
Stem 2
51 (0.010) ....>D:.510_:[..G]G-----C[..C]:.525_:D<.... (0.012) |
52 (0.018) ....>D:.511_:[..U]U-----A[..A]:.524_:D<.... (0.009) |
53 (0.011) ....>D:.512_:[..C]C-----G[..G]:.523_:D<.... (0.008) |
54 (0.016) ....>D:.513_:[..U]U-*---G[..G]:.522_:D<.... (0.007) |
Stem 3
44 (0.011) ....>D:.538_:[..A]A-**--C[..C]:.532_:D<.... (0.013) |
45 (0.013) ....>D:.539_:[..G]G-----C[..C]:.531_:D<.... (0.008) |
46 (0.008) ....>D:.540_:[..G]G-----C[..C]:.530_:D<.... (0.009) |
47 (0.010) ....>D:.541_:[..C]C-----G[..G]:.529_:D<.... (0.008) |
48 (0.010) ....>D:.542_:[..C]C-----G[..G]:.528_:D<.... (0.006) |
49 (0.009) ....>D:.543_:[..G]G-----C[..C]:.527_:D<.... (0.012) |
50 (0.009) ....>D:.544_:[..A]A-**--G[..G]:.526_:D<.... (0.009) |
Stem 4
37 (0.011) ....>D:.549_:[..G]G-----C[..C]:.565_:D<.... (0.012) |
38 (0.008) ....>D:.550_:[..G]G-----C[..C]:.564_:D<.... (0.010) |
39 (0.009) ....>D:.551_:[..G]G-----C[..C]:.563_:D<.... (0.014) |
40 (0.007) ....>D:.552_:[..G]G-----C[..C]:.562_:D<.... (0.011) |
41 (0.011) ....>D:.553_:[..G]G-----C[..C]:.561_:D<.... (0.009) |
Неканонические пары найденные в тРНК:
31 (0.010) ....>D:.502_:[..G]G-**--U[..U]:.571_:D<.... (0.016) |
43 (0.019) ....>D:.555_:[..U]U-**+-G[..G]:.517_:D<.... (0.022) x
44 (0.011) ....>D:.538_:[..A]A-**--C[..C]:.532_:D<.... (0.013) |
50 (0.009) ....>D:.544_:[..A]A-**--G[..G]:.526_:D<.... (0.009) |
54 (0.016) ....>D:.513_:[..U]U-*---G[..G]:.522_:D<.... (0.007) |
56 (0.010) ....>D:.514_:[..A]A-**--A[..A]:.521_:D<.... (0.010) |
|
Связи, которые не участвуют в формировании стеблей:
42 (0.017) ....>D:.554_:[..U]U-**--A[..A]:.558_:D<.... (0.008) |
43 (0.019) ....>D:.555_:[..U]U-**+-G[..G]:.517_:D<.... (0.022) x
55 (0.015) ....>D:.508_:[..U]U-**--A[..A]:.546_:D<.... (0.008) |
56 (0.010) ....>D:.514_:[..A]A-**--A[..A]:.521_:D<.... (0.010) |
57 (0.010) ....>D:.515_:[..G]G-**+-C[..C]:.548_:D<.... (0.008) x
58 (0.012) ....>D:.518_:[..G]G-----C[..C]:.556_:D<.... (0.032) +
В stacking.pdb файле была найдена модель, обладающая наибольшей площадью перекрывания (13,51). GC/GU

А также с наименьшей площадью перекрывания (0,0) UA/CG

` |



