Практикум 3. Построение и анализ дерева, содержащего паралоги
В данном практикуме было реконструировано дерево по гомологичным последовательностям белков.
Паралоги - гомологичные белки из одного организма. Возникают благодаря генной дупликации.
Ортологи- гомологичные белки, разделение общего предка которых произошло в результате видообразования. Таким образом, такие белки находятся в разных организмах.
При помощи создания базы данных из протеомов бактерий, использованных в практикуме 1, были найдены достоверные гомологи для белка :
CLPX_ECOLI. при пороги E-value = 0.001
Таблица 1. Расшифровки белков
| Сокращение |
Название белка |
| CLPX |
ATP-dependent Clp protease |
| HSLU |
ATP-dependent protease ATPase subunit |
| RUVB |
Holliday junction ATP-dependent DNA helicase |
| Q9KSW2 |
ATP-dependent Clp protease |
| Q2K4M2 |
ATP-dependent zinc metalloprotease |
| FTSH |
ATP-dependent zinc metalloprotease |
| Q9KU86 |
ATP-dependent zinc metalloprotease |
Затем было построено само дерево, при помощи программы Mega (метод:Neighbor-Joining) Ниже на Рис.1 представлено дерево.
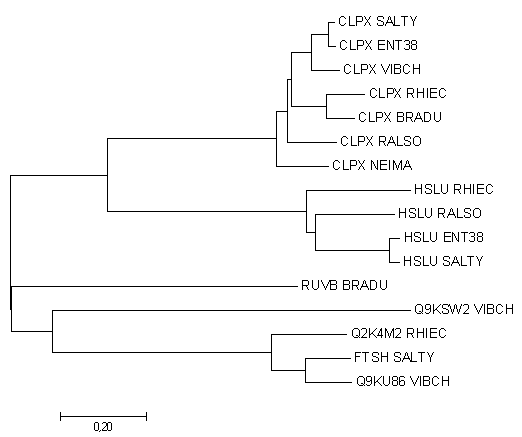
Рис.1
Мы видим, что группа белков CLPX является ортологичной, так как она образовалась в процессе видообразования. Еще одной такой группой
можно назвать HSLU. При этом для таких организмов как, RALSO, RHIEC,SALTY и ENT38, данные белки являются паралогами и образовались в результате дупликации.
Еще одним примером паралогов можно назвать белки Q9KU86 и Q9KSW2 из организма VIBCH, которые также образовались в результате дупликации.