Практикум 9. Поиск по PDB и содержание PDB файлов
1.Примеры атомов с коэффициентом заполнения (Occupancy), не равным 1
PDB ID: 3QM5 (Разрешение 0.91 ангстрем) Структура оксимиоглобина.
Например позиция 71 - лейцин. (2 альтернативных положения) Occupancy = 0.4 и 0.6 alter code - A,B
ATOM 625 N ALEU A 71 4.282 10.255 7.655 0.40 10.35 N
ATOM 626 N BLEU A 71 4.775 9.990 7.985 0.60 8.33 N
ATOM 627 CA ALEU A 71 5.281 10.848 8.550 0.40 11.37 C
ATOM 628 CA BLEU A 71 5.788 10.461 8.923 0.60 9.85 C
На рисунке 1 изображен данный лейцин, правее показано положение c оссupancy 0.6, левее с 0.4.
Боковой радикал данного аминокислотного остатка напрвлен в сторону альфа-спирали.
При визуализации также была наложена электронная плотность. Как видно по рисунку при уровне подрезки 1,
электронная плотность вокруг остатка занимает довольно большое пространство, покрывая оба положения.
В связи с тем, что радикал лейцина относится к алифатическим и не имеет в своем составе например
атомов кислорода или азота, расшифровка его положения может быть не однозначна.
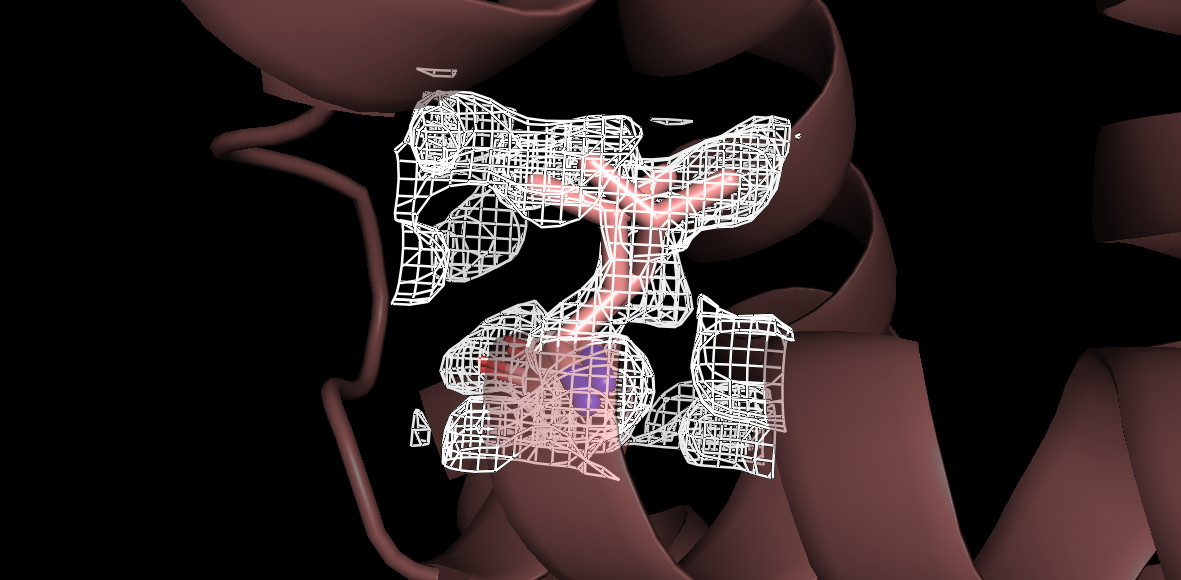
Рисунок 1. Альтернативные положения Leu 71 в структуре 3QM5 .
2.Не расшифрованные аминокислотные остатки
PDB ID: 3VRA (Разрешение 3.44 ангстрема) Структура митохондриальной родохинол-фумарат редуктазы.
Поле: REMARK 465
Часть записи:
REMARK 465 M RES C SSSEQI
REMARK 465 MET A 1
REMARK 465 LEU A 2
REMARK 465 ARG A 3
REMARK 465 ALA A 4
REMARK 465 VAL A 5
REMARK 465 ARG A 6
REMARK 465 ALA A 7
REMARK 465 LEU A 8
REMARK 465 ILE A 9
REMARK 465 CYS A 10
REMARK 465 ARG A 11
REMARK 465 ILE A 12
REMARK 465 GLY A 13
REMARK 465 ALA A 14
REMARK 465 ARG A 15
REMARK 465 ARG A 16
3. Структура белка, для которой не совпадают последовательность природного белка из Uniprot и
последовательность белка, который кристаллизовали
PDB ID: 6IQL. Структура дофаминового рецептора и цитохрома
В PDB представлены Uniprot ID:P51436, P0ABE7 (для рецептора мыши и цитохрома
E.coli.)
Данный рецептор у мыши состоит из 387 аминокислотных остатков. Цитохром из 128 аминокислотных остатков.
В качестве отличий в поле SEQADV отмечены: тэг для экспрессии, мутация и линкер
Ниже приведены строчки для А субъединицы (А и В идентичны).
DBREF 6IQL A 23 218 UNP P51436 DRD4_MOUSE 23 218
DBREF 6IQL A 1001 1105 UNP P0ABE7 C562_ECOLX 23 127
DBREF 6IQL A 304 387 UNP P51436 DRD4_MOUSE 304 387
SEQADV 6IQL ASP A 13 UNP P51436 EXPRESSION TAG
SEQADV 6IQL TYR A 14 UNP P51436 EXPRESSION TAG
SEQADV 6IQL LYS A 15 UNP P51436 EXPRESSION TAG
SEQADV 6IQL ASP A 16 UNP P51436 EXPRESSION TAG
SEQADV 6IQL ASP A 17 UNP P51436 EXPRESSION TAG
SEQADV 6IQL ASP A 18 UNP P51436 EXPRESSION TAG
SEQADV 6IQL ASP A 19 UNP P51436 EXPRESSION TAG
SEQADV 6IQL LYS A 20 UNP P51436 EXPRESSION TAG
SEQADV 6IQL GLU A 21 UNP P51436 EXPRESSION TAG
SEQADV 6IQL PHE A 22 UNP P51436 EXPRESSION TAG
SEQADV 6IQL TRP A 121 UNP P51436 PHE 121 ENGINEERED MUTATION
SEQADV 6IQL ARG A 181 UNP P51436 CYS 181 ENGINEERED MUTATION
SEQADV 6IQL ILE A 201 UNP P51436 PRO 201 ENGINEERED MUTATION
SEQADV 6IQL TRP A 1007 UNP P0ABE7 MET 29 ENGINEERED MUTATION
SEQADV 6IQL ILE A 1102 UNP P0ABE7 HIS 124 ENGINEERED MUTATION
SEQADV 6IQL LEU A 1106 UNP P0ABE7 LINKER
SEQADV 6IQL ALA A 317 UNP P51436 PRO 317 ENGINEERED MUTATION
В поле missing residues попало довольно большое количество аминокислотных остатков:
для А субъединицы - 55 аминокислотных остатков
(для последоватености белка мыши :с 13 - 30, 136 - 143, 178 - 182, 379 - 387
для бактериальной: 1044 - 1058).
REMARK 465 M RES C SSSEQI
REMARK 465 ASP A 13
REMARK 465 TYR A 14
REMARK 465 LYS A 15
REMARK 465 ASP A 16
REMARK 465 ASP A 17
REMARK 465 ASP A 18
REMARK 465 ASP A 19
REMARK 465 LYS A 20
REMARK 465 GLU A 21
REMARK 465 PHE A 22
REMARK 465 THR A 23
REMARK 465 GLY A 24
REMARK 465 ALA A 25
REMARK 465 GLY A 26
REMARK 465 LEU A 27
REMARK 465 GLY A 28
REMARK 465 GLY A 29
REMARK 465 ALA A 30
4. Наибольший и наименьший B-фактор в структуре.
PDB ID: 6IUR
Наибольший:
ATOM 18571 OE2 GLU F 565 205.357 -60.276 191.400 1.00 221.13 - B-фактор 221.13
Наименьший:
ATOM 792 CB VAL A 109 209.192 27.343 171.011 1.00 39.59 - В-фактор 39.59
Объяснение аномально большого значения B-фактора.
Как известно, определить достоверно глутамат бывает затруднительно.
Более того, он может входить в состав подвижной петли,
что будет приводить к неоднозначности положения остатка, тем самым понижая уровень электронной плотности.
И соответсвенно B-фактор будет большим.
На рисунке 2 изображен общий вид белка, на котором видно, что данный глутамат (изображен желтыми шариками в правом
вверхнем углу) располагается на самом краю цепи.
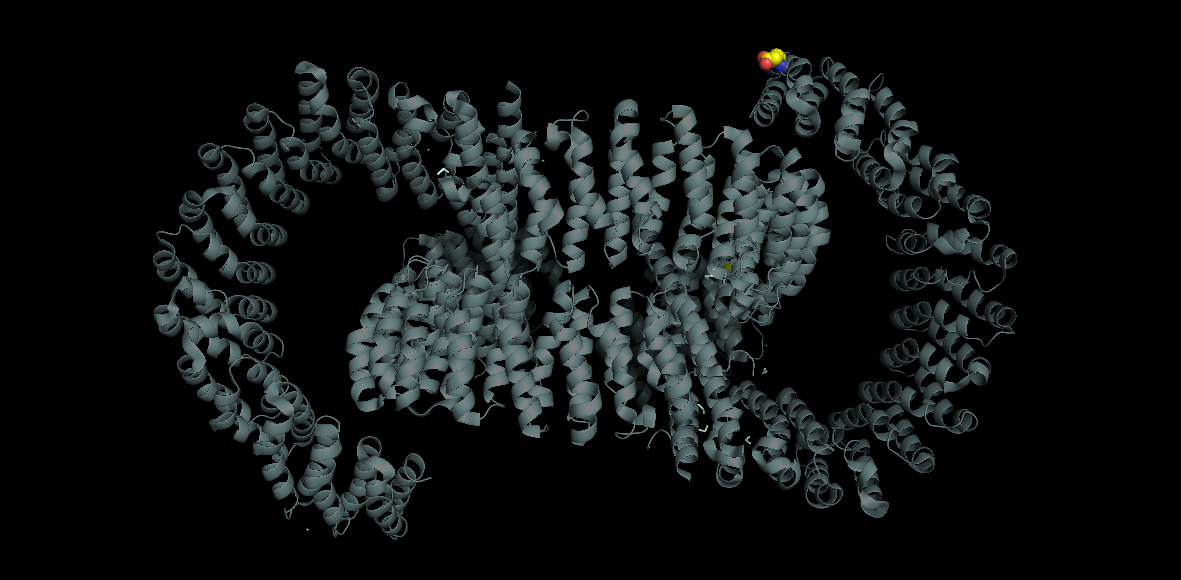
Рисунок 2. Общий вид белка PDB ID: 6IUR и Glu 565 .

