Жукова Мария
Факультет биоинженерии и биоинформатики Московского государственного университета имени М. В. Ломоносова, Москва, Россия
В данной работе представлен мини-обзор генома и протеома бактерии Sutcliffiella horikoshii : описаны результаты, полученные с помощью сервиса Google Sheets и программы на Python.
Sutcliffiella horikoshii - грамположительная, спорообразующая, термо- и алкалафильная бактерия, является облигарным аэробом [4], впервые описана, как Bacillus horikoshii. Названа в честь японского микробиолога Коки Хорикоши, который внёс фундаментальный вклад в изучение алкалифильных бактерий [1].
Бактерия принадлежит к домену Bacteria, филуму Bacillota, классу Bacilli, отряду Caryophanales, семейству Bacillaceae, роду Sutcliffiella, виду Sutcliffiella horikoshii [1].
Для написания мини-обзора были использованы файлы с последовательностью генома и таблица особенностей бактерии Sutcliffiella horikoshii из базы NCBI [2]. Статистические расчеты, построение таблиц, диаграмм, использование фильтров, функций (СЧЁТЕСЛИ, МИН , МАКС, СУММ, FILTER. ПОИСК, UNIQUE) были выполнены с помощью сервиса Google Sheets. Подсчет количества нуклеотидов по репликонам был произведен с помощью скрипта, написанного на Python, затем полученные данные были занесены в таблицу (1) на лист nucleotides, и построена диаграмма, иллюстрирующая процентное содержание нуклеотидов в хромосоме и плазмиде. Итоговые таблицы и код на Python представлены в сопроводительных материалах (1-3).
Геном бактерии содержит в себе одну хромосому, состоящую из 4277585 пар нуклеотидов, и одну плазмиду, состоящую из 18297 пар нуклеотидов.
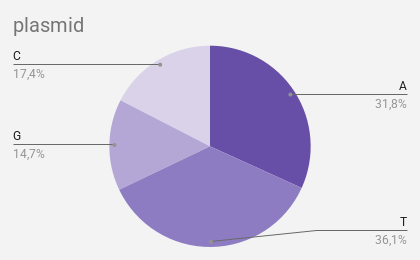
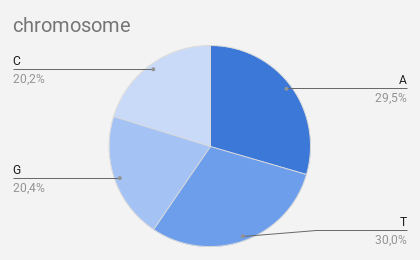
В таблице 1 представлено распределение нуклеотидов A, T, G, C по репликонам. Других нуклеотидов в геноме не встречается. На диаграммах (рисунок 1) представлено процентное содержание каждого нуклеотида в хромосоме и плазмиде. Процент GC-оснований в хромосоме составляет 40,6%, такое процентное содержание характерно для бактерий рода Sutcliffiella [3]. Процентное содержание GC-оснований в плазмиде равен 32,1%.
| genomic_accession | seq_type | A | T | G | C |
|---|---|---|---|---|---|
| NZ_CP020880.1 | chromosome | 1260321 | 1281219 | 872160 | 863885 |
| NZ_CP020881.1 | plasmid | 5815 | 6609 | 2682 | 3191 |
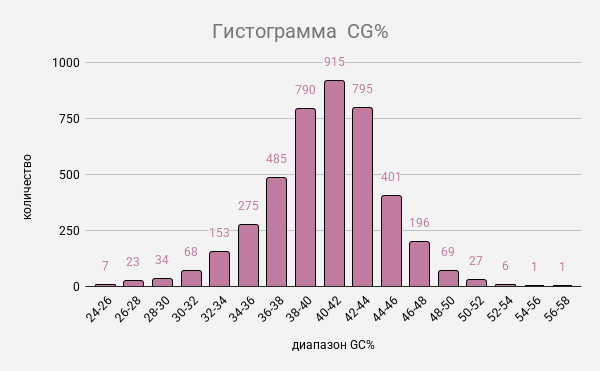
На рисунке 2 представлена гистограмма GC% (процентное содержание гуанина (G) и цитозина (C) среди всех нуклеотидов) по CDS с шагом 2. Наиболее распространенные CDS c GC составом равным 40-42%. Редко встречаются CDS c GC составом 54-58%.
Для каждого репликона было посчитано количество генов белков и различных генов РНК. Всего генов у бактерии Sutcliffiella horikoshii на хромосоме 4329, а на плазмиде лишь 19.
| genomic_accession | seq_type | protein_coding | pseudogene | tRNA | rRNA | tmRNA | ncRNA | SRP_RNA | RNase_P_RNA |
|---|---|---|---|---|---|---|---|---|---|
| NZ_CP020880.1 | chromosome | 4194 | 33 | 73 | 24 | 1 | 2 | 1 | 1 |
| NZ_CP020881.1 | plasmid | 19 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
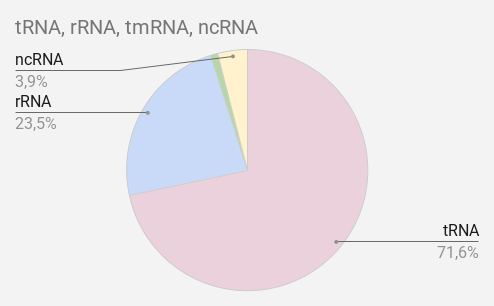
На основе данных из колонки # feature, листа feature_table, таблицы (1) было найдено количество различных типов РНК (tRNA, rRNA, tmRNA, ncRNA) и построена диаграмма (рисунок 3), иллюстрирующая их процентное соотношение. Можно заметить, что транспортные РНК (тРНК) являются наиболее распространённым типом РНК.
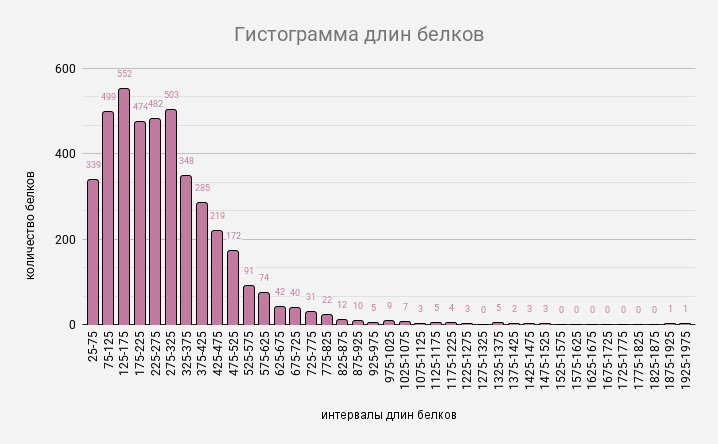
На рисунке 4 представлено распределение длин белков бактерии Sutcliffiella horikoshii с шагом 50. Наибольшая часть белков бактерии имеют длину от 125 до 175 остатков аминокислот. Минимальная длина белка - 26 аминокислотных остатков, а максимальная - 1964.
Было установлено, чтo в геноме бактерии Sutcliffiella horikoshii закодировано 4213 белков. Распределение белков генома по категориям (гипотетические, рибосомные, транспортные, другие) представлено в таблице 3. Построена диаграмма, иллюстрирующая процентное соотношение встречаемости категорий белков. Можно заметить, что в геноме Sutcliffiella horikoshii значительную долю (14.1%) занимают гипотетические белки, называются они так, потому что про них неизвестно ничего, кроме предполагаемой аминокислотной последовательности.
| hypothetical protein | transport | ribosomal | other | total |
|---|---|---|---|---|
| 592 | 351 | 62 | 3208 | 4213 |
Доля транспортных белков в протеоме бактерии составляет 8,3% (рисунок 6). В ходе работы было определено количество каждого уникального названия белка, на основе полученных данных составлена таблица 4, в которой представлены белки, встречающиеся в протеоме два и более раза. Среди транспортных белков бактерии Sutcliffiella horikoshii преобладает ABC transporter ATP-binding protein. Данный белок отвечает за импорт одного или нескольких различных субстратов, включая ионы и полиамины, с помощью энергии, получаемой при гидролизе АТФ [5]. Второй по распространенности транспортный белок -ABC transporter permease- участвует в переносе одного или нескольких субстратов, такие как сахара, ионы, аминокислоты, пептиды и другие вещества [6].
| название белка | количество |
|---|---|
| ABC transporter ATP-binding protein | 54 |
| ABC transporter permease | 34 |
| MFS transporter | 27 |
| ABC transporter permease subunit | 12 |
| sugar ABC transporter permease | 11 |
| ABC transporter substrate-binding protein | 9 |
| carbohydrate ABC transporter permease | 9 |
| GerAB/ArcD/ProY family transporter | 8 |
| DMT family transporter | 7 |
| iron ABC transporter permease | 7 |
| efflux RND transporter permease subunit | 5 |
| ABC-2 family transporter protein | 4 |
| dipeptide ABC transporter ATP-binding protein | 4 |
| glutathione ABC transporter substrate-binding protein | 4 |
| BCCT family transporter | 3 |
| nuclear transport factor 2 family protein | 3 |
| AI-2E family transporter | 3 |
| iron-siderophore ABC transporter substrate-binding protein | 3 |
| nickel ABC transporter permease | 3 |
| chromate transporter | 3 |
| ECF transporter S component | 3 |
| ABC transporter transmembrane domain-containing protein | 3 |
| amino acid ABC transporter permease | 3 |
| amino acid ABC transporter ATP-binding protein | 3 |
| MATE family efflux transporter | 3 |
| oligopeptide ABC transporter permease | 3 |
| metal ABC transporter permease | 3 |
| energy-coupling factor ABC transporter ATP-binding protein | 2 |
| branched-chain amino acid transport system II carrier protein | 2 |
| efflux RND transporter periplasmic adaptor subunit | 2 |
| glucose-specific PTS transporter subunit IIBC | 2 |
| LysE family transporter | 2 |
| cation diffusion facilitator family transporter | 2 |
| biotin transporter BioY | 2 |
| sn-glycerol-3-phosphate ABC transporter ATP-binding protein UgpC | 2 |
| sugar ABC transporter ATP-binding protein | 2 |
| AbgT family transporter | 2 |
| multidrug efflux SMR transporter | 2 |
| branched-chain amino acid ABC transporter permease | 2 |
| BMP family ABC transporter substrate-binding protein | 2 |
| peptide ABC transporter substrate-binding protein | 2 |
| ZIP family metal transporter | 2 |
| phosphonate ABC transporter, permease protein PhnE | 2 |
| sodium-dependent transporter | 2 |
| PTS glucose transporter subunit IIA | 2 |
| transporter substrate-binding domain-containing protein | 2 |
| metal ABC transporter ATP-binding protein | 2 |
| zinc ABC transporter substrate-binding protein | 2 |
| MetQ/NlpA family ABC transporter substrate-binding protein | 2 |
| methionine ABC transporter permease | 2 |
| EamA family transporter | 2 |
В работе был частично изучен геном бактерии Sutcliffiella horikoshii. Составлена таблица, иллюстрирующая распределение нуклеотидов A, T, G, C по репликонам, по данным из таблицы построены диаграммы частоты нуклеотидов в хромосоме и плазмиде, построена гистограмма GC% по CDS, составлена таблица - число генов белков и генов разных типов РНК для каждого репликона, на основе таблицы 2 построена диаграмма частоты генов, кодирующих различные типы РНК, построена гистограмма длин белков, изучено распределение белков по категориям.
(2)CDS frome genome of Sutcliffiella horikoshii
[2]NCBI-Sutcliffiella horikoshii
[4]https://www.bacdive.dsmz.de/strain/1142
[5]https://www.ncbi.nlm.nih.gov/Structure/sparcle/archview.html?archid=11485425#qinfo
[6]https://www.ncbi.nlm.nih.gov/Structure/sparcle/archview.html?archid=10008345