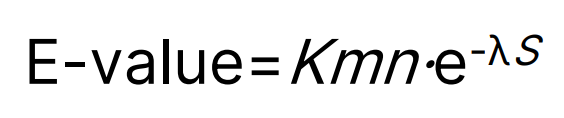Поиск был произведен по последовательности белка в fasta-формате.
Database: UniProtKB/Swiss-Prot(swissprot)
Algoritm: blastp(protein-protein BLAST)
Max target sequences: 500 (максимальное число находок было выставлено таким, чтобы были отображены все возможные гомологичные белки, а не только первые 100)
Expect threshold: 0.05 (Пороговое значение E-value)
Word size: 5
Matrix: BLOSUM62
Gap Costs: за открытие: 11 За продолжение: 1
Все остальные параметры были оставлены по умолчанию.
В результате было найдено 257 белковых последовательностей. Текстовую выдачу программы можно посмотреть здесь.
Для построения множественного выравнивания были выбраны первые 7 белков и исходный белок.
Проект выравнивания JalviewВсе 8 белков вероятно гомологичны друг другу, т.к есть много гомологичных участков (консерваивны на 100%): 7-8, 12-14, 28-31, 42-44, 46-47, 49-51, 54-55, 66-73, 82-83, 86-88, 90-91, 99-100, 117-119, 123-124, 154-159, 170-171, 177-178, 181-184, 187-188.
С помощью запроса (taxonomy_id:10239) AND (protein_name:polyprotein) было найдено 553624 полипротеина вируса. Был выбран белок Polyprotein P2A.
ID: P2A_CFMVN
Amino acids: 568 AA
AC: Q89504; Q0PW24; Q76PL5;
Название вируса: Cocksfoot mottle virus
Я выбрала зрелый белок Serine protease. Координаты белка 131..319. Средствами EMBOSS была вырезана последовательность этого зрелого белка. При запуске BLAST была получена данная текстовая выдача.
Далее было выполнено множественное выравнивание последовательности выбранного зрелого белка и всех белков в выдаче. Из выравнивания были удалены беки с AC Q83470 и P21405, у которых все идентичные участки были очень непродолжительные.
Проект выравнивания JalviewОставщиеся белки вероятно гомологичны друг другу, т.к можно выделить участки с 100% консервативностью: 15-19, 26-27, 43-44, 46-48, 61-62, 80-83, 87-89, 93-94, 95-97, 101-102, 104-106, 125-128, 146-150, 152-154, 160-161, 173-175.
При применении к прошлому поиску фильтра по огрганизмам, ограничивая поиск вирусами (Viruses), список находок изменился (текстовая выдача).Список находок изменился, добавился еще один белок(E-Value=0.004). E-Value всех находок из предыдушего запроса изменился (таблица 1).
| AC | без применения фильтра по организму | с применением фильтра по организму |
|---|---|---|
| Q89504.2 | 6e-135 | 3e-136 |
| Q0PW25.1 | 1e-128 | 6e-130 |
| Q83470.2 | 2e-25 | 7e-27 |
| P21405.2 | 2e-25 | 1e-26 |
| O72157.2 | 7e-23 | 3e-24 |
| O73564.2 | 9e-23 | 4e-24 |
Расмотрим находку Replicase polyprotein P2AB его E-value изменился с 2e-25 до 1e-26. Е-value вычисляется по формуле(рисунок 1), где n — размер базы данных. Поэтому для того, чтобы узнать долю вирусных белков в Swiss-Prot мы можем поделить значение E-value при использовании фильтра по организмам на значение E-value до применения фильтра по организмам(когда поиск производился по всему Swiss-Prot): 1e-26/2e-25=0.05(5%).Можно сделать вывод, что доля вирусных белков в Swiss-Prot примерно равна 5%