- Создание паттернов аминокислотных последовательностей
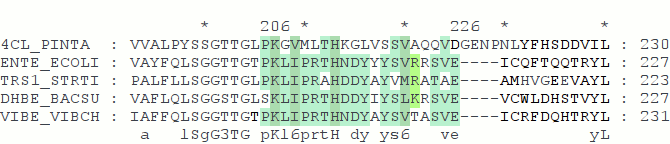
Выбранный участок выравнивания DhbE и его гомологов
Паттерн своими руками
| Характеристика паттерна | Паттерн | В скольких последовательностях банка Swiss-Prot найден мотив, удовлетворяющий паттерну? | Все ли последовательности из Вашего выравнивания найдены? |
| Фрагмент последовательности | SKLIPRTHDDYIYSLKRSVE | 1 | DHBE_BACSU |
| Сильный | [SP]-K-[LG]-[IV]-[PM]-[LR]-[TA]- H-[DNK]-[DG]-[YL]-[AYVI]-[YS]- [SV]-[LVM]-[KRAT]-[RAQ]-[STQ]-[AV]-[DE] | 6 | Все |
| Средний | [SP]-K-[LG]-[IVLA]-[PM]-[LR]-[TA]-H-X-{VFALIM}-[YL]-X-[YST]-[SV]-[LVM]-{DEF}-X(2)-[AV] | 18 (20) | Все |
| Слабый | K-[LG]-[IVLA]-X-{P}-X-H-{W}-X(3)-[STY]-X-[LVM] | 345 | Все |
Комментарий:
Я использовала фрагмент последовательности длиной 20 остатков, чтобы такой паттерн с большой вероятностью нашелся только в DhbE. В сильный паттерн я включила только все вариации из данного выравнивания, кроме выбранных мной для выравнивания белков нашелся ещё только один: EntE из E.&nsbp;coli O157:H7(в выравнивании использовался белок EntE из E.&nsbp;coli K12).
Средний паттрен умеет находить 20 гомологов.
Слабый паттерн находит много возможных гомологов, но также белки вроде ДНК- и актин-связывающих, участвующих в вакуолярном транспорте, рецепторов, ДНК-полимеразы III, EF-Tu из хлоропластов, ГАФД-дегидрогеназу, субъединицы вакуолярной АТФ-азы и митохондриальные транслокаторы Т_Т
Создать нечно среднее между средним и салбым паттернам у меня не получилось. Как не ужесточаю условия, все равно что-нибудь вроде полимеразы остается, и довольно большой процент. Возможно, стоит выбрать другую последовательность для паттерна.
Программа fuzzpro и сервер ExPASy дают разное количество белков.
- Все описанные в PROSITE мотивы в заданном белке DHBE_BACSU
Мотивы в DhbE
| Идентификатор документа PROSITE (AC) | Название мотива | Краткое описание мотива | Тип подписи (паттерн, профиль) | Паттерн (регулярное выражение) | Специфична ли подпись? | Сколько мотивов нашлось в белке? |
| PS00455 | AMP_BINDING | Putative AMP-binding domain signature | паттерн | [LIVMFY]-{E}-{VES}-[STG]- [STAG]-G-[ST]-[STEI]- [SG]-x-[PASLIVM]-[KR] | специфична | 1 |
| PS00005 | PKC_PHOSPHO_SITE | Protein kinase C phosphorylation site | паттерн | [ST]-x-[RK] | неспецифична | 10 |
| PS00008 | MYRISTYL | N-myristoylation site | паттерн | G-{EDRKHPFYW}-x(2)-[STAGCN]-{P} | неспецифична | 5 |
| PS00006 | CK2_PHOSPHO_SITE | Casein kinase II phosphorylation site | паттерн | [ST]-x(2)-[DE] | неспецифична | 11 |
| PS00007 | TYR_PHOSPHO_SITE | Tyrosine kinase phosphorylation site | паттерн | [RK]-x(2)-[DE]-x(3)- Yor[RK]-x(3)-[DE]-x(2)-Y | неспецифична | 2 |
| PS00001 | ASN_GLYCOSYLATION | N-glycosylation site | паттерн | N-{P}-[ST]-{P} | неспецифична | 1 |