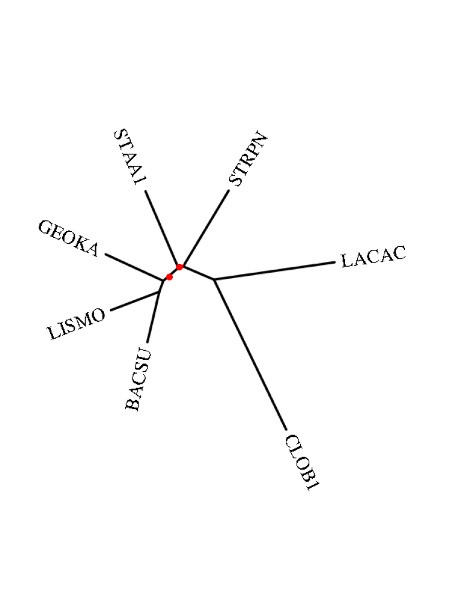
- Отобранные бактерии
| Название | Мнемоника |
| Bacillus subtilis | BACSU |
| Clostridium botulinum | CLOB1 |
| Geobacillus kaustophilus | GEOKA |
| Lactobacillus acidophilus | LACAC |
| Listeria monocytogenes | LISMO |
| Staphylococcus aureus | STAA1 |
| Streptococcus pneumoniae | STRPN |
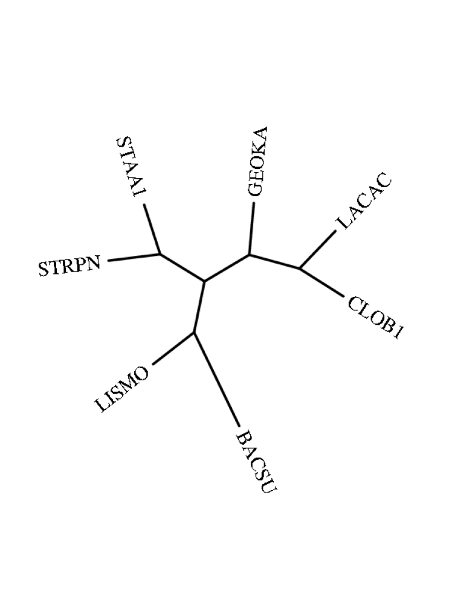
- Скобочная формула дерева
(CLOB1,((LACAC,STRPN),(STAA1,(LISMO,(BACSU,GEOKA)))))
- Изображение дерева
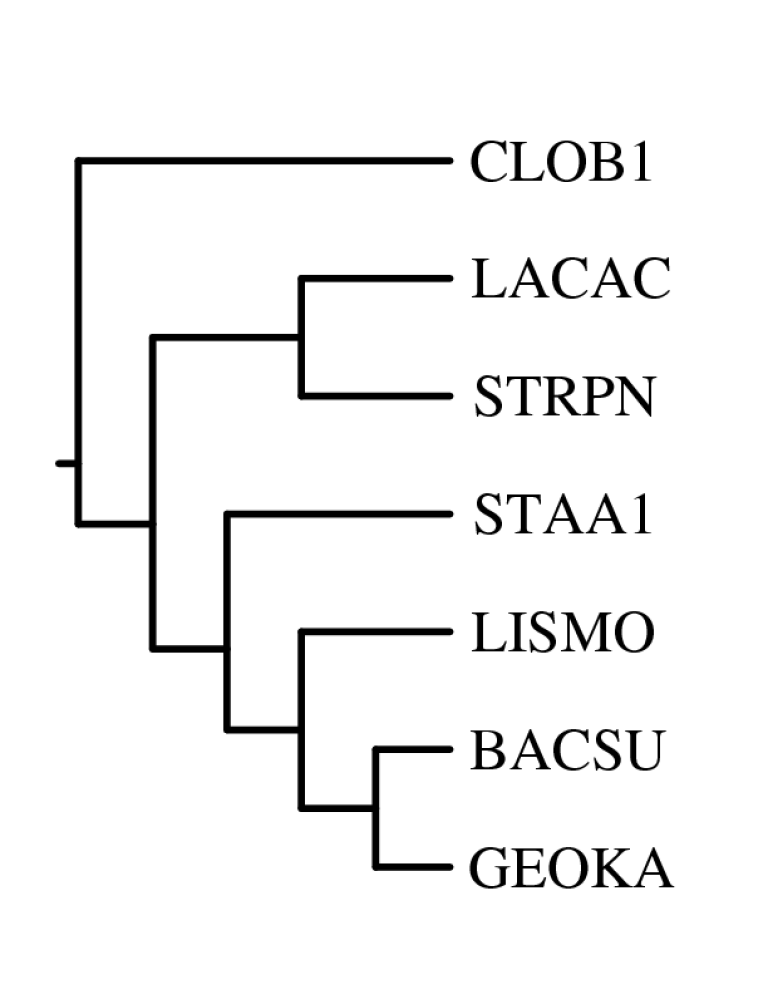
- Ветви дерева
Дерево содержит 4 нетривиальные ветви:
{LACAC, STRPN} против {CLOB1, STAA1, LISMO, BACSU, GEOKA}
{CLOB1, LACAC, STRPN} против {STAA1, LISMO, BACSU, GEOKA}
{CLOB1, LACAC, STRPN, STAA1} против {LISMO, BACSU, GEOKA}
{CLOB1, LACAC, STRPN, STAA1, LISMO} против {BACSU, GEOKA}
- Систематическое положение отобранных бактерий
| Название | Систематическое положение |
| Bacillus subtilis | Firmicutes; Bacilli; Bacillales; Bacillaceae; Bacillus |
| Clostridium botulinum | Firmicutes; Clostridia; Clostridiales; Clostridiaceae; Clostridium |
| Geobacillus kaustophilus | Firmicutes; Bacilli; Bacillales; Bacillaceae; Geobacillus |
| Lactobacillus acidophilus | Firmicutes; Bacilli; Lactobacillales; Lactobacillaceae; Lactobacillus |
| Listeria monocytogenes | Firmicutes; Bacilli; Bacillales; Listeriaceae; Listeria |
| Staphylococcus aureus | Firmicutes; Bacilli; Bacillales; Staphylococcaceae; Staphylococcus |
| Streptococcus pneumoniae | Firmicutes; Bacilli; Lactobacillales; Streptococcaceae; Streptococcus |
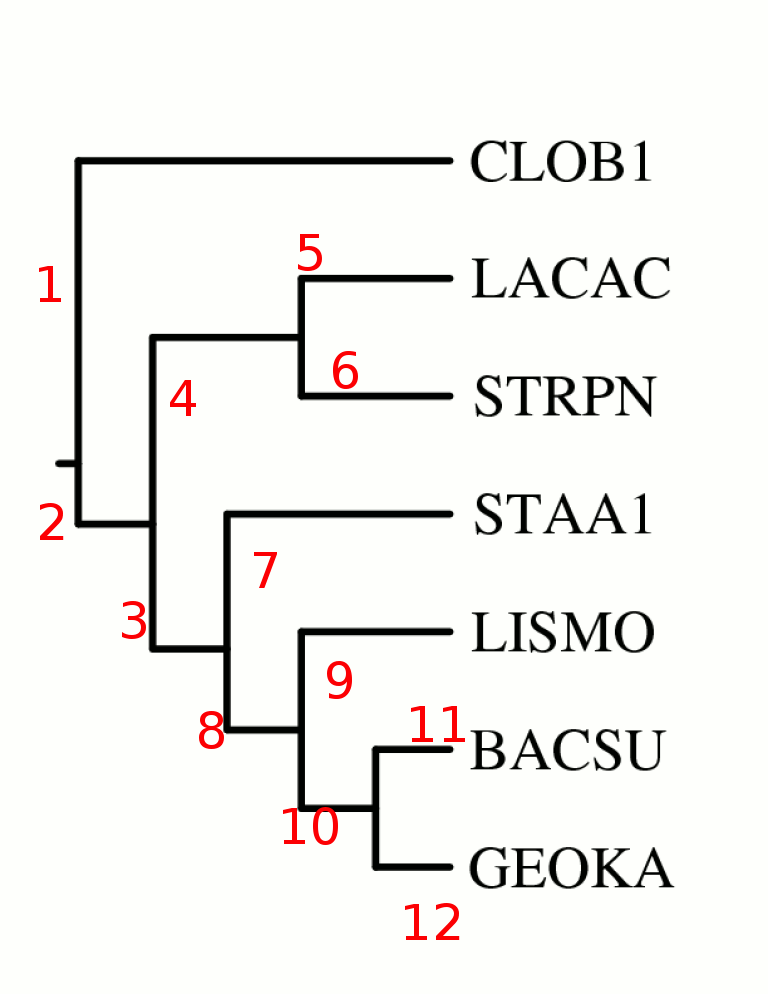
| Номер ветви | Выделяет таксон |
| 1 | Clostridia |
| 2 | Bacilli |
| 3 | Bacillales |
| 4 | Lactobacillales |
| 5 | Lactobacillaceae |
| 6 | Streptococcaceae |
| 7 | Staphylococcaceae |
| 8 | - |
| 9 | Listeriaceae |
| 10 | Bacillaceae |
| 11 | Bacillus |
| 12 | Geobacillus |
- Проведение реконструкции дерева программой fprotpars
Для проведения реконструкции дерева я выбрала фактор элонгации трансляции G (EFG)
Было получено 2 дерева
+--------------EFG_LISMO
!
+--4 +--EFG_STRPN
! ! +--------6
! ! ! +--EFG_STAA1
! +--5
! ! +-----EFG_GEOKA
1 +-----2
! ! +--EFG_LACAC
! +--3
! +--EFG_CLOB1
!
+-----------------EFG_BACSU
((EFG_LISMO,((EFG_STRPN,EFG_STAA1),(EFG_GEOKA,(EFG_LACAC,EFG_CLOB1)))), EFG_BACSU)
+--------------EFG_LISMO
!
+--4 +-----------EFG_GEOKA
! ! !
! +--2 +--EFG_STRPN
! ! +-----6
! ! ! +--EFG_STAA1
1 +--5
! ! +--EFG_LACAC
! +-----3
! +--EFG_CLOB1
!
+-----------------EFG_BACSU
((EFG_LISMO,(EFG_GEOKA,((EFG_STRPN,EFG_STAA1),(EFG_LACAC,EFG_CLOB1)))), EFG_BACSU)
- Определение эволюционных расстояний между прследовательностями программой fprotdist
Матрица расстояний
EFG_BACSU 0.000000 0.385376 0.182882 0.367870 0.158052 0.251550 0.275347
EFG_CLOB1 0.385376 0.000000 0.383249 0.495355 0.420966 0.461723 0.491656
EFG_GEOKA 0.182882 0.383249 0.000000 0.364273 0.203891 0.253015 0.261437
EFG_LACAC 0.367870 0.495355 0.364273 0.000000 0.379123 0.385480 0.360447
EFG_LISMO 0.158052 0.420966 0.203891 0.379123 0.000000 0.257595 0.268006
EFG_STAA1 0.251550 0.461723 0.253015 0.385480 0.257595 0.000000 0.273606
EFG_STRPN 0.275347 0.491656 0.261437 0.360447 0.268006 0.273606 0.000000
Насколько расстояния отклоняются от ультраметричности (на одном-двух примерах)? Насколько расстояния отклоняются от аддитивности (на каком-нибудь одном примере)?
Отклонения расстояний от ультраметричности:
В тройке CLOB1-GEOKA-LACAC отклонение небольшое (0.02):
| Пара | Расстояние |
| CLOB1-GEOKA | 0.383249 |
| GEOKA-LACAC | 0.364273 |
| CLOB1-LACAC | 0.495355 |
В тройке BACSU-CLOB1-STRPN отлонения значительны (0.11):
| Пара | Расстояние |
| CLOB1-BACSU | 0.385376 |
| STRPN-BACSU | 0.275347 |
| CLOB1-STRPN | 0.491656 |
Отклонение от свойства аддитивности, наблюдаемое в четверке: LACAC, BACSU, LISMO, CLOB1 составляет примерно 0.03
- Проведение реконструкции дерева программой fneighbor
По алгоритму Neighbor-Joining
+----EFG_LISMO ! ! +-----EFG_GEOKA ! ! 1-4 +---------------EFG_CLOB1 ! ! +-2 ! ! ! +------------EFG_LACAC ! +-5 ! ! +-------EFG_STAA1 ! +-3 ! +-------EFG_STRPN ! +---EFG_BACSU
Скобочная формула:
(EFG_LISMO:0.08568,(EFG_GEOKA:0.09158,((EFG_CLOB1:0.27969, EFG_LACAC:0.21566):0.02517,(EFG_STAA1:0.13214,EFG_STRPN:0.14147):0.01517):0.01150):0.02279,EFG_BACSU:0.07237);
По алгоритму UPGMA
+----EFG_BACSU
+-1
+-2 +----EFG_LISMO
! !
+-3 +-----EFG_GEOKA
! !
+--4 +------EFG_STAA1
! !
+-5 +-------EFG_STRPN
! !
--6 +----------EFG_LACAC
!
+------------EFG_CLOB1
Скобочная формула:
((((((EFG_BACSU:0.07903,EFG_LISMO:0.07903):0.01767,EFG_GEOKA:0.09669):0.03033, EFG_STAA1:0.12703):0.00777,EFG_STRPN:0.13480):0.05092,EFG_LACAC:0.18572):0.03414, EFG_CLOB1:0.21986);
Деревья, полученные по разным алгоритмам, сильно отличаются от исходного. Дерево, полученное программой fprotpars (первое) имеет с реальным деревом 0 общих ветвей, по алгоритму N-J - 1 общую ветвь, по алгоритму UPGMA 2 общие ветви. На иллюстрациях общие ветви помечены красными кружками.
Исходное дерево
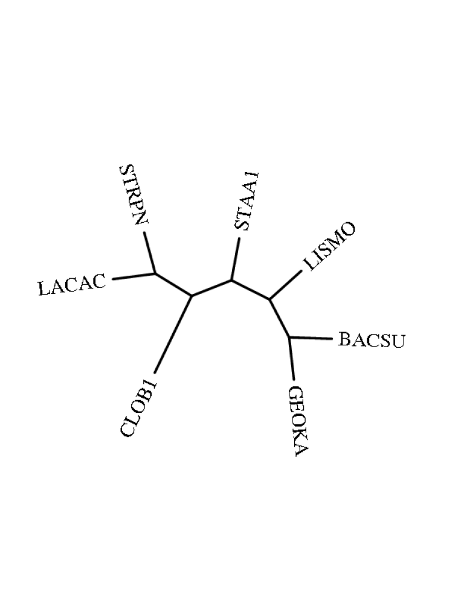
Дерево, полученное программой fprotpars
Дерево, полученное по алгоритму Neighbor-Joining
Дерево, полученное по алгоритму UPGMA