- Построение дерева по нуклеотидным последовательностям
Получение последовательностей 16S RNA.
Мнемоника EMBL acsession Координаты BACSU AL009126 9810..11364 CLOB1 CP000726 9282..10783 GEOKA BA000043 10421..11973 LACAC CP000033 59255..60826 LISMO CP001602 263875..265429 STAA1 AP009324 531922..533476 STRPN CP000918 16782..18239
Дерево,полученное программой fdnapars по алгоритму максимальной парсимонии.
+-bacsu
+--5
| | +--staa1
@--3 +--4
| | +--lismo
+--2 |
| | +---geoka
| |
| +-------strpn
|
1-----lacac
|
+------clob1
Скобочная формула:
((((bacsu:0.02533,(staa1:0.04673,lismo:0.04964):0.01982):0.02704, geoka:0.05800):0.03739,strpn:0.12427):0.03581,lacac:0.09246,clob1:0.12452);
Полученное дерево имеет с правильным 1 общюю ветвь:
{strpn, lacac, clob1} против {bacsu, geoka, lismo, staa1}
(На схеме показана значком "@")
Полученный результат совпадает по точности с реконструкцией по белковому фактору элонгации программой protpars (два лучших дерева имели 0 и 1 совпадающих ветвей).
Дерево,полученное программой ffitch по алгоритму Фитча-Марголиаша.
+-bacsu
@-1
+-5 +---geoka
! !
@-4 +--staa1
! !
+-3 +--lismo
! !
! +-----strpn
!
2-----lacac
!
+------clob1
Скобочная формула:
(((((bacsu:0.03145,geoka:0.06417):0.00613,staa1:0.04964):0.00823, lismo:0.04413):0.01275,strpn:0.09150):0.00458,lacac:0.09261,clob1:0.11294);
Полученное дерево имеет с правильным 2 общие ветви:
{strpn, lacac, clob1} против {bacsu, geoka, lismo, staa1}
{strpn, lacac, clob1, lismo, staa1} против {bacsu, geoka}
(На схеме показаны значком "@")
Полученный результат совпадает по точности с реконструкцией по белковому фактору элонгации программой fneighbor по алгоритму UPGMA (лучшee деревo имелo 2 совпадающих ветви).
Исходное дерево:
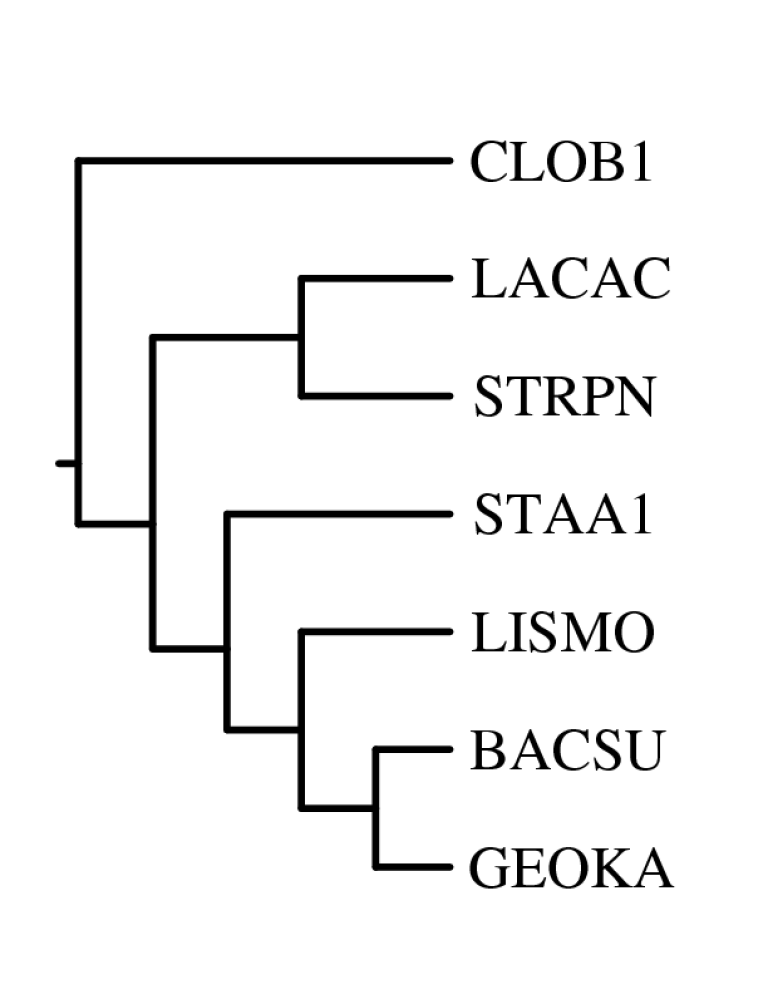
- Построение и анализ дерева, содержащего паралоги
Для нахождения гомологов белка CLPX_BACSU была использована программа blastp на сервере kodomo и файл с белками бактерий proteo.fasta
Было найдено 25 белков с e-value < 0.1, 21 из них включены в итоговое дерево.
Дерево построено программой fneighbor по алгоритму UPGMA.
+------CLPX_STRPN
+----------9
! ! +------CLPX_CLOB1
! +-7
! ! +-----CLPX_STAA1
! +-6
! ! +---CLPX_LISMO
+---------------------17 +--2
! ! ! +-CLPX_BACSU
! ! +-1
! ! +-CLPX_GEOKA
! !
! ! +---------HSLU_LACAC
! +------13
! ! +-------HSLU_STAA1
! +-11
! ! +------HSLU_LISMO
+-18 +-8
! ! ! +---CLPY_BACSU
! ! +--3
! ! +---HSLU_GEOKA
! !
! ! +-------RUVB_STRPN
! ! +-10
! ! ! ! +-----RUVB_LISMO
+-------19 ! ! +-5
! ! +------------------------------12 ! +----RUVB_BACSU
! ! ! +-4
! ! ! +----RUVB_GEOKA
! ! !
! ! +--------RUVB_CLOB1
! !
-20 ! +---------------CLPB_LISMO
! +--------------------------16
! ! +---------CLPE_BACSU
! +----14
! +---------CLPE_STRPN
!
! +------------FTSH_BACSU
+-------------------------------------15
+------------FTSH_STRPN
Белки CLPX, HSLU, RUBV, CLPE, FTSH из разных организмов являются ортологами. Наример: FTSH_BACSU и FTSH_STRPN, CLPX_BACSU и CLPX_STAA1, RUVB_LISMO и RUVB_GEOKA.
Представленные на дереве белки из одного организма являются паралогами. Например: CLPX_BACSU и CLPY_BACSU, RUVB_STRPN и CLPE_STRPN, CLPX_GEOKA и RUVB_GEOKA.