- Определить, в каких мметаболических путях участвует фермент ECHB_HUMAN
EC: 2.3.1.16
Идентификатор KEGG - hsa:3032
hsa00062, fatty acid elongation, элонгация жирных кислот
hsa00071, fatty acid metabolism, метаболизм жирных кислот
hsa00280, valine, leucine and isoleucine degradation, деградация валина, лейцина и изолейцина
hsa01100, metabolic pathways, метаболические пути
картинка
- Найти в KEGG структурные формулы заданных соединений: L-аспартата и L-лизина.
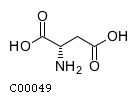
L-аспартат, L-Aspartate, C00049
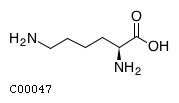
L-лизин, L-Lysine, C00047
- Найти в KEGG метаболический путь, ведущий от L-аспартата к L-лизину.
ko00300
Карта: биосинтез лизина
Путь: L-аспартат → L-лизин
картинка
- Сравнить метаболические пути у разных организмов.
| Организм | Цепочка реакций возможна (да/нет/неизвестно) |
Обоснование |
| Bacillus subtilis | нет | нет 1 фермента, катализирующий 5-ую стадию (EC 1.4.1.16) но есть чуть более длинный обходной путь |
| Archaeoglobus fulgidus | да | есть все ферменты |
| Arabidopsis thaliana | да | есть все ферменты |
| Homo sapiens | нет | нет ни одного фермента данного пути |
картинка
- Мое мнение о KEGG.
Вообще здорово. Только переход из reference pathway к конкретному организму не очень удобно сделан, и жалко, что нельзя раскрасить соединения тыкая по карте. Вряд ли это сложно реализовать технически...