- Параметры:
Силовое поле используемое при построении топологии топологии - amber99sb
Заряд системы - -10
Причины этого значения - фосфаты ДНК
Размер и форму ячейки - кубическая, отступ 1,5 нм
Минимизация энергии:
Алогритм минимизации энергии - steepest descent, наискорейшего спуска
Алгоритм расчёта электростатики и Ван-дер-Ваальсовых взаимодействий - cut-off
Модель, которой описывался растворитель - TIP3P
Утряска растворителя:
Для биополимеров, укажите параметр который обуславливает неподвижность биополимера - constraints = all-bonds
Число шагов - 10000
Длина шага - 0.001 пс
Алгоритм расчёта электростатики и Ван-дер-Ваальсовых взаимодействий - PME, Particle Mesh Ewald и Cut-off, соотв
Алгоритмы термостата и баростата - V-rescale и нет соответственно.
Основной расчёт МД:
Время моделирования, количество процессоров, эффективность маштабирования - 10 нс, 16,
Если моделирование окончилось с ошибкой, указать ошибку и вероятную причину.
Длину траектории -
Число шагов - 5000000
Длина шага - 0.002 пс
Алгоритм интегратора - md
Алгоритм расчёта электростатики и Ван-дер-Ваальсовых взаимодействий - PME, Particle Mesh Ewald и Cut-off, соотв.
Алгоритмы термостата и баростата - V-rescale и Berendsen, соотв.
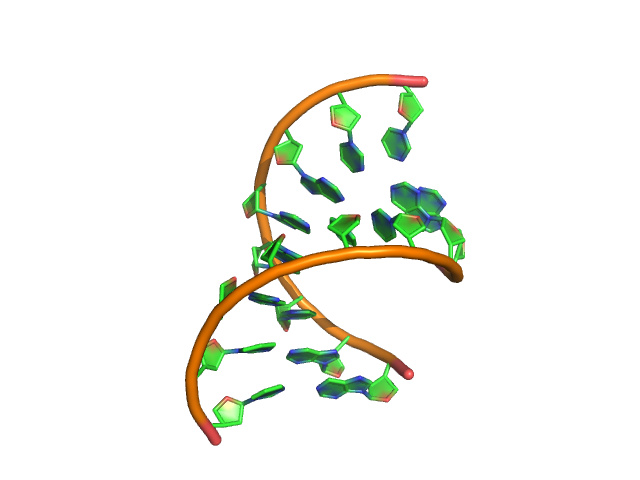
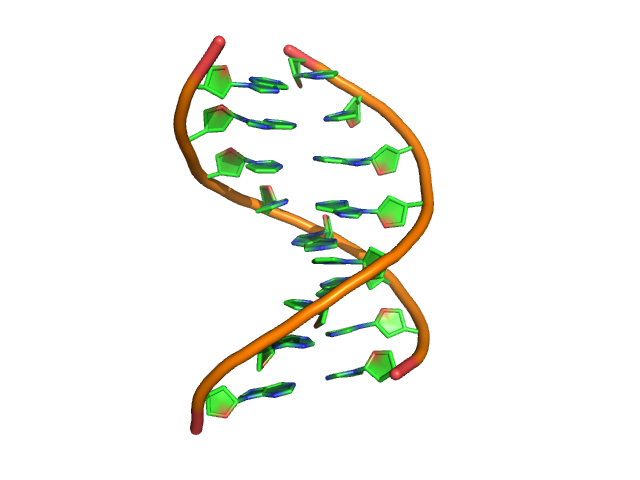
- А и В формы ДНК.
Переход происходит примерно на 90м шаге.
А-форма
B-форма
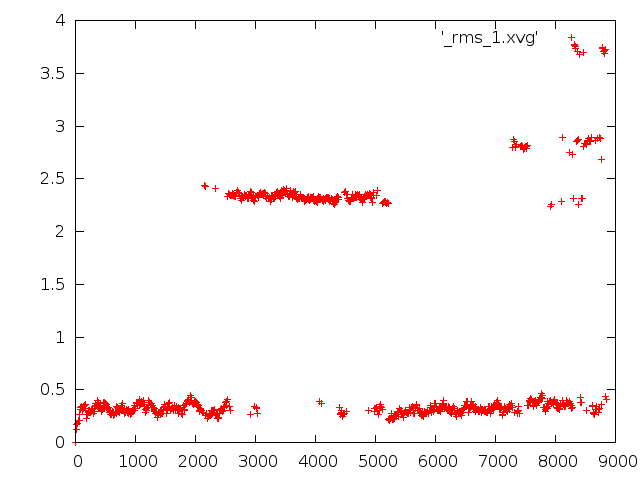
- Определение средне-квадратичного отколнения в ходе моделирования.
RMSD от стартовой структуры
RMSD от структуры, отстающей на 400 фреймов.
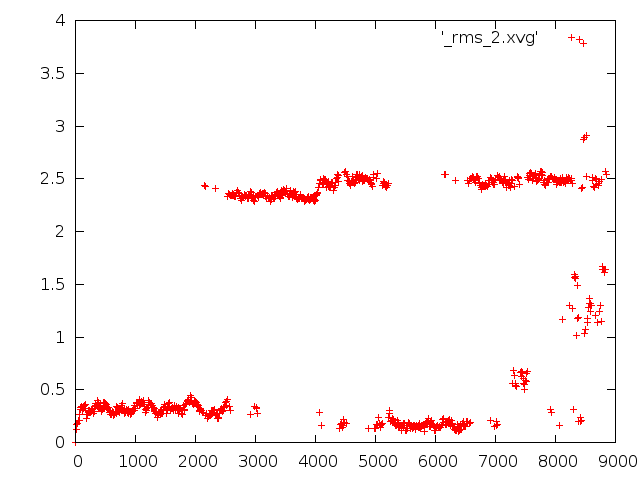
В случае отклонения от стартовой структуры, rmsd сначала немгого увеличивается, потом выходит на плато. Резкие скачки rmsd в области 250-500 соответствуют плавлению дуплекса. В случае rmsd от структуры 400 кадров назад плавление опять все портит.
- Определение изменения гидрофобной и гидрофильной поверхности в ходе конформационного перехода.
Изменение гидрофобной поверхности ДНК
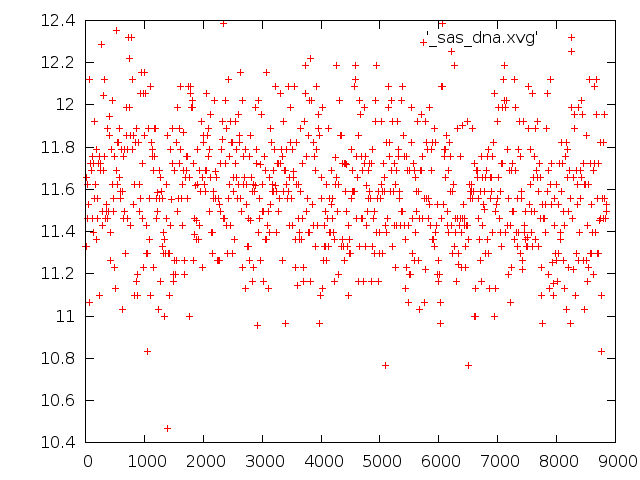
Какого-либо явного тренда нет. В области 250-500 и в районе 700 вроде бы небольшие горбы, опать-таки связанные с плавлением.
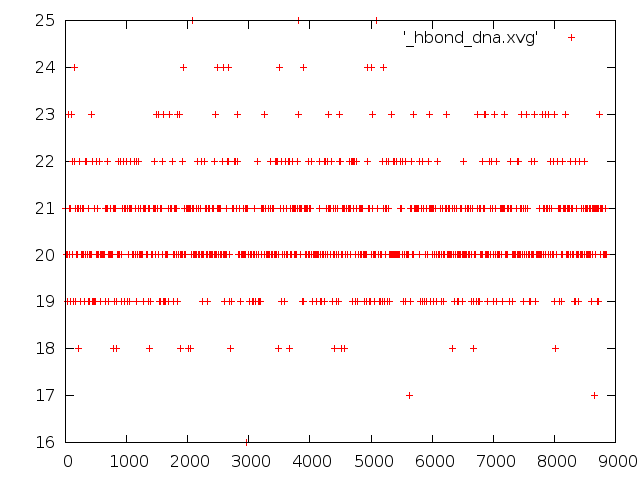
- Pасчёт количества образуемых водородных связей.
Количество водородных связей в ДНК.
Количество водородных связей между ДНК и растворителем.
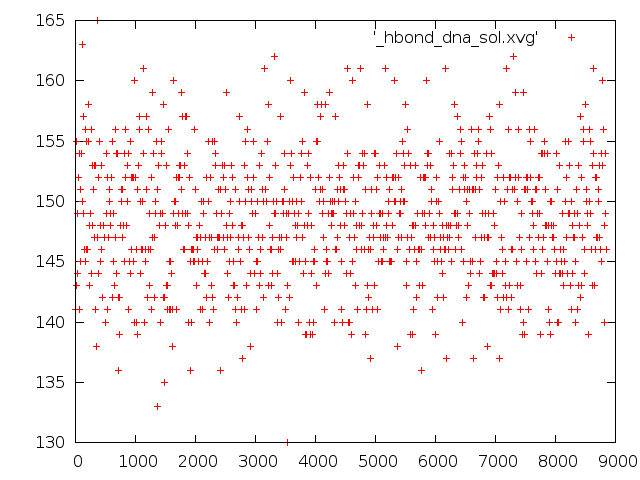
Судя по первому графику, в дуплексе 20-21 водородная связь. (А вообще вроде 14 должно быть. Странно, почему они не уменьшаются при плавлении дуплекса? ) Количество водородных связей с растворителем примерно постоянно.