Практикум 15. Сборка de novo
1. Подготовка ридов
Были взяты риды из проекта с id: SRR4240378.
Для использования программы trimmomatic ILLUMINACLIP адаптеры из директориии /mnt/scratch/NGS/adapters были собраны в один fasta файл. Для удаления адаптеров была использована команда:
java -jar /usr/share/java/trimmomatic.jar SE -phred33 SRR4240378.fastq.gz SRR4240378_cleaned.fastq.gz ILLUMINACLIP:adapters.fasta:2:7:7Для триммирования нуклеотидов с качеством прочтения ниже 20, и удаления ридов короче 32 нуклеотидов была использована команда:
java -jar /usr/share/java/trimmomatic.jar SE -phred33 SRR4240378_cleaned.fastq.gz SRR4240378_filt.fastq.gz TRAILING:20 MINLEN:32После этих шагов из 4420587 ридов осталось 4154738, размер файла уменьшился с 91 до 84 Мб.
2. Сборка
K-меры были получены командой:
velveth velveth_1 31 -short -fastq SRR4240378_filt.fastq.gzКонтиги были собраны командой:
velvetg velveth_1Ниже представллены некоторые характеристики полученной сборки:
N50: 8084
3 наиболее длинных контига:
- ID: 8, длина: 36746, покрытие: 20.02
- ID: 56, длина: 19371, покрытие: 20.55
- ID: 18, длина: 18954, покрытие: 20.79
3. Анализ
Контиги были выравнены на геном бактерии Buchnera aphidicola (AC: CP009253) с помощью NCBI megablast с использованием опции: Align two or more sequences.
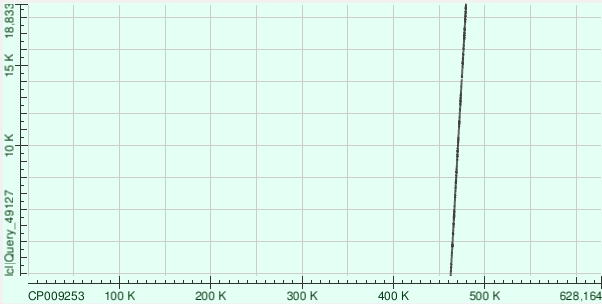
Контиг 8
Выравнялся кусочно в 7 местах на хромосому, характеристики ниже представлены для самого длинного выравнивания:
- Длина: 8614
- Координаты: 500370-508773
- E-value: 0.0
- Замены: 1756
- Гэпы: 345
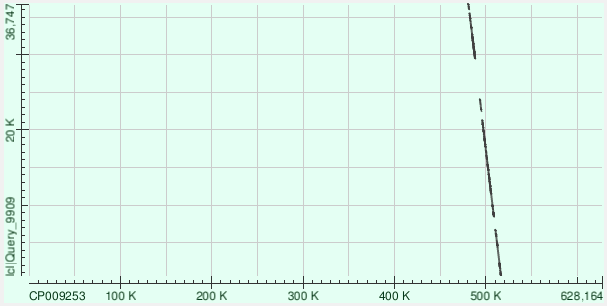
Контиг 56
Выравнялся кусочно в 2 местах на хромосому, ниже представлены характеристики для самого длинного выравнивания и карта локального сходства:
- Длина: 9826
- Координаты: 573092-582689
- E-value: 0.0
- Замены: 2148
- Гэпы: 463
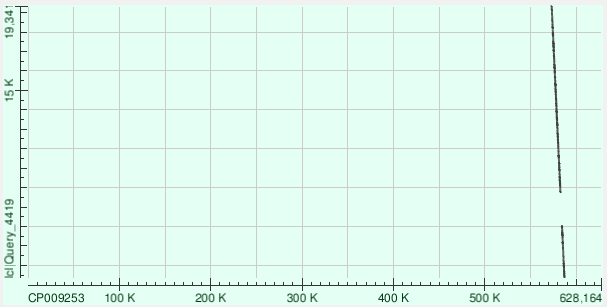
Контиг 18
Выравнялся кусочно в 3 местах на хромосому, ниже представлены характеристики для самого длинного выравнивания и карта локального сходства:
- Длина: 7387
- Координаты: 467412-474667
- E-value: 0.0
- Замены: 1491
- Гэпы: 206