Практикум 8. Нуклеотидный BLAST.
Задание 1. Определение функции и таксономии нуклеотидной последовательности.
Вариантом BLAST был выбран blastn, так как он используется для поиска сколько-нибудь похожих последовательностей, а про полученную в результате анализа хроматограммы последовательность нет никакой дополнительной информации.
Параметры поиска:
- Program: blastn
- Word size: 11
- Expect value: 0.05
- Hitlist size: 100
- Match/Mismatch scores: 2,-3
- Gapcosts: 5,2
- Low Complexity Filter: Yes
- Filter string: L;m;
- Genetic Code: 1
Предположительно, данная последовательность принадлежит многощетинковому червю (Polychaeta) Polycirrus medusa, так как ему принадлежат 4 находки с самым высоким процентом идентичности. С высокой долей вероятности организм, из которого была получена анализируемая последовательность, принадлежит семейству: Terebellidae, на них приходится 124 находки. Данный ген скорее всего кодирует первую субъединицу цитохром-оксидазы, так как 92 найденные последовательности являются генами этого белка.
Задание 2. Поиск генов белков в неаннотированной нуклеотидной последовательности.
Для проведения BLAST была выбрана программа blastx, так как она проводит трансляцию нуклеотидной последовательности в белковую, что, предположительно, больше подходит для поиска белков, имеющих схожую функцию, по которой можно будет судить о функции гена в контиге.
Ссылка на контиг (из генома mCanLor1.2 Canis lupus): Контиг 27
Параметры поиска:
- Program: blastx
- Word size: 6
- Expect value: 0.01
- Hitlist size: 100
- Gapcosts: 11,1
- Matrix: BLOSUM62
- Low Complexity Filter: Yes
- Filter string: L;
- Genetic Code: 1
- Window Size: 40
- Threshold: 21
- Composition-based stats: 2
BLAST проводился с исключением рода Canis по базе данных swissprot (содержит проверенные последовательности белков, поиск по базе nr не проходил, вероятно, из-за слишком большого количества последовательностей в ней).
В данном контиге примерно в координатах 18780 - 19708 находится ген, который, с высокой долей вероятности кодирует фактор дифференциации роста, так как большая часть найденных последовательностей выравнивается на этот участок и относятся к генам факторов дифференциации. Также можно предположить, что это ген фактора дифференцировки роста 6 (GDF-6), так как 5 лучших находок являются генами (GDF-6).
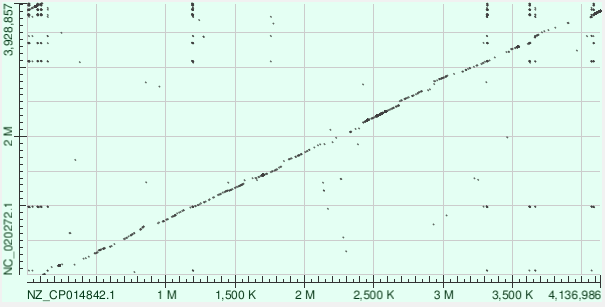
Задание 3. Интепретация карты локального сходства гомологичных хромосом двух бактерий.
Были выбраны: Bacillus licheniformis (NZ_CP014842.1) и Bacillus amyloliquefaciens (NC_020272.1). На карте локального сходства видно, что геномы бактерий в основном схожи, но по уровню шума можно судить, что они тем не менее отличаются. Из крупных геномных перестроек видно несколько делеций/инсерций, например на уровне 2,7 мегабаз.