Практикум 4. Паралоги, визуализация.
Результаты blastp
| Белок | Score (bits) | E-value |
|---|---|---|
| CLPX_STRAW | 535 | 0.0 |
| CLPX_RHOJR | 534 | 0.0 |
| CLPX_MYCVP | 523 | 0.0 |
| CLPX_ARTS2 | 516 | 0.0 |
| CLPX_LEIXX | 509 | 2e-180 |
| CLPX_CLAMS | 491 | 3e-173 |
| CLPX_BIFLO | 432 | 2e-149 |
| A0K1M3_ARTS2 | 54.3 | 2e-149 |
| Q8G871_BIFLO | 51.2 | 1e-06 |
| Q0S6Y7_RHOJR | 47.8 | 1e-05 |
| Q0S8C7_RHOJR | 47.0 | 3e-05 |
| RUVB_BIFLO | 42.7 | 4e-04 |
| Q6ACQ0_LEIXX | 43.1 | 4e-04 |
| Q8G3S2_BIFLO | 43.1 | 4e-04 |
| A1TG29_MYCVP | 43.1 | 5e-04 |
| RUVB_ARTS2 | 42.4 | 6e-04 |
| Q82QV8_STRAW | 41.6 | 7e-04 |
Реконструкция и визуализация
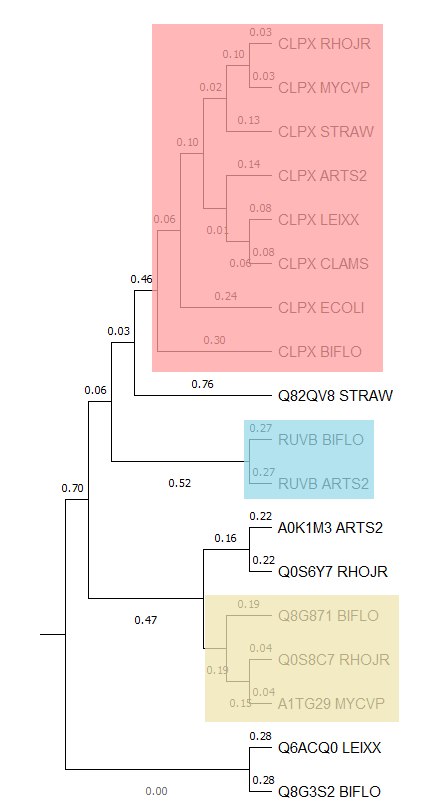
Дерево было реконструировано в программе MEGA с использованием метода UPGMA. Дереве Newick-формате приведено по ссылке.
Примеры пар ортологов: CLPX_RHOJR и CLPX_MYCVP, CLPX_LEIXX и CLPX_CLAMS, RUVB_BIFLO и RUVB_ARTS2.
Примеры пар паралогов: CLPX_BIFLO и RUVB_BIFLO, CLPX_MYCVP и A1TG29_MYCVP, RUVB_ARTS2 и A0K1M3_ARTS2.
Дерево с выделенными ортологичными группами.
Дерево с объединенными ортологичныим группами.
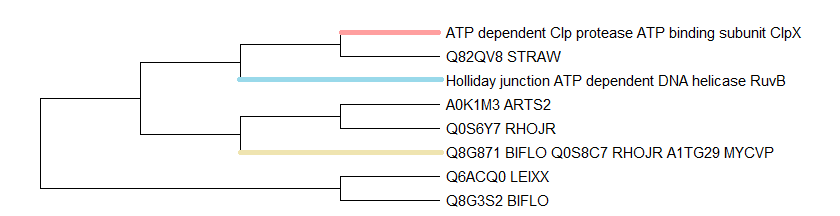
В группе, выделенной красным, находится белок: АТФ-связывающая субъединица АТФ-зависимой протеазы Clp, обнаружен во всех бакткриях. В группе, выделенной синим, находится белок: хеликаза структуры Холидея RuvB, обнаружена в бактериях: Bifidobacterium longum, Arthrobacter sp. В группе, выделенной желтым, находятся автоматически аннотированные белки из базы данных TrEMBL.
Отличия реконструированной филогении проще всего наблюдать на примере группы, выделенной красным, так как в нее вошли все бактерии. По составу ветвей дерево совпадает с эталонным, однако в эталонном дереве укоренение в ветвь, отделяющую MYCVP и RHOJR от остальных, а в дереве гомологов укоренение - в ветвь, отделяющую BIFLO.