В этом практикуме была найдена белковая последовательность и ген δ-субъединицы АТФ-синтазы из Ischnura elegans, которые были использованы для поиска гомологов из неродственных организмов с помощью разных алгоритмов Blast.
Идентификатор белка: XP_046391852.1
Последовательность белкаИдентификатор нуклеотидной записи, содержащей ген белка: NC_060251
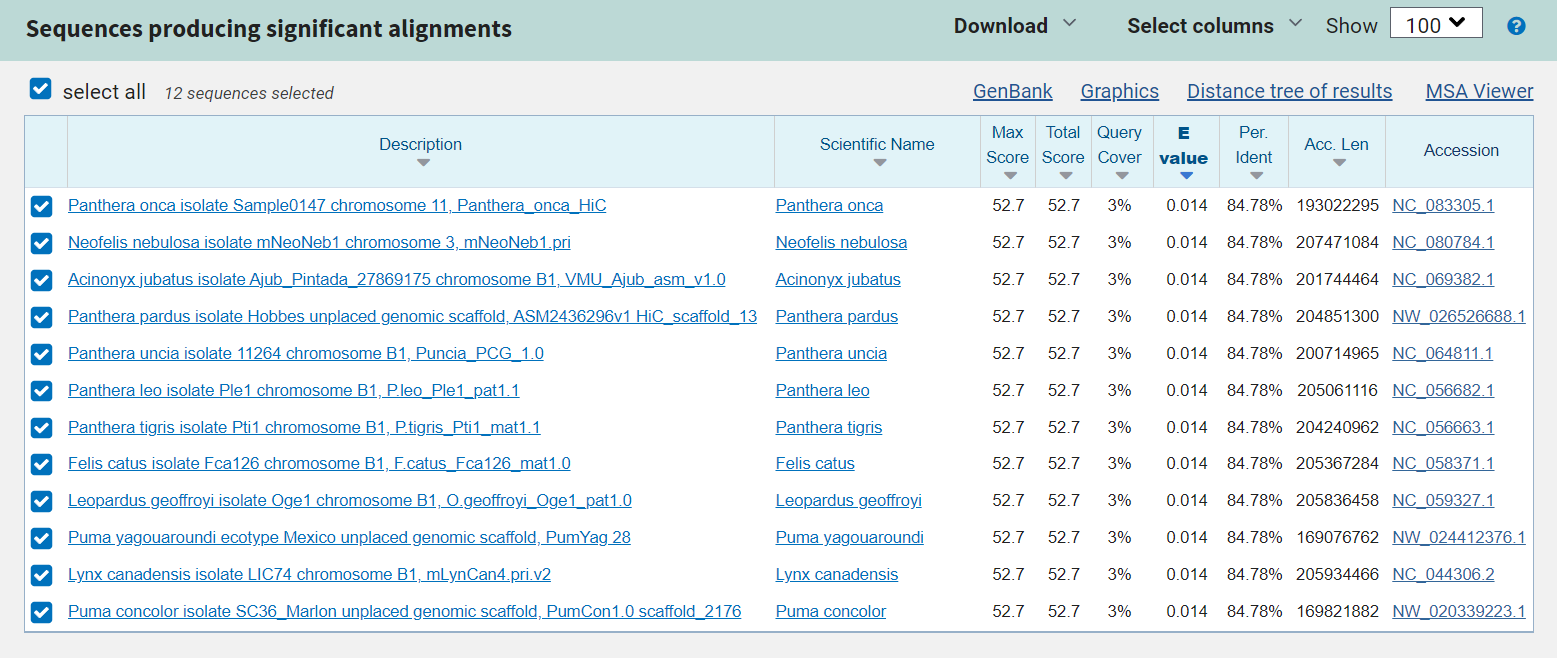
Окрестность генаПоскольку стрекозы являются первичноротыми, то в качестве удаленного семейства я решил выбрать Кошачьих (Felidae). Поиск производился по базе refseq_genomes, содержащей 15 сборок выбранного семейства.
1. Я отдал предпочтение blasn, поскольку он позволяет находить последовательности с невысоким процентом совпадений. Поиск с длиной слов 11 и 7 нашел 12 гомологов в сборках.

2. Поскольку мне доступна аминокислотная последовательность белка, то разумно проводить поиск с помощью tblastn, чтобы исключить несовпадения из-за вырожденности генетического кода. Поиск с длиной 5 нашел гомологи во всех 15 сборках.
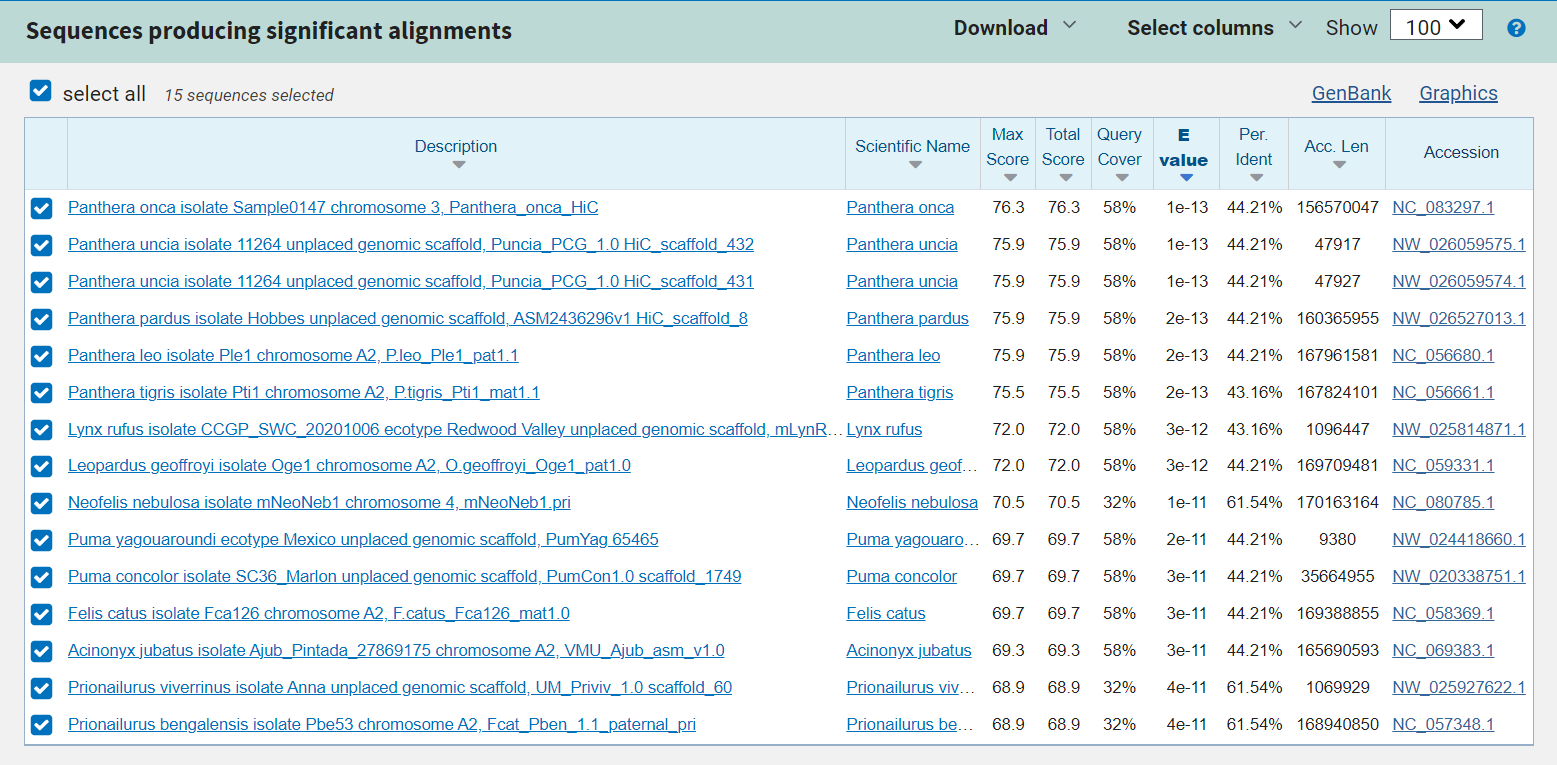
Число находок с помощью tblastn было ожидаемо. Удивило количество найденных гомологов с помощью blastn: поскольку генетический код вырожденный, а организмы далеки друг от друга, я ожидал что совпадений не будет.
В этом задании был проведен поиск гомологов генов рРНК из Escherichia coli в геноме Ischnura elegans.
Команда для индексации генома:
makeblastdb -in GCF_921293095.1_ioIscEleg1.1_genomic.fna -dbtype nucl
Поиск проводился с помощью алгоритма Blastn, так как было необходимо искать в некодирующих белки участках отдаленных организмов.
16S рРНК выполняет струтурную роль в малой субъединице, связывает мРНК и стабилизирует правильное спаривание кодона и антикодона. Основные функции 23S рРНК это структурная для большой субъединицы и пептидилтрансферазная. Использованные команды:
blastn -task blastn -query 16S.fasta -db GCF_921293095.1_ioIscEleg1.1_genomic.fna -out 16s_blastn.txt -evalue 0.05 -word_size 7
blastn -task blastn -query 23S.fasta -db GCF_921293095.1_ioIscEleg1.1_genomic.fna -out 23s_blastn.txt -evalue 0.05 -word_size 7
Для 16S рРНК было найдено 59 находок, а для 23S рРНК —68. С наилучшим счетом для обоих рРНК оказалась сборка 13 хромосомы( NC_060258.1), скэффолды в обоих случаях имеют одинаковый вес. Для поиска гомологов я отсортировал таблицу по координатам и отбросил находки с высоким e-value. Для 16S рРНК нашлось 20 гомологов (больше всего пришлось на скэффолд NW_025791680.1 — 4 гомолога), а для 23S рРНК — 38 (7 приходится на скэффолд NW_025791662.1). Высокое число гомологов на скэффолд объяснимо, т.к. гены, кодирующие предшественник рРНК, имеют много копий в геноме. Согласно NCBI хромосома 13 ( NC_060258.1) содержит гены 5.3s, 18s, 26s рРНК
Выдача для 16S рРНК:текст и таблица.Выдача для 23S рРНК:текст и таблица