В данном практикуме я прводил поиск последовательности Шайна — Дальгарно в геноме бактерии Pseudomonas aeruginosa. Последовательность Шайна — Дальгарно является сайтом связывания рибосом, расположеным на расстояннии около 10 нуклеотидов до старт-кодона. Консенсус — AGGAGG
Для поиска были получены последовательности длиной 100 нуклеотидов перед старт кодономи и последовательности такой же длины после старт-кодонов в качестве контроля. Использовалась программа bedtools:
bedtools flank -i GCF_003025345.2_ASM302534v2_genomic.gff -g GCF_003025345.2_ASM302534v2_genomic.fna.fai -l 100 -r 0 -s > promoters.bed
bedtools flank -i GCF_003025345.2_ASM302534v2_genomic.gff -g GCF_003025345.2_ASM302534v2_genomic.fna.fai -l 0 -r 100 -s > control.bed
Извлечение в fasta-файл:
bedtools getfasta -fi GCF_003025345.2_ASM302534v2_genomic.fna -bed promoters.bed -fo promoters.fasta
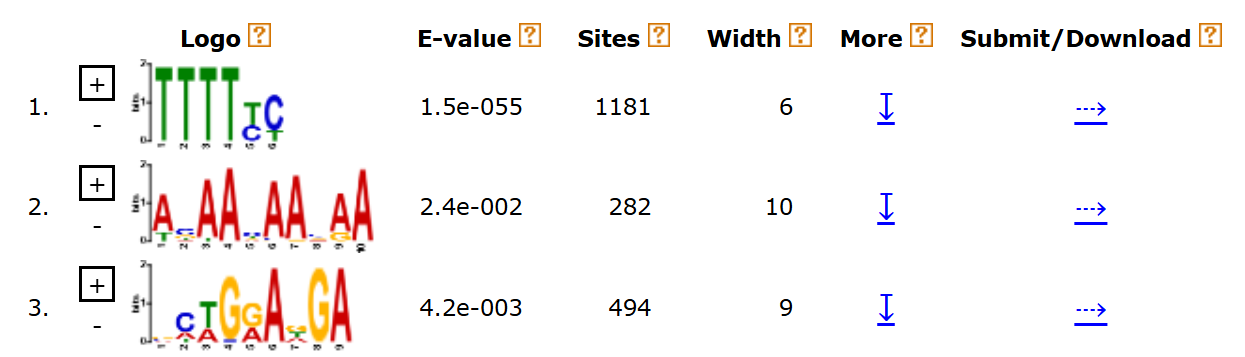
meme promoters.fasta -o meme_promoters -dna -nmotifs 3 -minw 6 -maxw 10 -revcompВыдача MEME

Было найдено 3 мотива длинной 6, 9 и 10 нуклеотидов.
Выдача для промоторной области и выдача для контроля
fimo meme_promoters/meme.txt promoters.fasta
fimo --o fimo_control meme_promoters/meme.txt control.fasta
В выдаче для промотора и контроля не нашлось мотива TTTTYC,хотя он имеет лучший p-value. Второй и третий мотив были найдены в промоторной области (всего 1060 находок) и в контроле (всего 787 находок). При этом нигде совсем не нашлось мотива TTTTYC, хотя он имеет лучший p-value (искал в tsv файлах). Как вывод: найденные мотивы не являются последовательностями Шайна — Дальгарно.