Задание 1
Проект Jalview доступен по ссылке
Для анализа был выбран домен PF00070 из Pfam. Этот домен является маленьким NADH-связывающим доменом внутри более крупного FAD-связывающего домена.
Число белков: 2,012(1 из swissprot ), с известной 3D структурой 790
Выравнивание seed: 87 последовательностей
Затем было скачано и проанализировано в программе Jalview выравнивание seed. Похожих последовательностей(с 90 % идентичностью) не было найдено
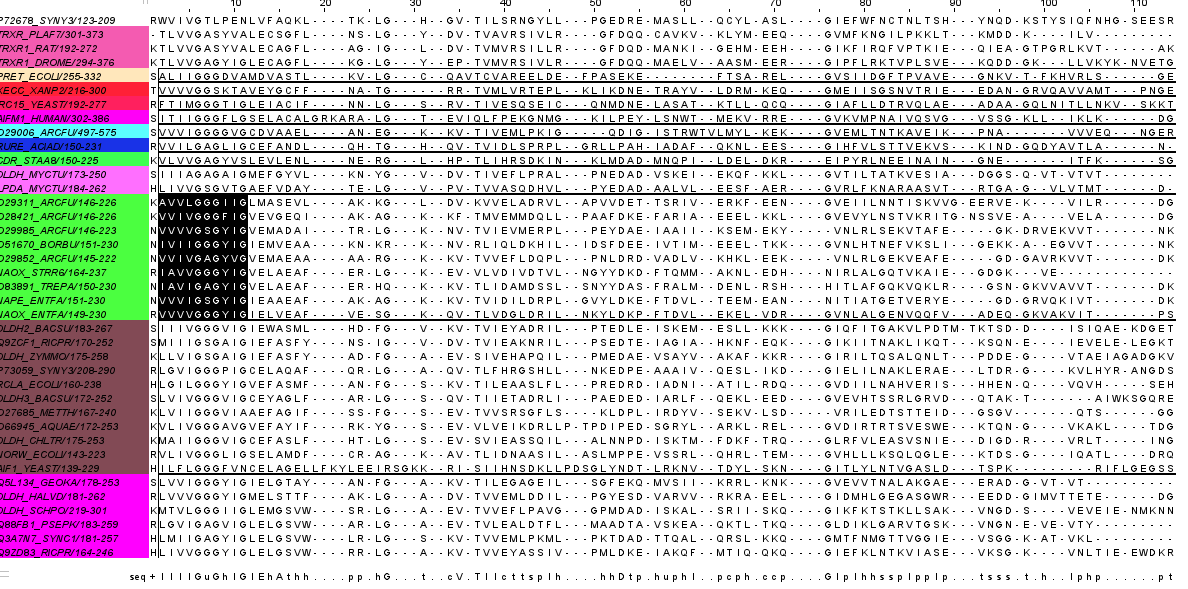
Был найден мотив с визуально высоким ИС(колонки 4-13)
Паттерн Jalview этого мотива: [VI]{2}G[GAS][GSD].{4}[ED], кроме того этот мотив встретился в других частях последовтаельностей.
64 последовательности имеют этот мотив
В формате Prosite этот мотив: [VI](2)-G-[GAS]-[GSD]-x(4)-[ED]
Затем был проведен поиск по этому паттерну в базе данных SwissProt в PROSITE. Было найдено 2987 находок в 2977 последовательностях.
Задание 2
Затем в Jalview было построено филогенетическое дерево с помощью NJ(BLOSUM62) - все построения доступны по ссылке
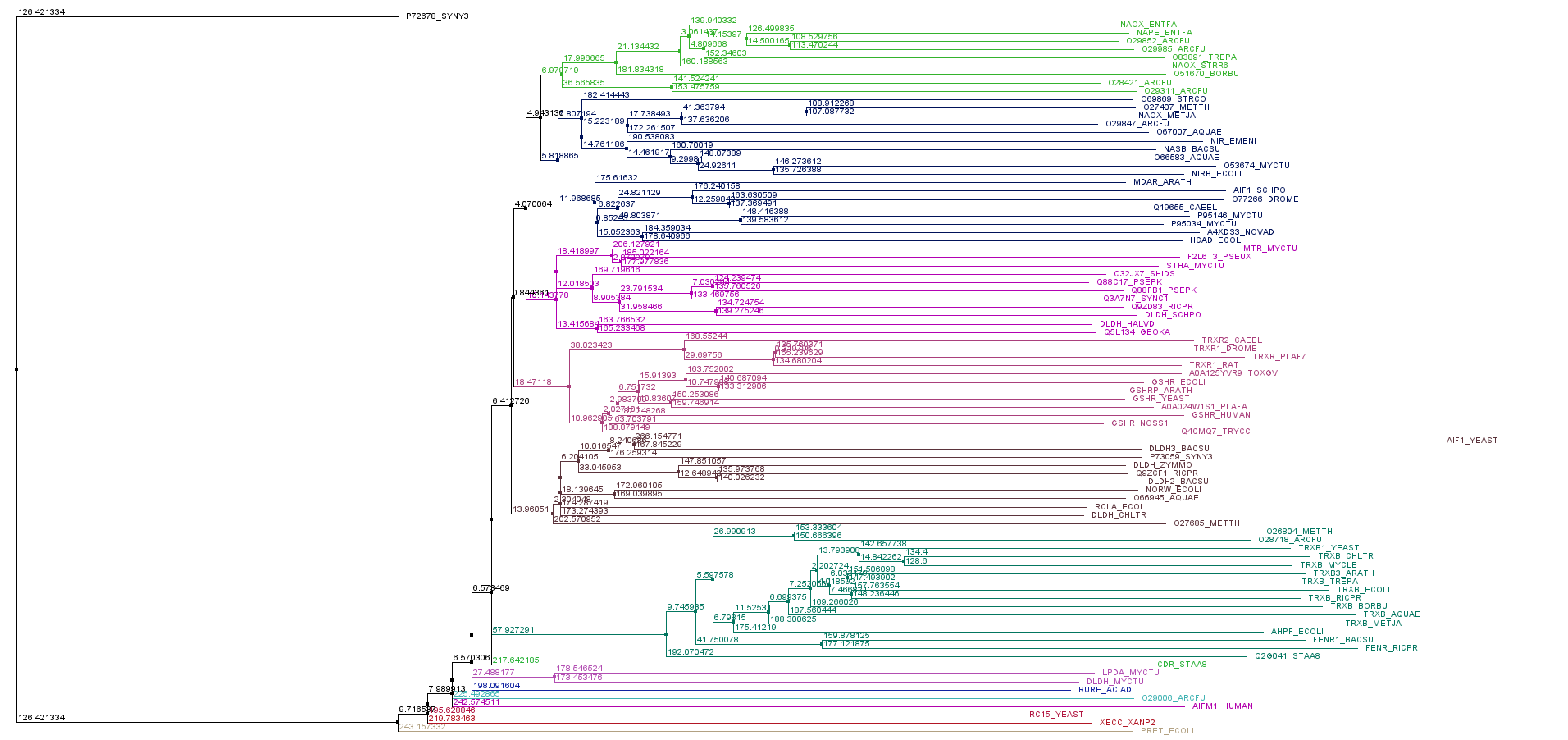
Была выделена клада, состоящая из 9 последовтаельностей
В выбранной кладе был найден мотив: [VIA][VA][VI][VIL]G[GAS]G[YFI][IV]G, который был найден в 15 последовательностях во всем выравнивании, 9 из которых в выбранной кладе, остальные 6 не приурочены к какой-то определенной кладе.Думаю, что можно назвать этот мотив специфичным к выбранной кладе.
Задание 3. PSI-BLAST
Был выбран AC белка P74518 из списка. Белок из цианобактерии Synechocystis sp., необходимый для димеризации активных 70S рибосом в 100S, которые трансляционно неактивны.
Стабилизировть результат удалось за 4 итерации.
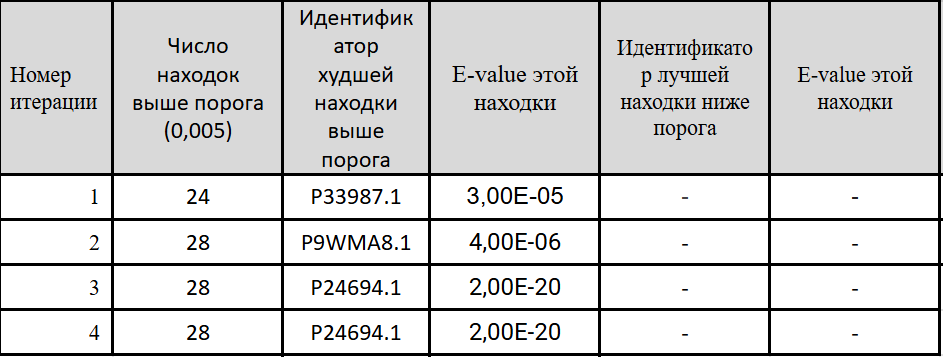
Исходя из названия, можно сделать вывод что белки выполняют примерно ту же функцию, что и исследуемый.
Для данного семейства не бело белков с E-value выше порога, поэтому разница E-value между худшей "правильной" находкой и "лучшей" неправильной довольно высока.
Думаю, что вероятнее всего, что находки действительно составляют семейство гомологичных белков.
Задание 4. Поиск мотивов de novo с помощью MEME
Были выбраны последовательности (201 последовтаельность), имеющие следующую доменную архитектуру. Из них была отобрана часть(85).
Затем была запущена программа MEME на кодомо с помощью следующей команды:
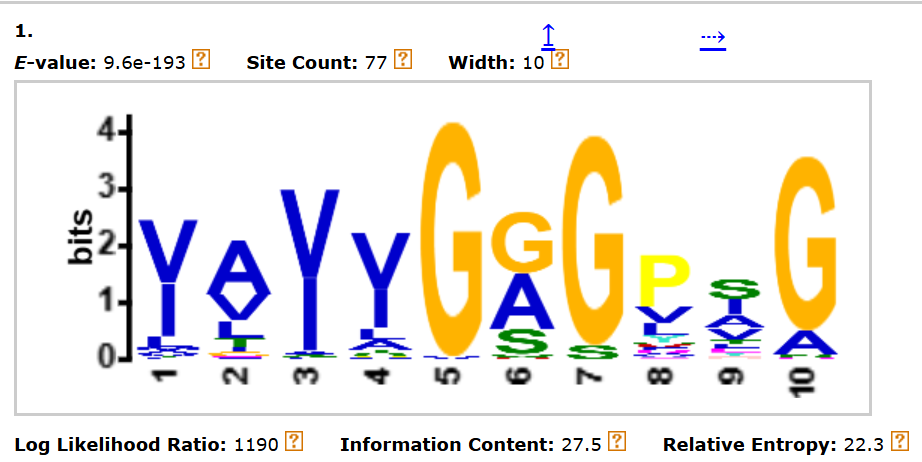
meme seq4_red.fa -o resultss_meme -minw 4 -maxw 10 -nmotifs 10
Самой первой находкой был мотив, очень похожий на мотив из п.1. с высоким уровнем достоверности.
Для программы FIMO я отобрала 297 последовательностей из 1142 чтобы проверить наличие данного мотива в остальных последовательностях. Программа была запущена с помощью следующей команды:
fimo resultss_meme/meme.txt seq5.fa
В 260 последовательностях из 297 был найден этот мотив.
5.Представленность сайта GATC в геноме Rickettsia asiatica
В данном задании была построена гистограмма контрастов obs/exp по методу Карлина с соав. для сайта GATC и, для сравнения, всех сайтов длины 4, полученных перестановками (без повторений) букв A, T, G, C.
Это было сделано с помощью этой команды:
cbcalc -s sites.txt -M -o GATC.txt Arc.fasta
Выдача доступна по ссылке
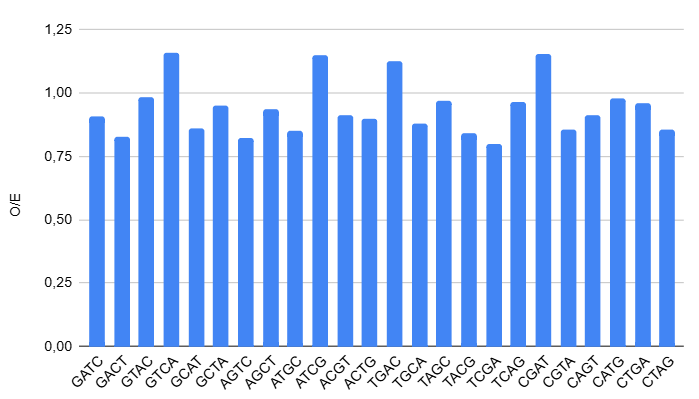
Бактерии используют системы рестрикции-модификации , чтобы защищаться от фагов и других мобильных генетических элементов.
Если у бактерии есть рестриктаза , которая распознаёт сайт GATC, но нет соответствующей модификационной метилазы, то собственные сайты GATC будут разрезаться , что вредно для клетки.
Чтобы избежать этого, такие сайты могут исчезать из генома в процессе эволюции.
В геноме бактерии Rickettsia asiatica частота встречамости сайта GATC лишь немного ниже ожидаемой(показатель O/E - 0.909)