В данном практикуме было выполнено выравнивание последовательностей цитохромов B и реконструировано филогенетичсекое дерево тремя способами:
1. Программой fastme с оценкой эволюционных расстояний как p-distance
2. Программой fastme с оценкой эволюционных расстояний с помощью MtREV
3. Программой iqtree с параметрами по умолчанию
Выравнивание было выполнено с помощью программы muscle на kodomo.
Реконструирование дерева с помощью fastme было выполнено с помощью команд:
fastme -i cyb.phy -o ppp.tre -pp
fastme -i cyb.phy -o mmm.tre -pM
Реконструирование дерева с помощью iqtree было выполнено с помощью команды:
iqtree -s cyb.phy
Изображение реконстрированных деревьев
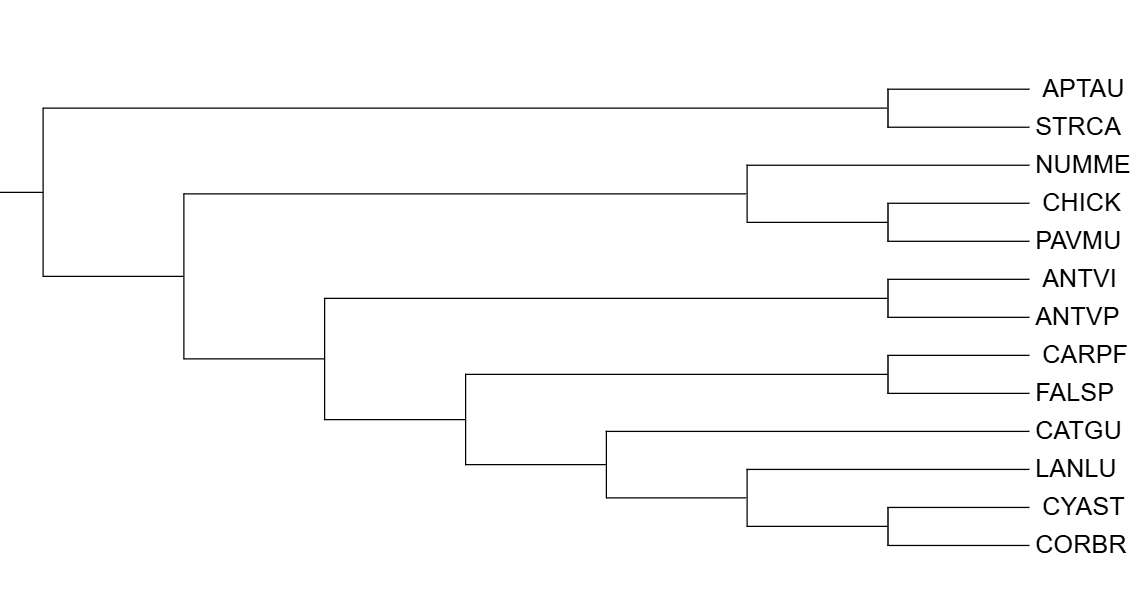
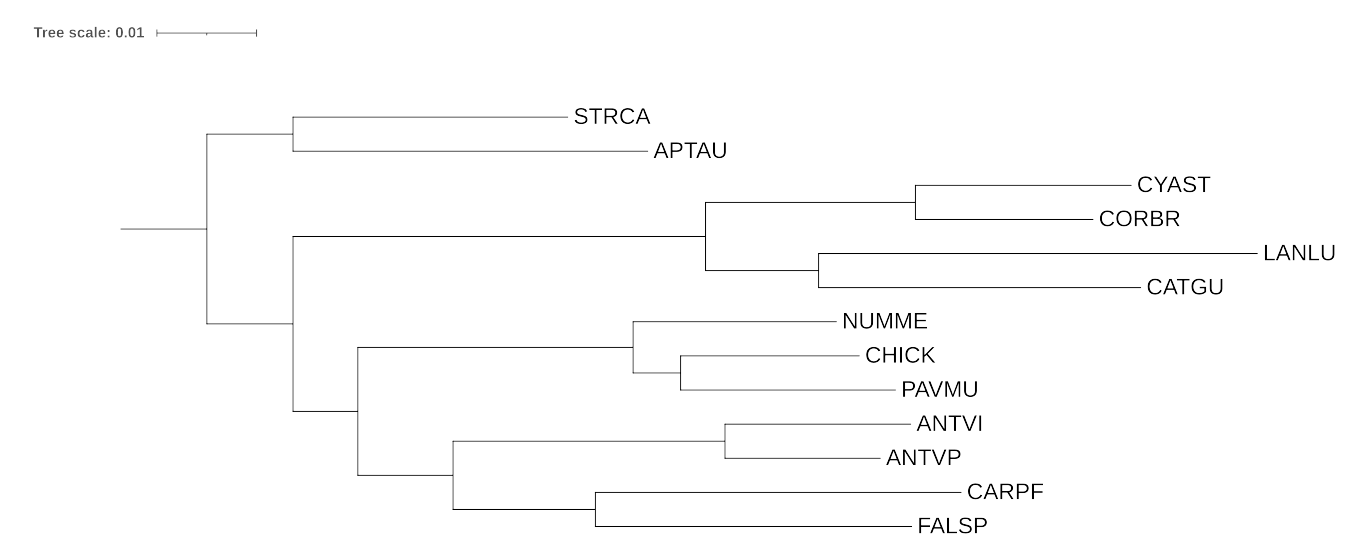
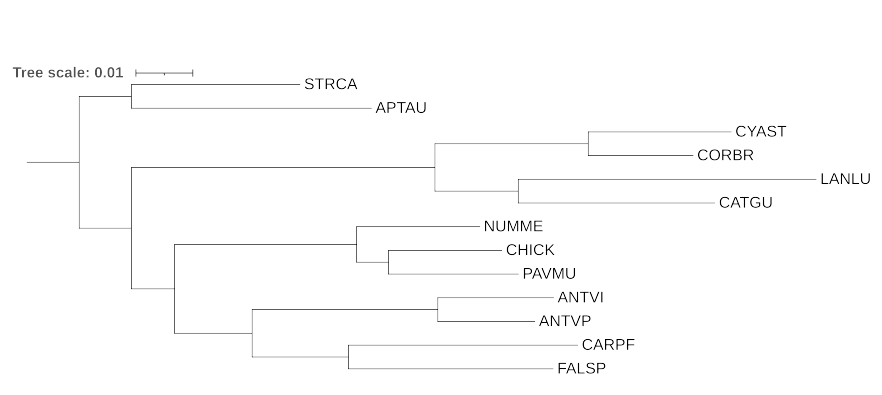
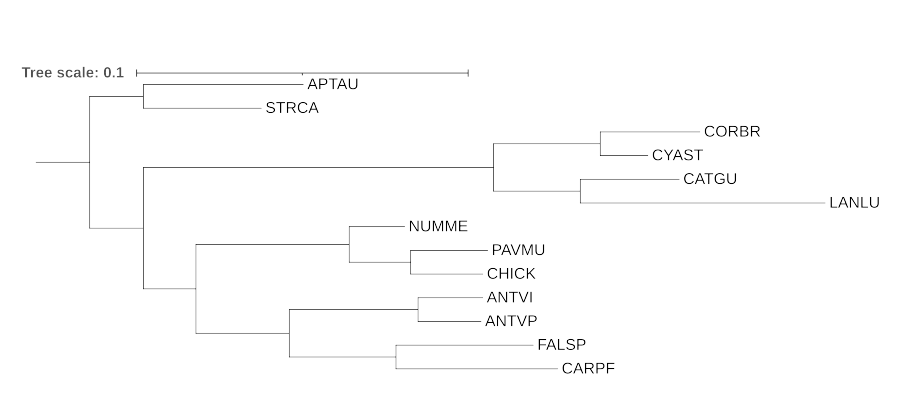
Все три реконтруированных дерева оказались очень похожи.
Ошибочно не реконструированные клады:
1. (LANLU,(CYAST,CORBR));
2.((CARPF,FALSP),(CATGU,(LANLU,(CYAST,CORBR)));
3.((ANTVI,ANTVP),((CARPF,FALSP),(CATGU,(LANLU,(CYAST,CORBR))));
Ошибочно реконструированнные клады:
1.(LANLU,CATGU);
2.((ANTVI,ANTVP),(CARPF,FALSP));
3.((NUMME,(CHICK,PAVMU)),((ANTVI,ANTVP),(CARPF,FALSP)));