1. Реконструкция дерева по нуклеотидным последовательностям
Для выбранных животных было реконструировано дерево по последовательностям малой РНК митохондриальных рибосом (12S rRNA) с помощью программы iqtree (выравнивание проводилось с помощью muscle)
У организма Catharus guttatus(CATGU) не нашлось последовательности митохондриального генома, поэтому была взята последовательность близкородственного вида Catharus fuscescens(CATFU).
К сожалению, не нашлось полной последовательности митохондриального генома ни у Cyanocitta stelleri(CYAST), ни у близкородственных видов, поэтому дерево было построенно без этой последовательности.
Так как в прошлом практикуме все 3 дерева, построенных по последовательностям митохондриального белка оказались одинаковы, то сравнение проводится только с одним из них.
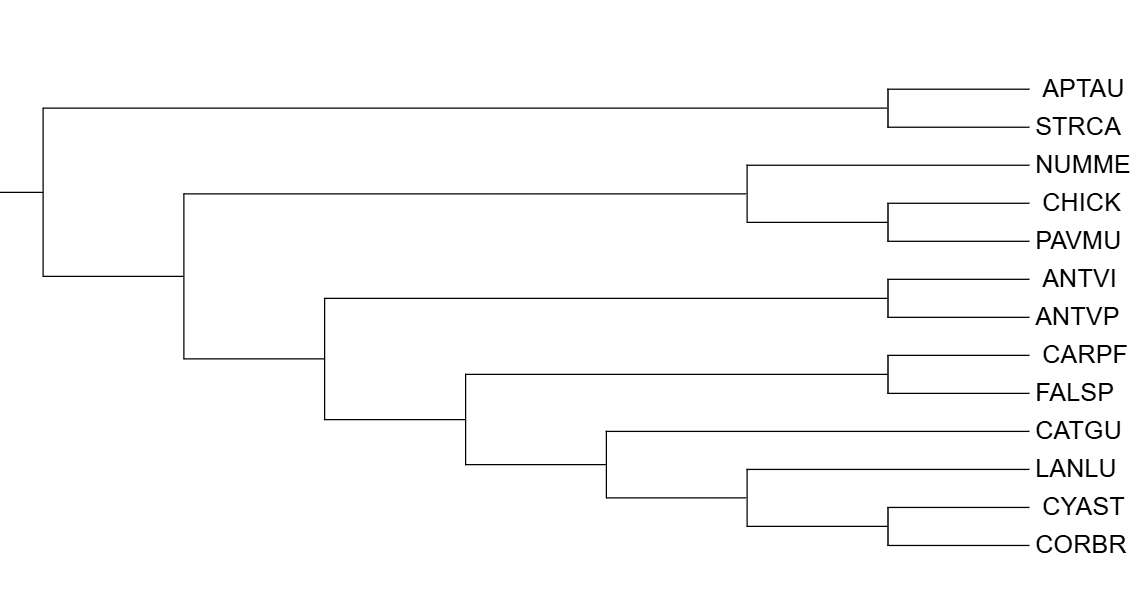
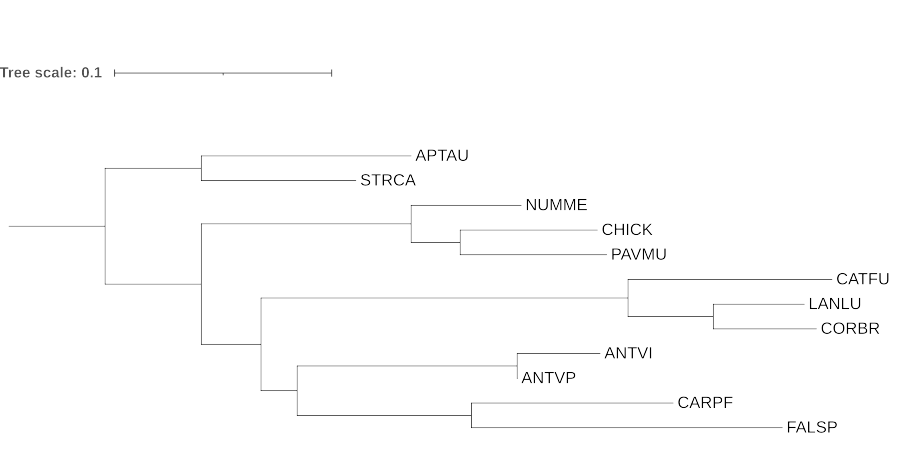
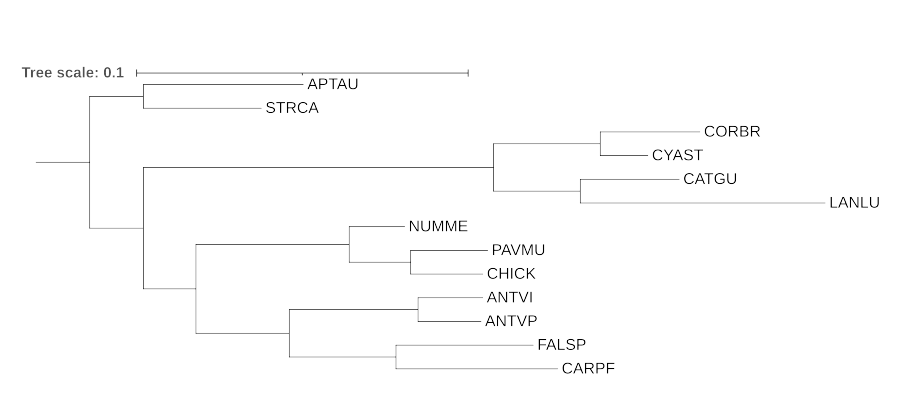
Сравнение с деревом, построенным с помощью NCBI:
В целом дерево достаточно похоже, но есть ряд различий:
Неточность заключается в том, что вместо клады ((ANTVI,ANTVP),((CARPF,FALSP),(CATGU,(LANLU,(CYAST,CORBR)))) реконструирована клада ((CATFU,(LANLU,CORBR)),((ANTVI,ANTVP),(CARPF,FALSP)))
В целом можно сказать, что дерево, построенное на основе последовательностей 12s rRNA оказалось ближе к дереву, построенноу по NCBI по сравнению с деревом, построенном на основе последовательностей цитохрома b.
2. Укоренение во внешнюю группу
В качестве внешней группы был выбран Тритон обычновенный(Lissotriton vulgaris, LISVU), который приналделижит таксону Земноводные и достаточно далеко отстоит от остальных организмов, принадлежащих таксону Птицы.
Дерево было построено с помощью программы iqtree и укоренено в ветвь, ведущую к внешней группе.
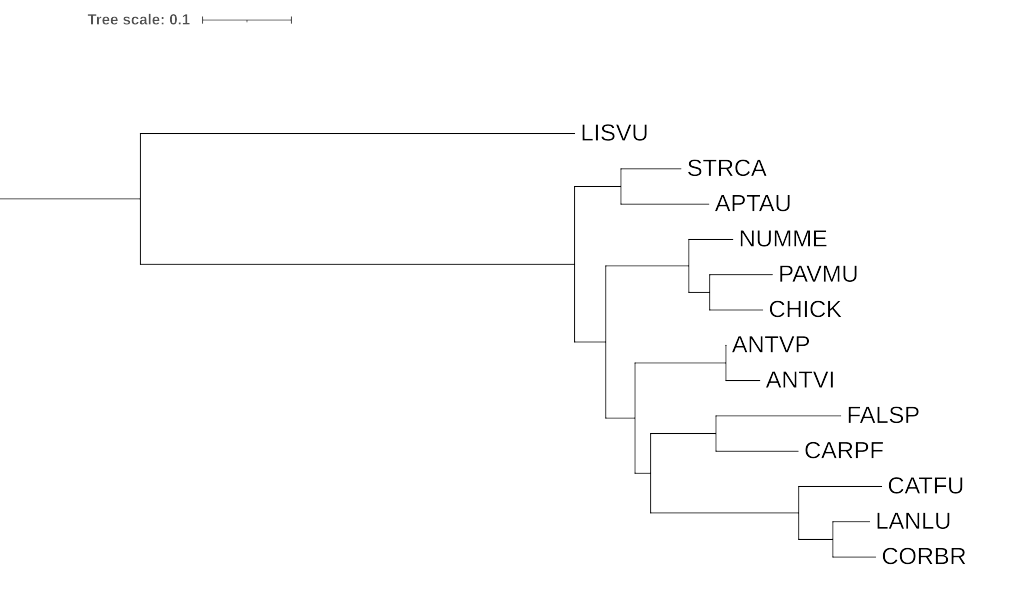
Получилось дерево полностью повторяющее дерево, построенное с помощью NCBI Taxonomy.
3. Бутстреп
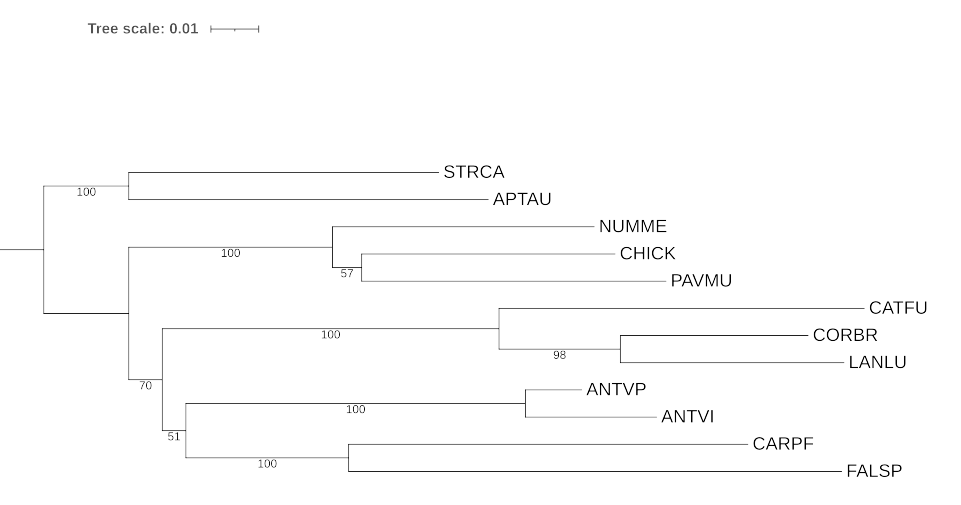
По числам на ветвях мы можем судить о достоверности каждой ветви
Наименьший вес(51) действительно у неправильно реконструированной клады ((ANTVP,ANTVI),(CARPF,FASPF)); неправильно реконструированная клада ((CATFU,(LANLU,CORBR)),((ANTVI,ANTVP),(CARPF,FALSP))) также имеет небольшой вес. Но существуют и противоречия, так правильно рекоструированная клада (CHICK,PAVMU) имеет также маленький вес