1. Составление списка гомологичных белков, включающих паралоги
Для выполнения задания были выбраны 8 бактерий отдела Pseudomonadota:
- Acidiphilium cryptum(ACICJ)
- Bartonella henselae(BARHE)
- Burkholderia mallei (BURMA)
- Shewanella denitrificans(SHEDO)
- Polynucleobacter asymbioticus(POLAQ)
- Paracoccus denitrificans(PARDP)
- Saccharophagus degradans(SACD2)
- Thiobacillus denitrificans(THIDA)
В протеомах этих бактерий был осуществлен поиск гомологов белка CLPX_ECOLI программой blastp с порогом e-value 0.0001.
Выдача поиска доступна по ссылке
Для выполнения использовались следующие команды:
cat * > proteomes.fasta
makeblastdb -in proteomes.fasta -dbtype prot
blastp -task blastp -query P0A6H1.fasta -db proteomes.fasta -out out.txt -outfmt 6 -evalue 0.0001
2. Реконструкция и визуализация
Предварительно были получены fasta-последовательности находок и проведено выравнивание с помощью программы muscle.
Дерево было реконструировано с помощью программы fastme с оценкой эволюционных расстояний как p-distance. Дерево было укоренено в среднюю точку.
Формула Newick для дерева доступна по ссылке
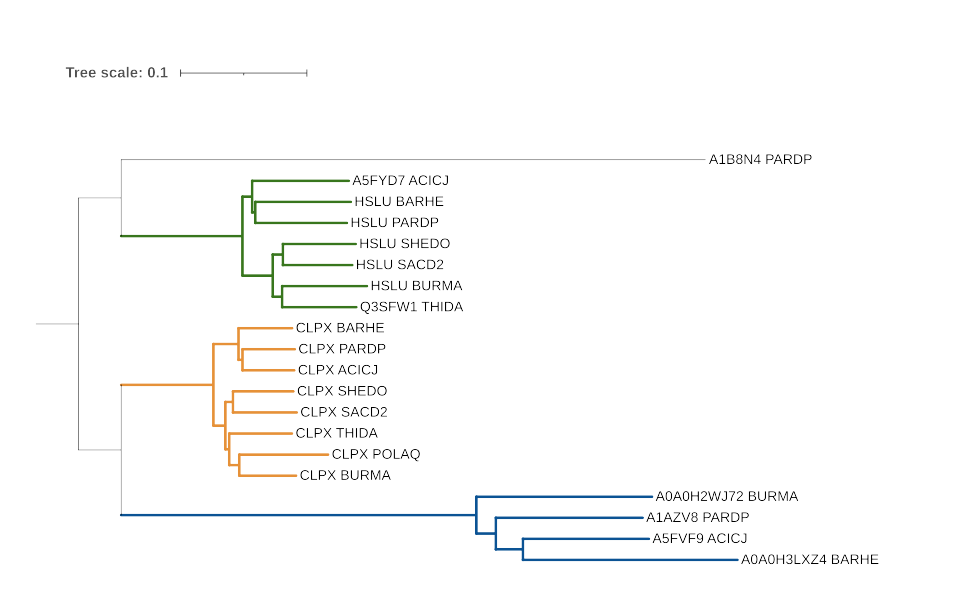
Ортологи — это гомологичные белки из разных организмов, разошедшиеся в процессе видообразования.
Паралоги — это гомологичные белки, принадлежащие одному организму.
Примеры ортологов на полученном дереве: HSLU_BARHE и HSLU_PARDP; HSLU_SHEDO и HSLU_SACD2; CLPX_PARDP и CLPX_ACICJ
Примеры паралогов на полученном дереве: CLPX_BARHE и HSLU_BARHE; CLPX_BURMA и HSLU_BURMA; Q3SFW1_THIDA и CLPX_THIDA
Сравнение реконструированной филогении ортологических групп с филогенией бактерий
Верной филогений я считала указанную в задании к данному праткикуму
1. Ортологическая группа с белками HSLU (обозначена зеленым цветом)
В данную группу не вошёл белок из бактерии POLAQ. Реконструированная филогения белков соответсвует филогении бактерий.
2. Ортологическая группа с белками CLPX (обозначена оранжевым цветом)
В эту группу не вошли белки всех выбранных бактерий, но филогения реконстрирована не совсем верно. Так вместо клады (ACICJ,(BARHE, PARDP)) появляется клада (BARHE, (PARDP,ACICJ)).
3. Ортологическая группа, обозначенная синим цветом.
В данную группу не вошли белки следующих бактерий: SHEDO, SACD2, THIDA, POLAQ. С оставшимися бактериями филогения реконструирована также не совсем верно: вместо клады (ACICJ,(BARHE, PARDP)) появляется клада (PARDP, (ACICJ, BARHE))