Докинг низкомолекулярных лигандов в структуру белка
Цель данного занятия ознакомится с возможностями докинга низкомолекулярного лиганда в структуру белка В этом занятии мы будем пользоваться пакетом Autodock Vina и Autodock tools.
Работать будем с белком лизоцимом, структура которого была построена на основе гомологичного моделирования на прошлом занятии.
- Для начала попробуем провести докинг одного из мономеров сахара (NAG) из прошлого занятия.
В банке pdb нашли SMILES нотацию для NAG. Это удобно сделать на странице структуры 1lmp. Сохраним эту анотацию в файл nag.smi.
C помощью obgen постройте 3D структуру этого сахара в pdb формате.
obgen nag.smi > mol-файл
babel -imol nag.mol -opdb nag.pdb
файл nag.pdb
Скриптом prepare_ligand4.py из пакета Autodock tools создайте pdbqt файл лиганда. На выходе получаем файл nag.pdbqt.
Так же, скриптом prepare_receptor4.py из пакета Autodock tools создадим pdbqt файл нашего белка (seq.B99990001.pdbqt).
Теперь надо создать файл с параметрами докинга vina.cfg. Для докинга необходимо указать область структуры белка в которой будет происходить поиск места для связывания. Удобно его задать как куб с неким центором. Координаты центра мы определим из модели комплекса, которую мы построили на прошлом занятии. Выберем атом сахара, находящийся в центре сайта связывания (атом N2B), и извлечем из pdb-файла его координаты (40.833 41.640 26.495).
- Проводим первый докинг:
vina --config vina.cfg --receptor prot.pdbqt --ligand nag.pdbqt --out nag_prot.pdbqt --log nag_prot.log
файлы nag_prot.pdbqt и nag_prot.log.
Энергии 3-х лучших расположений и геометрическая разница между ними:
mode |
affinity(kcal/mol) |
dist from (rmsd l.b.) |
best mode (rmsd u.b.) |
1 |
-5.2 |
0.000 |
0.000 |
2 |
-5.2 |
2.010 |
2.186 |
3 |
-4.7 |
3.069 |
4.524 |
В PyMol загрузим файлы nag_prot.pdbqt и prot.pdbqt. Включим анимацию. Отобразим все состояния на одной картинке.
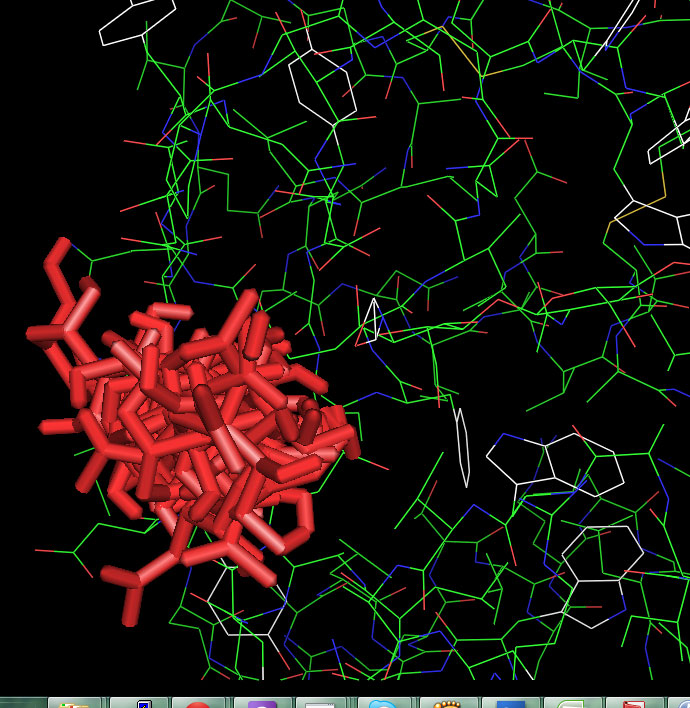
Здесь мы видим, что лиганду доступен некоторый объём в центре связывания и он, возможно, связывается с белком не очень жёстко.
- Проведём докинг рассматривая подвижность некоторых боковых радикалов белка. Сначала разобьем белок на две части, подвижную и неподвижную. Для подвижной части выберем 3 аминокислоты которые вы использовали в прошлом задании для позиционирования лиганда.
prepare_flexreceptor4.py -r prot.pdbqt -s GLU1_ASN5_ASP13
и проведем докинг:
vina --config vina.cfg --receptor prot.pdbqt --ligand nag.pdbqt --out nag_prot.pdbqt --log nag_prot.log
файлы vina_prot_flex.pdbqt и vina_prot_flex.log.
Энергии 3-х лучших расположений и геометрическая разница между ними:
mode |
affinity (kcal/mol) |
dist from (rmsd l.b.) |
best mode (rmsd u.b.) |
1 |
-4.7 |
0.000 |
0.000 |
2 |
-4.7 |
1.510 |
1.594 |
3 |
-4.3 |
1.536 |
4.087 |
* В PyMol загрузим файлы nag_prot_flex.pdbqt и prot_rigid.pdbqt. Включим анимацию. Отобразим все состояния на одной картинке.
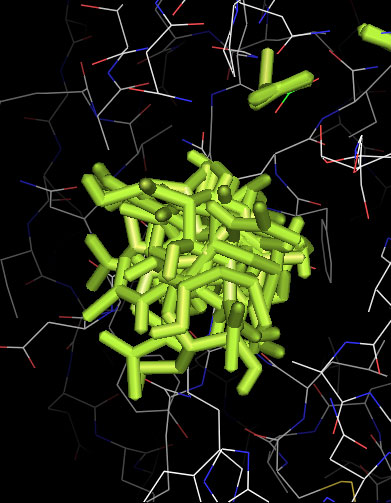
Здесь мы видим, что лиганду доступен больший объём в центре связывания за счет подвижности частей белка,но не смотря на это, свободная энергия гораздо выше, что говорит о некоторой невыгодности по сравнению с жесткой структурой белка, хотя подвижная структура наиболее близка приближена к биологической модели.
NAG содержит в себе СH3C(=O)NH группу. Создадим 3 лиганда где метильный радикал этой группы будет заменён на OH (nag2.smi), NH2 (nag3.smi), H (nag4.smi). Для каждого из этих лигандов проведём обыкновенный докинг.
файлы для :
|
mode |
affinity (kcal/mol) |
dist from (rmsd l.b.) |
best mode (rmsd u.b.) |
2 |
1 |
-5.2 |
0.000 |
0.000 |
|
2 |
-5.0 |
2.403 |
5.397 |
|
3 |
-4.8 |
2.196 |
5.108 |
|
|
|
|
|
3 |
1 |
-5.6 |
0.000 |
0.000 |
|
2 |
-5.4 |
2.932 |
5.143 |
|
3 |
-5.4 |
1.739 |
2.130 |
|
|
|
|
|
4 |
1 |
-4.9 |
0.000 |
0.000 |
|
2 |
-4.7 |
2.464 |
4.453 |
|
3 |
-4.6 |
2.080 |
5.032 |
