Освоение возможностей PyMol как пакета для моделирования
Суть задания состоит в поэтапном освоении возможностей PyMol как пакета для моделирования.
Мутагенез в белке записи 1LMP
Средствами Tc1/Tk интерфейса (Wizard->Mutagenesis) проведем мутацию в белке записи 1LMP, которая должна привести к потере связывания с лигандом.
HET NAG A 130 14
HET NAG A 131 14
HET NDG A 132 15
В качестве лиганда выступают NDG (2-(acetylamino)-2-deoxy-a-d-glucopyranose) и NAG (N-ACETYL-D-GLUCOSAMINE).
На рис. можно видеть лиганд и а.о. asp52, взаимодействующий с лигандом, а также представлена структура белка.
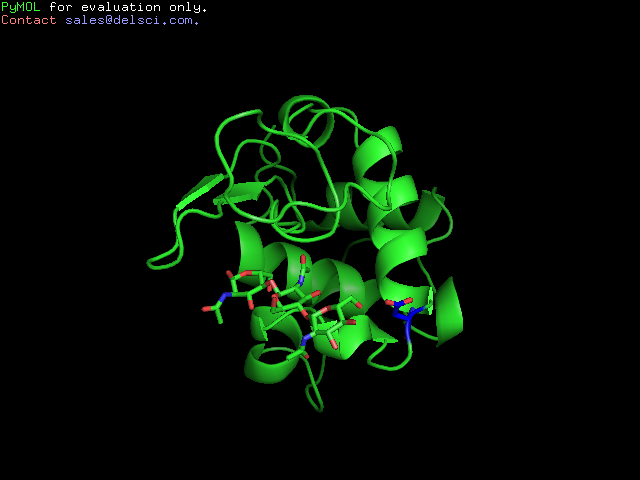
Проведем мутацию в аминокислоте, образующей водородную связь с лигандом. Заменим asp на glu. Предполагается, что это ухудшит взаимодействие с лигандом.
Создание анимационного ролика
Используя команды mset, mview, super, translate, создадим анимационный ролик, где происходит совмещение белков и показывается место мутации. Ролик был размещен на youtube: http://www.youtube.com/watch?v=thYEpHDOsNA
Создание валентинки
Создадим поли-аланиновую последовательность в форме валентинки. Для изменения формы будем использовать режим Editing и манипулирование торсионными углами. Ctrl+RightClick выбор связи, Ctrl+LeftClick вращение.
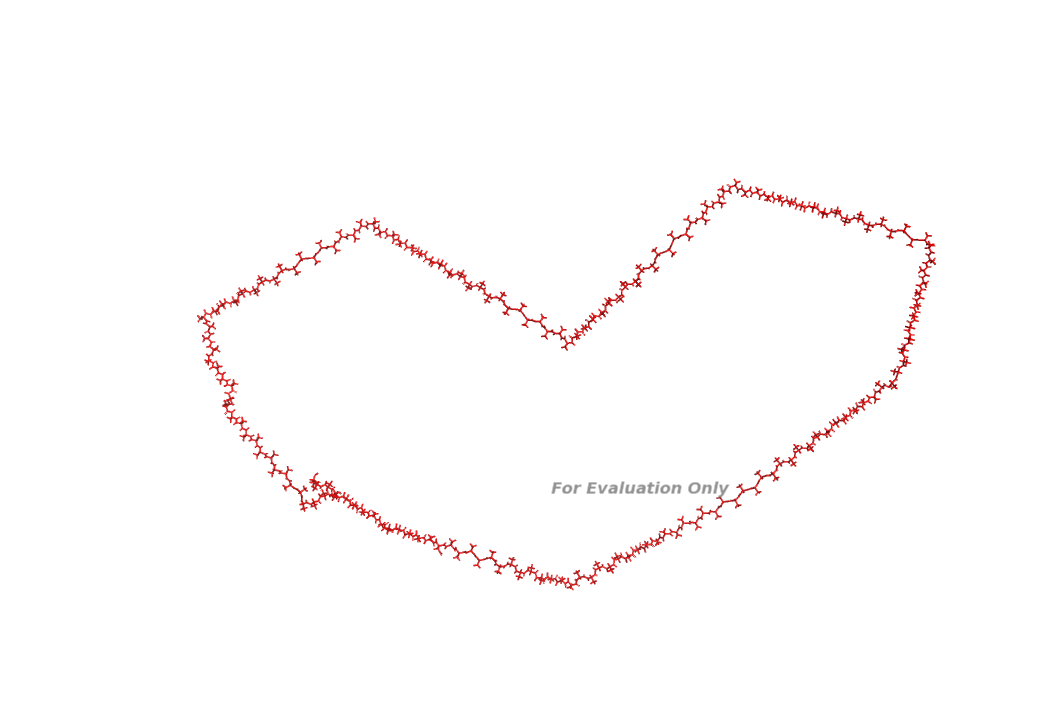
Построения поли-аланиновой альфа-спирали длиной 100 аминокислот
Напишем скрипт для построения поли-аланиновой альфа-спирали длиной 100 аминокислот.
frag ala
python
for x in range(2,100):
cmd.edit("/ala///" + str(x) + "/C")
editor.attach_amino_acid("pk1","ala")
for x in range(3,99):
cmd.set_dihedral("/ala///" + str(x-1) + "/N", "/ala///" + str(x-1) + "/CA", "/ala///" + str(x-1) + "/C",
"/ala///" + str(x) + "/N", -47)
cmd.set_dihedral("/ala///" + str(x-1) + "/CA", "/ala///" + str(x-1) + "/C", "/ala///" + str(x) + "/N",
"/ala///" + str(x) + "/CA", 180)
cmd.set_dihedral("/ala///" + str(x-1) + "/C", "/ala///" + str(x) + "/N", "/ala///" + str(x) + "/CA",
"/ala///" + str(x) + "/C", -57)
python end
