Изучение работы методов контроля температуры в GROMACS
- Подготовим файл координат и файл топологии. Создадим индекс файл в котором будет группа из одной молекулы этана на основе 38 gro файлов с молекулами этана.
make_ndx -f box_38.gro -o 1.ndx
- Выбераем остаток номер 1. Появляется новая группа. Теперь создадим gro файл с одной молекулой и зададим ячейку. При запуске editconf выбераем номер, соответствующей группе из одной молекулы.
editconf -f box_38.gro -o et1.gro -n 1.ndx
#зададим ячейку и расположим молекулу по центру ячейку
editconf -f et1.gro -o et.gro -d 2 -c
- Исправим файл топологии et.top из прошлого задания. В разделе [ molecules ] изменим количество молекул этана с 38 на 1.
- Даны 5 файлов с разными параметрами контроля температуры:
- an.mdp - метод Андерсена для контроля температуры.
- be.mdp - метод Берендсена для контроля температуры.
- nh.mdp - метод Нуза-Хувера для контроля температуры.
- sd.mdp - метод стохастической молекулярной динамики.
- vr.mdp - метод "Velocity rescale" для контроля температуры.
Напишем скрипт bash для работы с 5ю системами.
- Создаём отдельную папку для каждого метода.
mkdir ${i}
- Построим входные файлы для молекулярно-динамического движка mdrun с помощью grompp:
grompp -f ${i}.mdp -c et.gro -p et.top -o et_${i}.tpr
- У нас получилось 5 tpr файлов. Теперь для каждого из них запустим mdrun.
mdrun -deffnm et_${i} -v -nt 1
Анализ
Для каждой из 5 систем проведем конвертацию в pdb и просмотрим в PyMol.
trjconv -f ${i}/et_${i}.trr -s ${i}/et_${i}.tpr -o ${i}/et_${i}.pdb
- Наблюдения:
- В методе Андерсена наблюдаются небольшие колебания по длинам связей и валентным углам. Вращение в данном случае отсутствует.
- В методе Берендсена сперва мы наблюдать различные колебания и вращения.
- В методе Нуза-Хувера мы наблюдаем небольшие колебания и вращение по связи С-С.
- В методе стохастической молекулярной динамике молекула очень быстро вращается и перемещается. Сложно уследить что происходит.
- Метод "Velocity rescale". Можно наблюдать более амплитудные колебания и уменьшенное вращение по связи С-С.
Сравним потенциальную энергию связи и кинетическую энергию для каждой из 5 систем
g_energy -f et_${i}.edr -o et_${i}_en.xvg
- Построим графики изменения энергий. Для этого в баш скрипте добавим строчки скрипта для Gnuplot:
echo -e "set datafile commentschars '#@&'
set term 'png'
set output '${i}/en_${i}.png'
plot '${i}/et_${i}_en.xvg' using 1:2, '${i}/et_${i}_en.xvg' using 1:3" > ${i}/en_${i}.gnu
gnuplot < ${i}/en_${i}.gnu
Рассмотрим распределение длинны связи С-С за время моделирования
- Сначала создадим индекс файл с одной связью.
В текстовом редакторе создим файл b.ndx со следующим содержимым:
[ b ]
1 2
- запустим утилиту по анализу связей g_bond:
g_bond -f et_${i}.trr -s et_${i}.tpr -o bond_${i}.xvg -n b.ndx
Построим графики распределения длинн связей. Для этого в баш скрипте добавим строчки скрипта для Gnuplot:
echo "set datafile commentschars '#@&'
set term 'png'
set output '${i}/bond_${i}.png'
plot '${i}/bond_${i}.xvg' with boxes" > ${i}/bond_${i}.gnu
gnuplot < ${i}/bond_${i}.gnu
Полученные изображения:
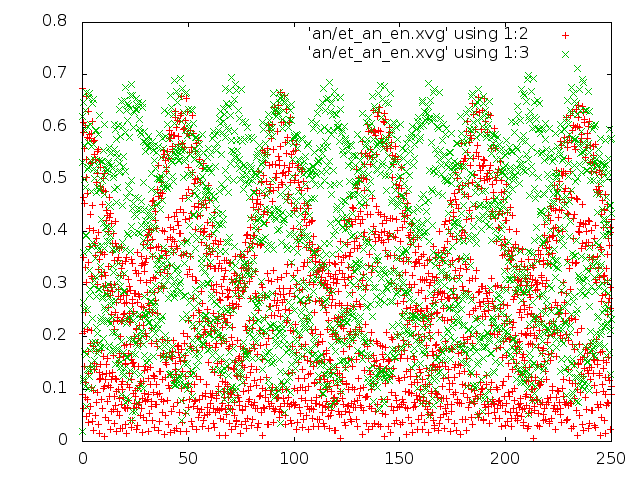
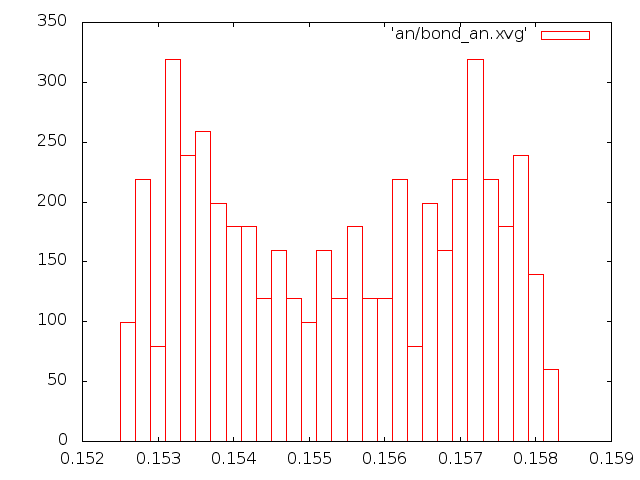
- метод Андерсена.
- энергия для каждого состояния: красным цветом потенциальная энергия связей, зелёным - кинетическая энергия
- распределения длин связей
- энергия для каждого состояния: красным цветом потенциальная энергия связей, зелёным - кинетическая энергия
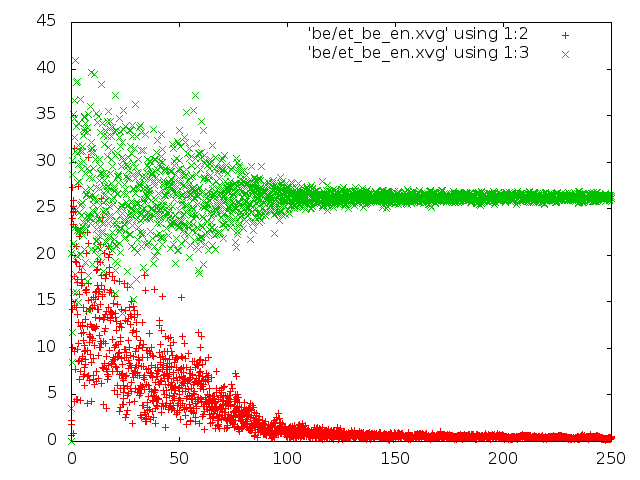
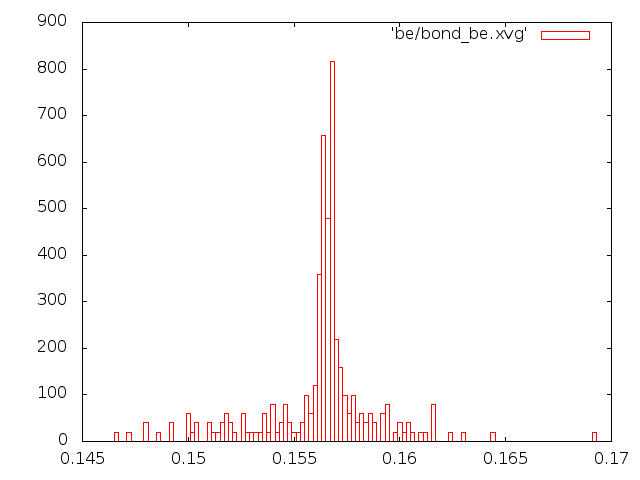
- метод Берендсена.
- энергия для каждого состояния: красным цветом потенциальная энергия связей, зелёным - кинетическая энергия
- распределения длин связей
- энергия для каждого состояния: красным цветом потенциальная энергия связей, зелёным - кинетическая энергия
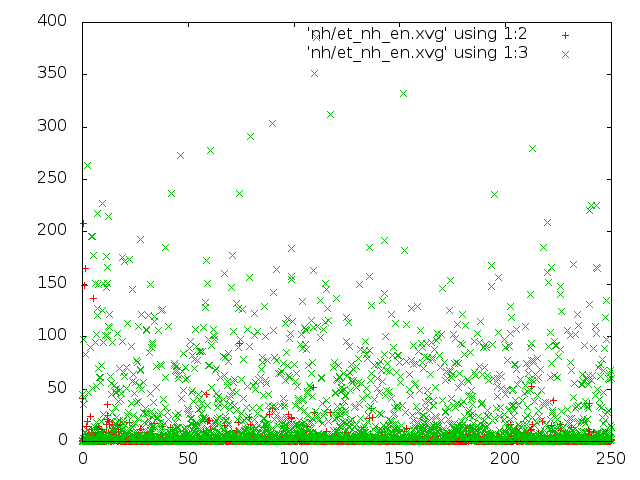
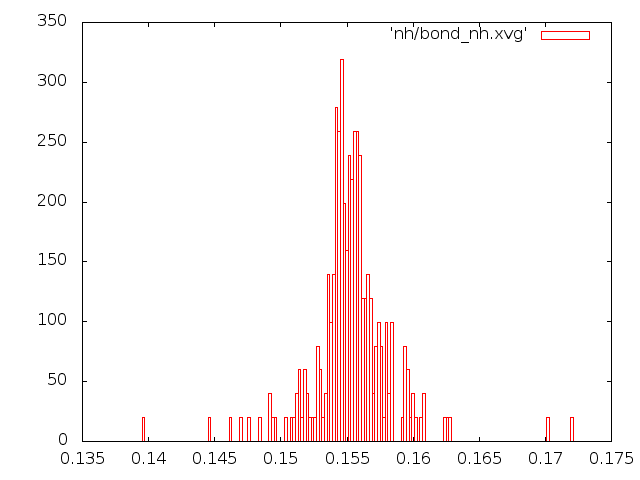
- метод Нуза-Хувера.
- энергия для каждого состояния: красным цветом потенциальная энергия связей, зелёным - кинетическая энергия
- распределения длин связей
- энергия для каждого состояния: красным цветом потенциальная энергия связей, зелёным - кинетическая энергия
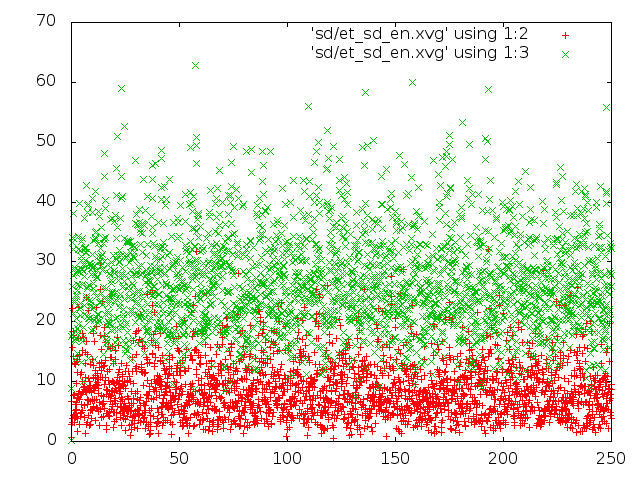
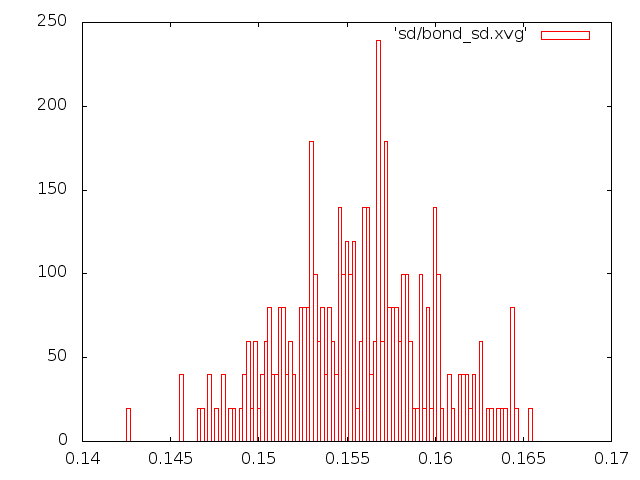
- стохастический метод.
- энергия для каждого состояния: красным цветом потенциальная энергия связей, зелёным - кинетическая энергия
- распределения длин связей
- энергия для каждого состояния: красным цветом потенциальная энергия связей, зелёным - кинетическая энергия
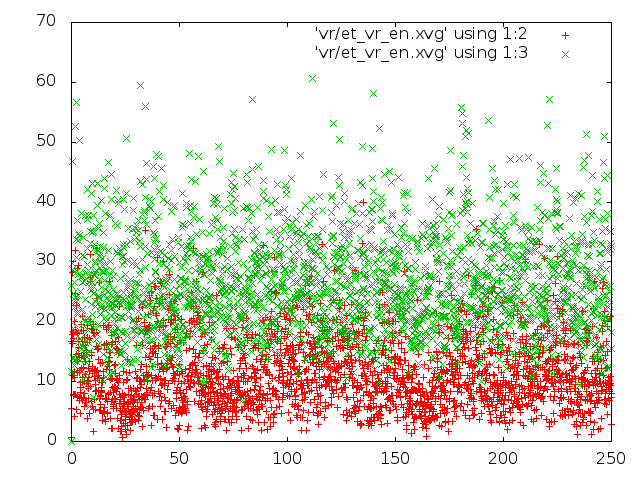
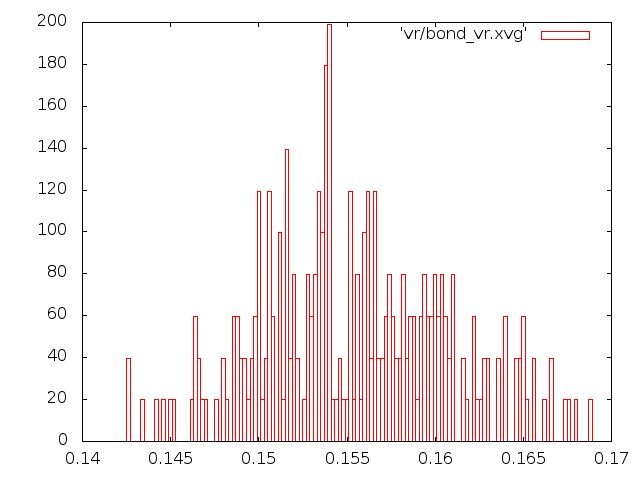
- Метод "Velocity rescale".
- энергия для каждого состояния: красным цветом потенциальная энергия связей, зелёным - кинетическая энергия
- распределения длин связей
- энергия для каждого состояния: красным цветом потенциальная энергия связей, зелёным - кинетическая энергия
- Исходя из всех наблюдений, можно сказать, что самым реалистичным является метод "Velocity rescale". Метод Андерсена и Берендсена совершенно не соответствует общему виду распределения Больцмана.