Знакомство с PyMol
При помощи программы PyMOL было обнаружено, что в структуре записи 1KMY банка PDB имеется ион железа. На рисунке показаны ион железа и остатки, которые с ним связаны координационными связями.

Изучение записи 1GT0
- В записи описан транскрипционный фактор SOX-2.
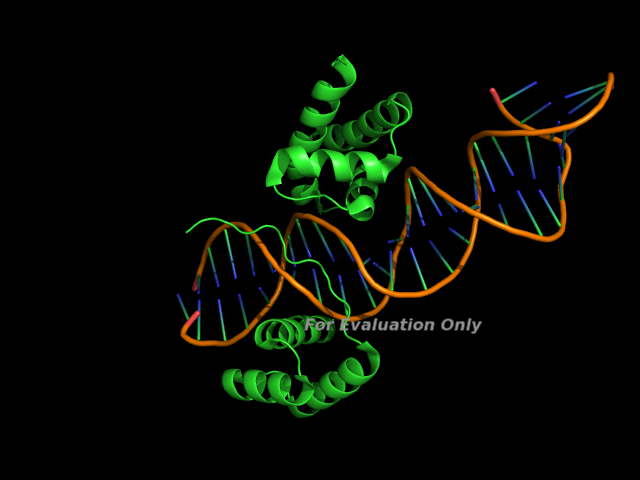
Как выяснилось, при изучении в программе PyMOL, в участке цепи C с 75 по 100 остаток имеется разрыв, который начинается с 77 по 96 а.о. цепи С. Скорее всего этот участок просто является вариабельной областью, которая в кристалле при ренгенно-структурном анализе, тяжело или может быть невозможно зафиксировалась.
Изучение записи 1DLP
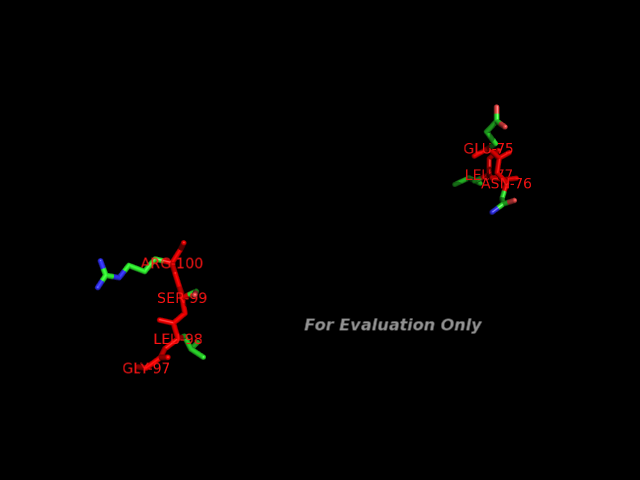
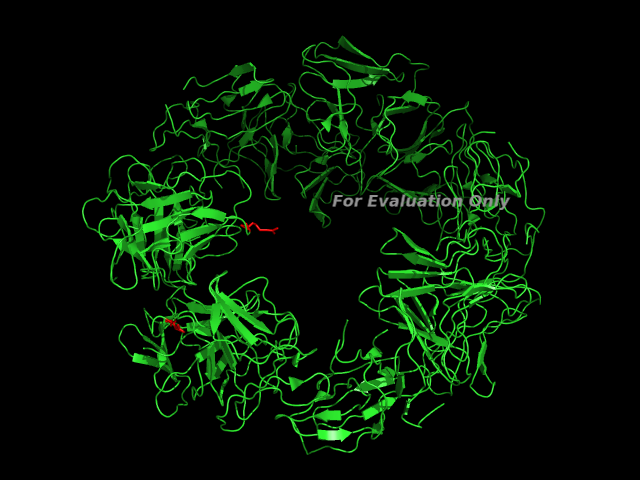
В записи представлена структура сахар-связывающего белка (LECTIN SCAFET PRECURSOR). Красным цветом отмечены аминокислотные остатки 136 цепи A и 167 цепи С.
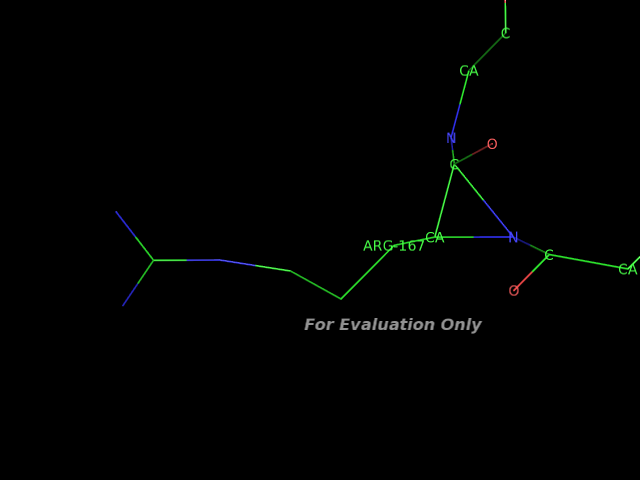
В результате анализа выяснилось, что аргинин 167 цепи С содержит дополнительную связь между остовными атомами: азотом и и углеродом, что в действительности не может быть.
Также, аспаргин 136 цепи A помимо ложной связи между остовными атомами имеет связи в функциональной группе в несвойственных положениях: CB-ND1 и CB-OD1.
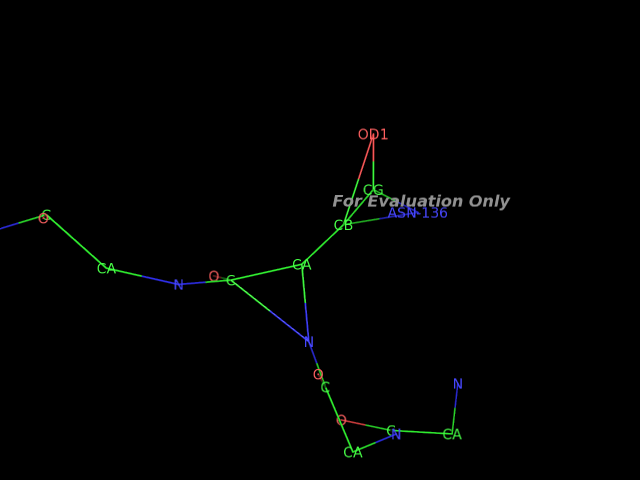
Возможно, из-за того, что атомы находятся на расстояние сравнимым с расстоянием ковалентной связи, то в связи с этим программа автоматически посчитала ее за полноценную ковалентную связь, что изображено на картинках выше.
Скрипт для PyMOL, показывающий аспаргин 136 цепи A. (для просмотра, после применения скрипта, необходимо приблизить а.о.)
