С помощью Putty, протокола ssh присоединилась
к серверу kodomo.cmm.msu.ru. С помощью
команд ls и cd .. перешла в рабочую директорию Term3/Pr2.
Команды, указывающие путь к пакету 3DNA:
export PATH=${PATH}:/home/preps/golovin/progs/X3DNA/bin
export X3DNA=/home/preps/golovin/progs/X3DNA
Дальнейшие действия:
fiber -h # Получаем справку по fiber, результаты описаны выше.
fiber -a gatc-a.pdb
# Получаем такой результат:
agalicina@kodomo:~/Term3/Pr2$ fiber -a gatc-a.pdb # Введенная команда.
Fiber data in directory: /home/preps/golovin/progs/X3DNA/FIBER/# Берем данные для работы из директории...
Structure #1; Twist: 32.7 (degrees); Rise: 2.548 (Angstrom) # Выбран тип структуры, характеристики.
Input your base sequence with only A,C,G & T: # Введите базовую последовательность, содержащую только A,C,G и T:
1. From a data file (complete sequence) # из файла,
2. From keyboard (enter only the repeating sequence) # с клавиатуры.
Your choice (1 or 2, Dft: 2): 2 # (Вводим ответ) Пользователем выбран вариант 2.
Repeating unit (Dft: polyA): gatc # Повторяющаяся единица: (вводим) gatc
Repeating unit: GATC
Number of repeats (Dft: 10): 5 # Количество повторов: (вводим) 5
# Создан файл gatc-a.pdb с требуемой структурой.
fiber -a gatc-a.pdb
# Получаем такой результат:
agalicina@kodomo:~/Term3/Pr2$ fiber -b delete.pdb
Fiber data in directory: /home/preps/golovin/progs/X3DNA/FIBER/
Structure #4; Twist: 36.0 (degrees); Rise: 3.375 (Angstrom)
Input your base sequence with only A,C,G & T:
1. From a data file (complete sequence)
2. From keyboard (enter only the repeating sequence)
Your choice (1 or 2, Dft: 2): 2
Repeating unit (Dft: polyA): gatc
Repeating unit: GATC
Number of repeats (Dft: 10): 5
# Создан файл gatc-b.pdb с требуемой структурой.
fiber -a gatc-a.pdb
# Получаем такой результат:
agalicina@kodomo:~/Term3/Pr2$ fiber -z delete.pdb # Введенная команда.
Fiber data in directory: /home/preps/golovin/progs/X3DNA/FIBER/# Берем данные для работы из директории...
Structure #15; Twist: -60.0 (degrees); Rise: 7.250 (Angstrom) # Выбран тип структуры, характеристики.
Repeating unit: GC:GC # Повторяющаяся единица - GC:GC
Number of repeats (Dft: 10): 5 # Количество повторов: (вводим) 5
# Создан файл gc-z.pdb с требуемой структурой.
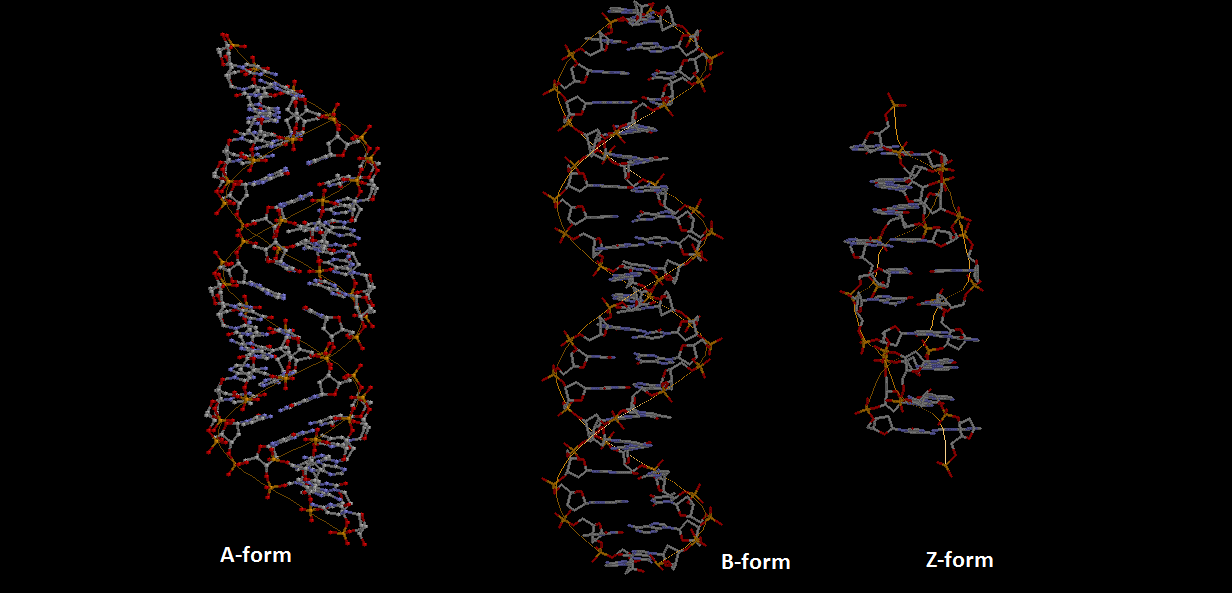
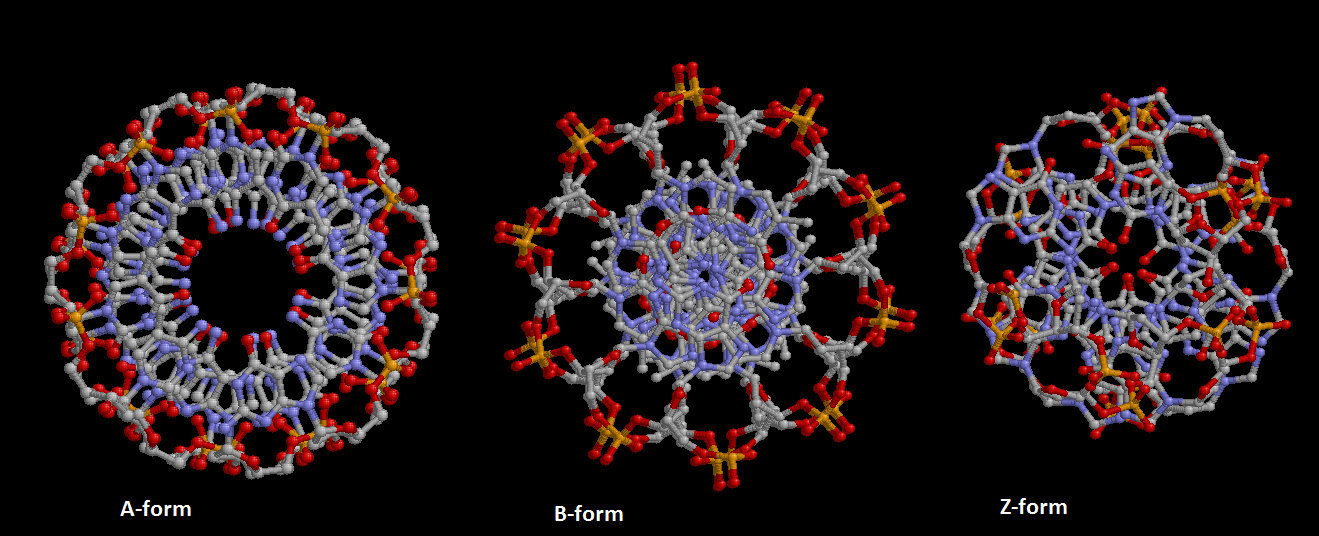
Полученные файлы:
gatc-a.pdb
gatc-b.pdb
gc-z.pdb
Проверка файлов в RasMol: