Задача: найти в геноме Geobacillus thermodenitrificans последовательности, кодирующие
похожие на белок RESA из Bacillus subtilis.
Для решения задачи выбираю из пакета BLAST+ программу tbblastn (запрос - белок, база данных - нуклеотидные последовательности).
С помощью команды makeblastdb создаю три файла базы данных по геному данной бактерии.
На всякий случай создаю файл-справку по команде. Интересующие возможности:
** Input options
-in
Input file/database name; the data type is automatically detected, it may
be any of the following:
FASTA file(s) and/or
BLAST database(s)
Default = `-'
-dbtype
Molecule type of input
Default = `prot'
** Output options
-out
Name of BLAST database to be created
Default = input file name provided to -in argumentRequired if multiple
file(s)/database(s) are provided as input
Выполняю команду, получаю три файла: gt.nhr, gt.nin и gt.nsq (не прилагаю к работе,
так как файлы бинарные и не читаются "глазами"):
makeblastdb -in gt_genome.fasta -out gt -dbtype nucl
Теперь читаю информацию о выбранной программе tblastn (tblastn_help.txt):
*** Input query options
-query
Input file name
Default = `-'
*** General search options
-db
BLAST database name
-out
Output file name
Default = `-'
-evalue
Expectation value (E) threshold for saving hits
Default = `10'
Предварительно получаю последовательность RESA_BACSU, хотя её можно взять со страницы прошлого семестра (resa.fasta):
seqret sw:resa_bacsu resa.fasta
Выполняю команду и получаю файлы:
tblastn -query resa.fasta -db gt -evalue 0.001 -out tblastn.txt
Получаю файл: tblastn.txt
|
Число находок с E-value < 0,001 |
1 |
|
E-value лучшей находки |
9e-08
|
|
Название последовательности с лучшей находкой |
CP000557 CP000557.1 Geobacillus thermodenitrificans NG80-2, complete
genome. (Bacterioferritin comigratory protein - из описания генома)
|
|
Координаты лучшей находки (от-до) |
от 544245 до 544550
|
|
Доля последовательности вашего белка, вошедшая в выравнивание с лучшей находкой |
(137-36+1)/179 = 0.569
|
Выравнивание:
> CP000557 CP000557.1 Geobacillus thermodenitrificans NG80-2, complete
genome.
Length=729146
Score = 48.1 bits (113), Expect = 9e-08, Method: Compositional matrix adjust.
Identities = 32/104 (31%), Positives = 51/104 (50%), Gaps = 4/104 (3%)
Frame = +3
Query 36 ISEGSDAPNFVLEDTNGKRIELSDLKGKGVFLNFWGTWCEP-CKKEFPYMANQYKHFKSQ 94
I+ G AP+F L +NGK + LSD +G+ V L F+ P C E ++Y+ F
Sbjct 544245 IAIGQPAPDFTLPASNGKMVSLSDFRGQYVVLYFYPKDMTPGCTTEACDFRDRYEQFT-- 544418
Query 95 GVEIVAVNVGESKIAVH-NFMKSYGVNFPVVLDTDRQVLDAYDV 137
G+ V + V + H F++ Y + F ++ D V + Y V
Sbjct 544419 GLNAVILGVSTDPVKRHETFIEKYQLPFLLLSDEQHHVAELYGV 544550
Фрагмент из описания генома, перекрывающийся с находкой:
544239..544715
/codon_start=1
/transl_table=11
/locus_tag="GTNG_0489"
/product="Bacterioferritin comigratory protein"
/db_xref="GOA:A4IKL7"
/db_xref="InterPro:IPR000866"
/db_xref="InterPro:IPR012336"
/db_xref="InterPro:IPR024706"
/db_xref="UniProtKB/TrEMBL:A4IKL7"
/protein_id="ABO65871.1"
/translation="MIIAIGQPAPDFTLPASNGKMVSLSDFRGQYVVLYFYPKDMTPGC
TTEACDFRDRYEQFTGLNAVILGVSTDPVKRHETFIEKYQLPFLLLSDEQHHVAELYGV
WKKKRNFGKEYMGIERSTFIIAPDGTLVKEWRGVKVKGHVDEALAEVAQLASSR"
Из заданной директории с kodomo скачала файл
с последовательностью нуклеотидов. Задача: получить информацию об этой последовательности, используя
BLASTN на EBI.
Последовательность действий.
Захожу на сайт http://www.ebi.ac.uk/.
Меню Tools -> Similarity&Homology -> NCBI BLAST
Ссылка Nucleotide Databases
Снятие галочки напротив EMBL Release
Треугольник EMBL Release
Треугольник EMBL Prokaryote
Галочка напротив EMBL Standard Prokaryote
Данная последовательность и вид окна результатов с
показанными последовательностями:
ttccggggtatcgtacccgtcgaccgtctaccttcgcttcctgacccgcaggcaatccag
ttctggatgggtaaaaacgctcacatgctccattacgcgattgggcgtgacggcgacgcg
gtcaatttcttcgctgttgttgagggacctcaaccctggttggacgagacccgctgggtg
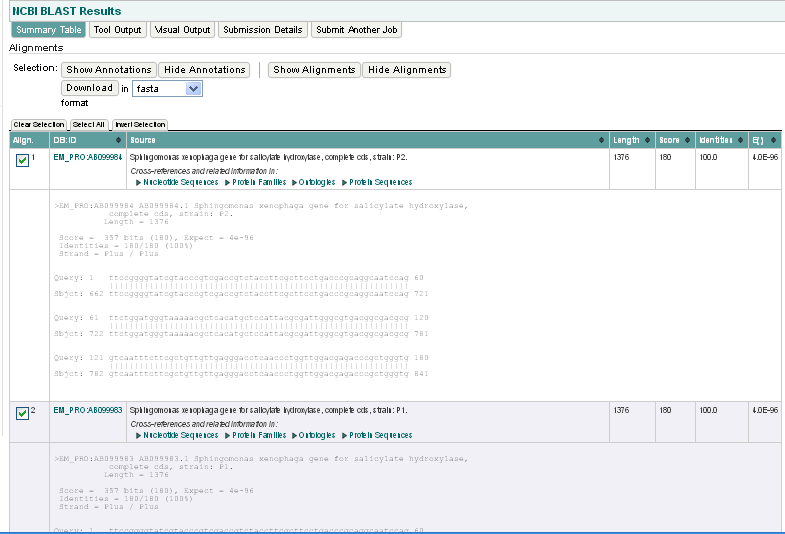
Полностью идентичных находок - шесть. Все находки - ген салицилат гидроксилазы
из разных штаммов Sphingomonas xenophaga.
Таксономия Sphingomonas xenophaga:
Bacteria
Proteobacteria
Alphaproteobacteria
Sphingomonadales
Sphingomonadaceae
Sphingobium
Результат (общее название записей
"Sphingomonas xenophaga gene for salicylate
hydroxylase, complete cds"):
| EM_PRO | Штамм | Начало | Конец | Соответствие цепей ДНК | Описание в FT | Направление белка |
| AB099984 | P2 | 662 | 841 | комплементарное | salicylate hydroxylase P96555 | прямое |
| AB099983 | P1 | 662 | 841 | комплементарное | salicylate hydroxylase P96556 | прямое |
| AB099982 | AJ8 | 662 | 841 | комплементарное | salicylate hydroxylase P96557 | прямое |
| AB099981 | AJ6 | 662 | 841 | комплементарное | salicylate hydroxylase P96558 | прямое |
| AB099980 | TB4 | 662 | 841 | комплементарное | salicylate hydroxylase P96559 | прямое |
| AB000564 | - | 917 | 1096 | комплементарное | salicylate hydroxylase P96560 | прямое
|
Через ссылку на запись EMBL в файле
последовательности белка RESA_BACSU
нашла координаты гена этого белка в
полном геноме Bacillus subtilis subsp. subtilis str. 168 (запись AL009126, участок complement(2420804..2421343)).
Вырезала с помощью seqret c опцией -sask.
Произвела поиск гомологов с помощью BLASTN.
Получила, что программма не находит никаких гомологов. Выходной файл:
file.txt
Поиск по гену в геноме - гораздо более строгий, чем поиск по белковому запросу. Причина -
меньшая консервативность последовательности ДНК по сравнению с белком, не учитывается значимость сходства отдельных триплетов. Таким образом, поиском Query ДНК по геномной базе данных
гораздо имеет большую точность, но меньшую чувствительность.
При изменении длины слова находки начинают появляться.

