Укоренение в среднюю точку
Укореним дерево, построенное ранее методом neighbor-joining (см. Реконструкция и сравнение деревьев. Расстояния между последовательностями.) Дерево выглядело так: +-------BURCA
+------------------1
! +------RALPJ
!
! +------VIBCH
! +--------3
! ! +-----VIBFM
2------------4
! ! +------ECOLI
! +-5
! +---------PROMH
!
+--------------------------------BRAJA
Укореним дерево в среднюю точку. Для этого воспользуемся программой retree пакета PHYLIP.
Для этого скопируем файл со скобочной формулой, выданный программой neighbor, в файл с именем intree, после чего запустим программу retree пакета PHYLIP. Укореним дерево в среднюю точку и сохраним полученную скобочную формулу в файле outtree.tre. Теперь запустим полученный файл в программе TreeDyn и сохраним изображение в формате GIF. Итак, укорененное программой retree дерево выглядит следующим образом:
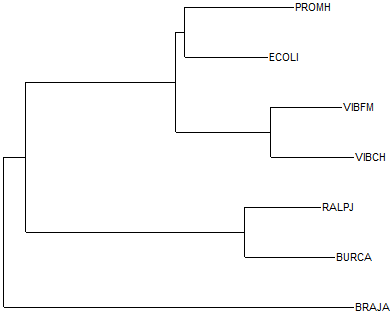
Сравним это дерево с правильным деревом:
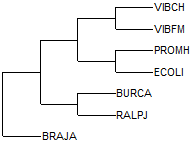
Как видно, деревья полностью совпадают. Программа retree укоренила полученное методом neighbor-joining дерево правильно. Укоренение произошло по тривиальной ветви {BRAJA}, что говорит об отделении класса Alphaproteobacteria от других представленных классов раньше всех. Значит, при достаточном количестве данных и при достаточном расстоянии между последовательностями может применяться укоренение в среднюю точку.
Использование аутгруппы
Реконструируем программой fprotpars укорененное дерево отобранных бактерий по семейству белков EFTS, используя в качестве аутгруппы белок EFTS_BACSU (белок того же семейства из сенной палочки - Bacillus subtilis). Для этого добавим в файл efts.fasta с последовательностями белка EFTS из выбранных бактерий последовательность EFTS из бактерии BACSU с помощью команды:seqret sw:EFTS_BACSU stdout >> efts.fastaПереименуем измененный файл, присвоив ему имя efts1.fasta. Теперь построим выравнивание всех последовательностей, используя программу muscle. На выходе получаем файл efts1_aligned.fasta, который теперь подадим на вход программе fprotpars. В результате получаем файл c деревом и его скобочной формулой. Обработаем полученное дерево программой retree, указав в качестве действия "select an Outgroup", а в качестве номера - тот, что программа retree присвоит листу BACSU (в нашем случае это номер 1). Получим файл outtree1.tre cо скобочной формулой дерева. Уберем из формулы аутгруппы и откроем файл в программе TreeDyn. Итак, укорененное с помощью аутгруппы дерево выглядит так:
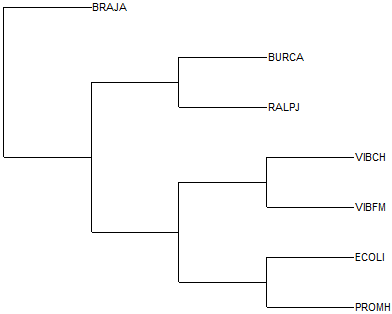
Это дерево полностью совпадает с правильным деревом. Значит, для укоренения дерева можно применять хорошо подобранную аутгруппу, далеко отстоящую от представленных таксонов.
Бутстрэп
Проведем бутстрэп-анализ филогении, используя программу fprotpars. Для этого вначале создадим 100 бутстрэп-реплик выравнивания белков протеобактерий программой fseqboot. На вход программе подадим файл efts_aligned.fasta с выравненными последовательностями белков. На выходе получаем файл efts_aligned.fseqboot со 100 бутстрэп-репликами.Теперь создадим по полученным репликам деревья программой fprotpars, для чего просто подадим выходной файл программы fseqboot на вход программе fprotpars. В результате получаем файл efts2_aligned.fprotpars со 100 изображениями деревьев, соответствующим репликам, и файл efts2_aligned.treefile со скобочными формулами всех этих деревьев.
Ну и, наконец, создадим из полученных деревьев единое дерево по принципу "расширенного большинства" (extended majority rule tree). Для этого подадим на вход программе fconsense файл с деревьями, выданный программой fprotpars. На выходе имеем файл efts3_aligned.fconsense с единым деревом. Оно выглядит следующим образом:
+------BURCA
+100.0-|
+100.0-| +------RALPJ
| |
+-79.5-| +-------------BRAJA
| |
| | +------VIBFM
+------| +-------100.0-|
| | +------VIBCH
| |
| +---------------------------PROMH
|
+----------------------------------ECOLI
Если укоренить это дерево по тривиальной ветви {BRAJA}, то оно совпадет со всеми полученными ранее деревьями, то есть бутстрэп-анализ привел нас к правильному результату. Интересно, что почти все таксоны бактерий, представленные среди выбранных 7 бактерий, получили 100%-ую поддержку бутстрэп-анализом. Исключение составляет лишь порядок Enterobacteriales, получивший поддержку в 79,5% (к которому относятся бактерии PROMH и ECOLI). Теперь посмотрим на ветви выходного файла, не получившие большинства. Это всего две ветви: ветвь {PROMH, VIBFM, VIBCH}, получившая 12%-ю поддержку, и ветвь {ECOLI, VIBFM, VIBCH}, получившая 8,5%-ю поддержку.
Обе эти ветви не являются правильными и именно они "виновны" в не 100%-й поддержке порядка Enterobacteriales (100 - (8,5 + 12) = 79,5). В целом, бутстрэп-анализ дал верный результат и может применяться для построения неукорененного дерева.