Построение дерева
Отобранные бактерии
| Название | Мнемоника |
| Bradyrhizobium japonicum | BRAJA |
| Burkholderia cenocepacia | BURCA |
| Ralstonia pickettii | RALPJ |
| Escherichia coli | ECOLI |
| Vibrio cholerae | VIBCH |
| Vibrio fischeri | VIBFM |
| Proteus mirabilis | PROMH |
Скобочная формула дерева
(BRAJA,((RALPJ,BURCA),((ECOLI,PROMH),(VIBFM,VIBCH))));
Изображение дерева
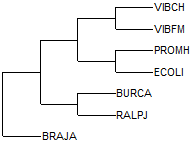
Ветви дерева
Дерево содержит 4 нетривиальные ветви:- {VIBCH,VIBFM} против {PROMH,ECOLI,BURCA,RALPJ,BRAJA};
- {PROMH,ECOLI} против {VIBCH,VIBFM,BURCA,RALPJ,BRAJA};
- {PROMH,ECOLI,VIBCH,VIBFM} против {BURCA,RALPJ,BRAJA};
- {BURCA,RALPJ} против {PROMH,ECOLI,VIBCH,VIBFM,BRAJA}.
| Название | Таксономия |
| Bradyrhizobium japonicum | Bacteria; Proteobacteria; Alphaproteobacteria; Rhizobiales; Bradyrhizobiaceae; Bradyrhizobium |
| Burkholderia cenocepacia | Bacteria; Proteobacteria; Betaproteobacteria; Burkholderiales; Burkholderiaceae; Burkholderia; Burkholderia cepacia complex |
| Ralstonia pickettii | Bacteria; Proteobacteria; Betaproteobacteria; Burkholderiales; Burkholderiaceae; Ralstonia |
| Escherichia coli | Bacteria; Proteobacteria; Gammaproteobacteria; Enterobacteriales; Enterobacteriaceae; Escherichia |
| Vibrio cholerae | Bacteria; Proteobacteria; Gammaproteobacteria; Vibrionales; Vibrionaceae; Vibrio |
| Vibrio fischeri | Bacteria; Proteobacteria; Gammaproteobacteria; Vibrionales; Vibrionaceae; Aliivibri |
| Proteus mirabilis | Bacteria; Proteobacteria; Gammaproteobacteria; Enterobacteriales; Enterobacteriaceae; Proteus |
Теперь посмотрим на построенное филогенетическое дерево и сравним его с данными таблицы:
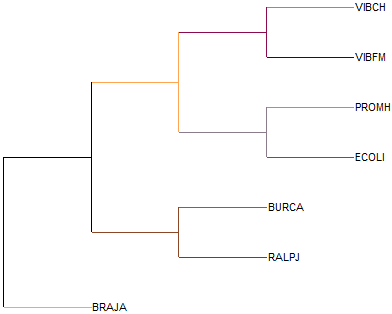
Как видно из таблицы, бактерия Bradyrhizobium japonicum единственная из выбранных принадлежит классу Alphaproteobacteria - на филогенетическом дереве она (BRAJA) также отделена собственной ветвью (соответствующая ветвь окрашена в золотой цвет). Из оставшихся 6 бактерий две (Burkholderia cenocepacia и Ralstonia pickettii - соответственно BURCA и RALPJ) принадлежат классу Betaproteobacteria, а остальные 4 бактерии - классу Gammaproteobacteria. На дереве эти два класса разделены двумя различными ветвями (ветвь Betaproteobacteria окрашена в коричнеый цвет, а ветвь Gammaproteobacteria - в бежевый цвет). Более того, если посмотреть на таксономию бактерий Burkholderia cenocepacia и Ralstonia pickettii (BURCA и RALPJ), то видно, что они принадлежат не только к одному классу, но и к одному порядку (Burkholderiales), и даже к одному семейству (Burkholderiaceae), так что общая коричневая ветвь этих двух бактерий объединяет в себе эти таксоны. Но вот роды у этих бактерий разные. Burkholderia cenocepacia (BURCA) принадлежит роду Burkholderia, а Ralstonia pickettii (RALPJ) - роду Ralstonia (на филогенетическом дереве эти роды отделены двумя ветвями: розовая ветвь соответствует роду Burkholderia, а красная ветвь - роду Ralstonia).
Теперь подробнее рассмотрим класс Gammaproteobacteria. Из таблицы мы видим, что две бактерии этого класса принадлежат порядку Enterobacteriales (бактерии Escherichia coli и Proteus mirabilis - соответственно ECOLI и PROMH), а две другие - порядку Vibrionales (Vibrio fischeri и Vibrio cholerae - cоответственно VIBFM и VIBCH). На филогенетическом дереве ветвь, соответствующая порядку Vibrionales, окрашена в фиолетовый цвет, а ветвь, соответствующая порядку Enterobacteriales, - в серый цвет. Бактерии порядка Enterobacteriales принадлежат одному семейству (Enterobacteriaceae), но разным родам: бактерия Escherichia coli (ECOLI) принадлежит роду Escherichia (темно-зеленая ветвь на филогенетическом дереве), а бактерия Proteus mirabilis (PROMH) - роду Proteus (cалатовая ветвь на филогенетическом дереве). Бактерии порядка Vibrionales тоже принадлежат одному семейству (Vibrionaceae), но разным родам: бактерия Vibrio fischeri (VIBFM) принадлежит роду Aliivibri (темно-синяя ветвь на филогенетическом дереве), а бактерия Vibrio cholerae (VIBCH) - роду Vibrio (голубая ветвь на дереве).
Таким образом, мы видим, что построенное филогенетическое дерево правильно отражает таксономию выбранных нами бактерий.
seqret sw:EFTS_ABCDEгде ABCDE - мнемоника вида бактерии.
Теперь построим множественное выравнивание отобранных белков программой muscle. Для этого подадим на вход программе файл efts.fasta с последовательностями белка в fasta-формате. Воспользуемся командой:
muscle -in efts.fasta -out efts_aligned.fastaНа выходе получаем файл efts_aligned.fasta с выравненными последовательностями белков.
*Импортируем выравнивание в GeneDoc и изучим его подробней (см. Выравнивание белков EFTS).
На выходе имеем два файла: efts_aligned.fprotpars с изображением дерева и efts_aligned.treefile со скобочной формулой дерева.
Итак, скобочная формула реконструированного дерева выглядит так:
((((PROMH,ECOLI),(VIBFM,VIBCH)),(RALPJ,BURCA)),BRAJA);Далее его изображение:
+--PROMH
+-----6
! +--ECOLI
+-----5
! ! +--VIBFM
! +-----4
+--3 +--VIBCH
! !
! ! +--RALPJ
1 +-----------2
! +--BURCA
!
+-----------------BRAJA
Как видно из формулы и изображения, реконструированное по белку EFTS дерево полностью совпадает с исходным правильным деревом. Программа fprotpars выдала исходя из файла с выравненными последовательностями лишь одно дерево - и оно оказалось верным. Это говорит о том, что по изменениям в последовательностях ортологов белка EFTS можно в данном случае судить о родстве различных видов бактерий.
BRAJA 0.000000 1.026163 0.946313 1.058096 0.987580 0.885628 0.935298 BURCA 1.026163 0.000000 0.243689 0.919116 0.925749 0.822625 0.800204 RALPJ 0.946313 0.243689 0.000000 0.877333 0.903142 0.828251 0.841387 VIBCH 1.058096 0.919116 0.877333 0.000000 0.224188 0.380328 0.440862 VIBFM 0.987580 0.925749 0.903142 0.224188 0.000000 0.376486 0.418580 ECOLI 0.885628 0.822625 0.828251 0.380328 0.376486 0.000000 0.280400 PROMH 0.935298 0.800204 0.841387 0.440862 0.418580 0.280400 0.000000Теперь проанализируем таблицу на ультраметричность.
Для примера возьмем расстояния d(BURCA, ECOLI), d(BURCA, RALPJ) и d(RALPJ, ECOLI). Как видно из таблицы, d(BURCA, RALPJ) = 0.243689 < 0.822625 = d(BURCA, ECOLI). Если бы матрица была ультраметрической, то в таком случае, d(BURCA, ECOLI) было бы равно d(RALPJ, ECOLI). В нашем случае имеем d(BURCA, ECOLI) = 0.822625; d(ECOLI, RALPJ) = 0.828251. Эти числа почти совпадают, значит, для этих объектов гипотеза молекулярных часов справедлива.
В качестве второго примера возьмем расстояния d(VIBCH. VIBFM), d(VIBCH, ECOLI) и d(ECOLI, VIBFM). d(VIBCH, VIBFM) = 0.224188 < 0.380328 = d(VIBCH, ECOLI). В этом примере нужно сравнивать расстояния d(VIBCH, ECOLI) и d(VIBFM, ECOLI), которые должны быть равны в случае ультраметрической матрицы. В нашем случае d(VIBCH, ECOLI) = 0.380328; d(VIBFM, ECOLI) = 0.376486. Эти числа тоже почти равны между собой, значит и для этих объектов гипотеза молекулярных часов справедлива. Если посмотреть на расстояния между другими объектами, можно увидеть, что в целом, матрица достаточно близка к ультраметрической.
Проанализируем матрицу на аддитивность.
Для этого возьмем в качестве примера бактерии ECOLI, PROMH, VIBFM, VIBCH. Если свойство аддитивности для них выполнено, то d(ECOLI, VIBFM) + d(PROMH, VIBCH) = d(ECOLI, VIBCH) + d(PROMH, VIBFM), а d(ECOLI, PROMH) + d(VIBCH, VIBFM) должна быть меньше других сумм. В нашем случае d(ECOLI, PROMH) + d(VIBCH, VIBFM) = 0.280400 + 0.224188 = 0.504588. Это сумма получается действительно меньше, чем две другие суммы. Осталось лишь проверить эти оставшиеся суммы на равенство. d(VIBCH, ECOLI) + d(PROMH, VIBFM) = 0.380328 + 0.418580 = 0.798908; d(VIBFM, ECOLI) + d(VIBCH, PROMH) = 0.376486 + 0.440862 = 0.817348. Эти суммы отличаются между собой, но не очень сильно. То есть по крайней мере в этом примере расстояния не сильно отклоняются от аддитивности.
Алгоритм Neighbor-Joining выдал файл efts_aligned.fneighbor c неукорененным деревом и длинами ветвей. Изображение дерева выглядит так:
+-------BURCA +------------------1 ! +------RALPJ ! ! +------VIBCH ! +--------3 ! ! +-----VIBFM 2------------4 ! ! +------ECOLI ! +-5 ! +---------PROMH ! +--------------------------------BRAJAКак видно, это дерево совпадает с результатом программы fprotpars и с правильным деревом (если укоренить его по нижней ветви).
Алгоритм UPGMA выдал файл efts_alignedu.fneighbor c укорененным деревом и длинами ветвей. Изображение дерева выглядит так:
+----------------------------BRAJA
!
! +------BURCA
--6 +------------------2
! ! +------RALPJ
! !
+--5 +------VIBCH
! +----1
! ! +------VIBFM
+-------------4
! +-------ECOLI
+---3
+-------PROMH
Это дерево совпадает со всеми полученными прежде деревьями. Исходя из этого филогенетического дерева и из того, что матрица расстояний не сильно отличается от ультраметрической, то есть гипотиза молекулярных часов в данном случае справедлива, ветвь класса Alphaproteobacteria отделилась от других представленных классов (Betaproteobacteria и Gammaproteobacteria) раньше остальных.