Банки нуклеотидных последовательностей.
Характеристика сборки генома
Нужно оценить качество сборки генома произвольно выбранного эукариотического организма Ficedula albicollis (см. рис.1) Сборка геномов этого и организма , а также других представителей отряда воробьинообразных имеет важное значение в сравнительной геномике для выяснения эволюции птичьих геномов.

Рис.1 Изображение Ficedula albicollis мухоловки-белошейки.
Для этого генома было проведено 2 сборки (GCA_000247815.2 и GCA_000247815.1). Также для него известно 2 проекта по секвенированию (PRJNA208061 и PRJNA75089) и 1 образец (SAMN02981387).
Для сборки 2013 года. (GCA_000247815.2)
Описание образца (см. рис.2)
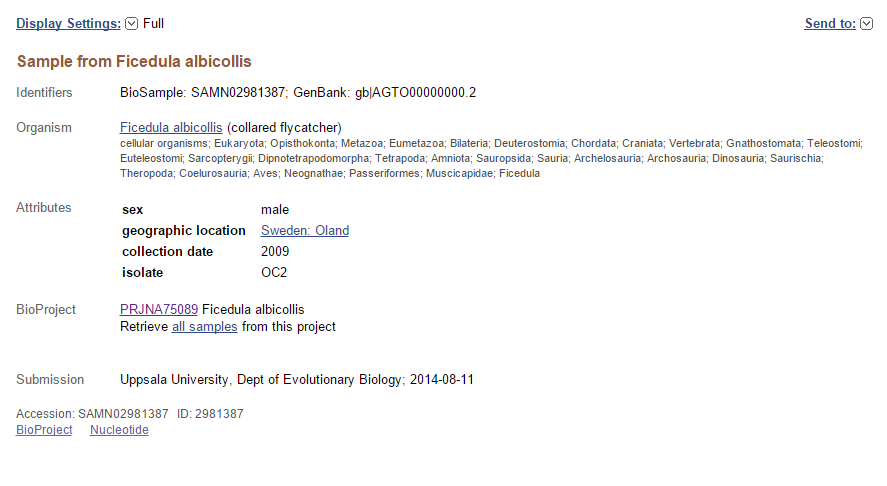
Рис.2 Описание BIOSAMPLE для сборки 2013 года
На этой странице сообщается об:
- Индентификаторе образца: в базе данных BIOSAMPLE (SAMN02981387) и GenBank (gb|AGTO00000000.2)
- Систематике организма Мухоловки-белошейки.
- Поле организма: мужской
- Географическом местонахождении вида: Эланд, Швеция
- Изоляте, к которму принадлежит организм: OC2
- Дате сбора: 2009
- Проектах по секвенированию, использующих данный образец: PRJNA75089 Ficedula albicollis
- Дате и месте завяления образца: Uppsala University, Dept of Evolutionary Biology; 2014-08-11
- Id образца: 2981387
Описание проекта по секвенированию (см. рис.3)
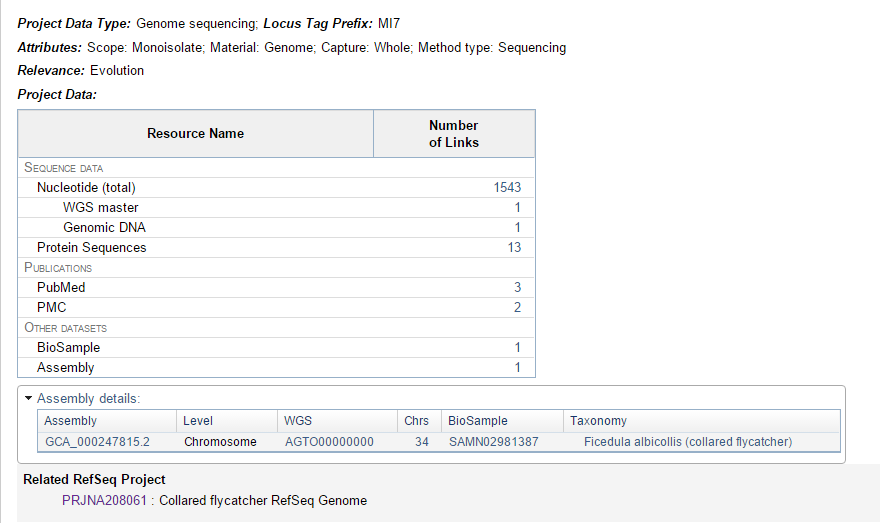
Рис.3 Описание BIOPROJECT для сборки 2013 года
Сборка 2013 года (GCA_000247815.2) содержит:
- 30,843 контигов
- 21,836 скэффолдов
- N50 (для контигов) - 410,964
- L50 (для контигов) - 771
- N50 (скэффолдов) - 6,542,656
- L50 (скэффолдов) - 48
Таблица контигов представлена здесь . Длина самого большого составляет 3298586 нуклеотидов (AC:AGTO02003581, name: contig02324),самого короткого (их несколько, выбран один) - 200 нуклеотидов(AC: AGTO02000301, name: contig02219)
Последовательность контига (contig04308) длиной 1,549,489 представлена здесь .
При быстром просмотре последовательности был обнаружен фрагмент из многократно (удивительно много раз) повторяющегся цитозина.
Таблица митохондриальных генов мха Oxystegus tenuirostris

Рис.4 Изображение мха Oxystegus tenuirostris
Составив запрос "Oxystegus tenuirostris and mitochondrion" в базе данных Nucleotide, получила две находки, по-разному датировнные, но содержащие идентичные последовательности. Далее перешла по ссылке Genome и получила информацию о всем митохондриальном геноме мха (см. рис.5) из таблички видно, что в митохондриальный геном содержит 66 генов, 39 из которых кодируют белки, 3 - рибосомальную РНК, 24 - транспортную РНК

Рис.5 Информация о геноме Oxystegus tenuirostris
Отсортированные по началу в геноме гены проедставлены в таблице
Ключи для таблицы особенностей (Feature Table)
На сайте Ebi (European Bioinformatics Institute) были найдены описания ключей из Feature Table. Далее приведены десять ключей с пояснениями и примерами.
- misc_feature Область биологического интереса, которая не может быть описана никаким другим ключом; новая или редкая характеристика.
Пример: Mus musculus
misc_feature 85..333 /gene="Ddo" /gene_synonym="5330420D20Rik; 5730402C02Rik; AI467244" /inference="COORDINATES: ab initio prediction:ORF Finder" /note="primary ORF has stop codon >50 nucleotides from the terminal splice site; nonsense-mediated mRNA decay (NMD) candidate" - RBS Сайт связывания рибосомы. Включен в regulatory_class (т.е. указвается в ключе regulatory). Именно в этом ключе указывается особенности (promoter, enhancer, rbs , tata-box и др.)
Пример: Enterobacter aerogenes
RBS Loc: c4693-4690 Loc: c4704-4701
- STS (sequence tagged site) Короткие последовательности ДНК ( 200 - 500 пар оснований),
которые могут быть амплифицированы при необходимых условиях
(для известны праймеры, необходмо подобрать так же количество циклов, ph среды и др.) в присутствии геномной ДНК.
Используются как маркеры в диагностике генетических болезней и картировании генома. Более подробно с методом STS можно ознакомиться здесь .
Пример: Homo sapiens
STS 3810..3911 /gene="ROBO2" /gene_synonym="SAX3" /standard_name="ROBO2" /db_xref="UniSTS:279030" - TATA_signal ТАТА-бокс, или бокс Хогнесса — консервативный мотив ДНК (цис-регуляторный элемент) эукариот, имеющий последовательность 5'-TATAAA-3'.
ТАТА-бокс располагается в промоторной области генов у архей и эукариот примерно на 30 нуклеотидов выше сайта начала транскрипции.
Пример: Faba bean necrotic yellows virus
regulatory 246..251 /regulatory_class="TATA_box" /gene="U4" /locus_tag="HZ18_sU4gp1" - N_region Дополнительные нуклеотиды, вставленные при пересттройке сегментов иммуноглобулинов.
Пример: synthetic construct
N_region 1..1008 /product="mature UshA domain" - LTR(long terminal repeat) Последовательность, которая повторяется на обоих концах последовательности. Обычно наблюдается у ретровирусов.
Пример: Pectobacterium phage PPWS1
LTR 1..127
- Precursor_RNA Любые виды РНК, которая еще не является зрелым продуктом РНК (т.е та, что содержит некодирующие участки);
может включать в себя ncRNA, рРНК, тРНК, 5 'нетранслируемой области (5'UTR),
кодирующие последовательности (CDS, экзон), промежуточные последовательности (интрон) и 3' нетранслируемой области (3'UTR).
Пример: Human herpesvirus 4 (Epstein-Barr virus)
precursor_RNA join(137963..138091,149198..149329,149540..149694, 149854..149965,154884..156354,156463..156545, 158242..160148) /gene="RPMS1" /locus_tag="HHV4_RPMS1" /product="microRNA BART-1" - mobile_element Участок генома, содержащий подвижные элементы.
Пример: Salmonella enterica subsp. enterica serovar Infantis
mobile_element 22386..23442 /mobile_element_type="insertion sequence" - sig_peptide Кодирующая последовательность сигнального пептида; кодирующая последовательность для N-концевого домена белка;
Пример: Myceliophthora thermophila
sig_peptide 1..51
- transit_peptide Последовательность, кодирующая пептид, который переносит другие белки в различные органеллы.
Пример: synthetic construct
transit_peptide 1..375 /note="Optimized transit peptide to chloroplasts"
Использование Blast для определения принадлежности последовательности к гену и таксономии организма.
В 6 практикуме была обработана хроматограмма и получена следующая последовательность. Чтобы определить, что закодировано в этой последовательности, был запущен Blastn c max количеством выровненных последовательностей равным 20000.
Определение принадлежности к гену.
Программа представила 20000 находок с наихудшим e-value = 1e-105. Все находки кодируют белок cytochrome oxidase subunit 1 (COI) (см. рис.6).
Рис.6 Структура белка cytochrome oxidase subunit 1
Этот белок кодируется митохондриальной ДНК. Является финальным ферментом электронной транспортной цепи в митохондриальном окислительном фосфолирировании. На этом основании можно быть уверенным, что в последовательности закодирован именно белок COI. При этом стоит заметить, что длина выбранной последовательности состовляет 695 нуклеотидов, а длина выравнивания - 662. Первые 33 нуклеотида не выравниваются. Предположение: некодирующая часть гена.
Определение таксономии.
Самые лучшие находки представлены на рис.7. Что было замечено:
- У первых двух находок (соответсвующих одному виду Polycirrus medusa) показатели довольно разительно отличаются от последующих.
- При просмотре первых трех выравниваний (ссылки на них приведены ниже) видно, что последовательности идентичны на 100 % (но по длине выравнивания различается на 2 нуклеотида), выравнивание же с последовательностью из организма Polycirrus sp. BOLD:AAI2761 содержит множество мест, где T замещен на С, А на G (еще есть единичные замены A на T и на С). Слово замена здесь употреблено только для описания различий. Ни в коем образе не предназначено для описания эволюционной истории. Уровень сродсвта (число замен на 100 н.п. составляет 93 %)
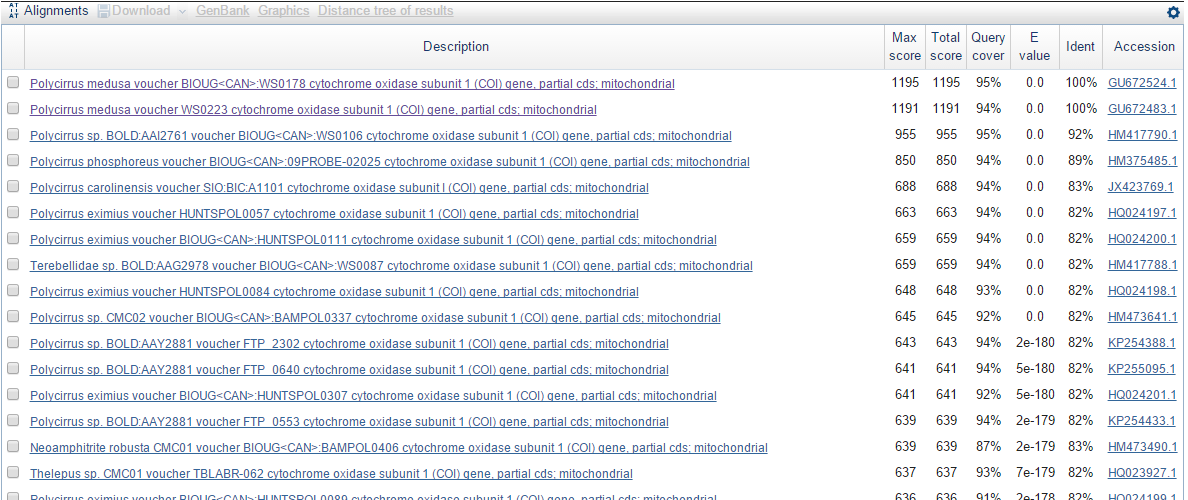
Рис.7 Лучшие находки, предложенные Blastn
Эти наблюдения делают возможным, по моему мнению, определить таксономию орагнизма с данной последовательностью с точностью до вида.(см.рис.8)
Этот эукариотический организм относится к полищетинковым червям. Полная систематика: Eukaryota; Metazoa; Lophotrochozoa; Annelida; Polychaeta; Scolecida; Terebellida; Terebellidae; Polycirrus, Polycirrus medusa.
Рис.8 Изображение Polycirrus medusa
Выравнивание выбранной последовательности с первой, со второй и с третьей находками
© 2014 Макарова Надежда