Выравнивание последовательностей
Практикум 10: Анализ множественных выравниваний
В данной работе обрабатывается множественное выравнивание, полученное в результате предыдущего
практикума.
Вертикальные блоки
Вертикальный блок определяется как участок множественного выравнивания, обладающий следующими свойствами:
Не содержит гэпов;
Длина - не менее 4 колонок;
Первая и последняя колонки - абсолютно консервативны или абсолютно функционально консервативны;
Процент абсолютно консервативных колонок больше 35%;
Не содержит более 10 колонок, не являющихся абсолютно консервативными
или абсолютно функционально консервативными, подряд;
Не может быть расширен без нарушения предыдущих свойств.
Launch in Jalview {"seqs":[{"name":"tr|F6GDD7|F6GDD7_LACS5/1-113","start":1,"end":113,"id":"1522087521","seq":"MKKVEAIIRKSKFSVVQEALQSVGVNFFTHWDVISIDNENQDVL------DLISSEANKENSKKCYL--SIIVNDDFKA------VTIKAILESA----STGAIGDGRIF---ISTIDEAYKIRTKEKGKKTLR----","order":1},{"name":"tr|B8J2I4|B8J2I4_DESDA/1-130","start":1,"end":130,"id":"1538941721","seq":"-MSSYTLPEDRSYTDEHIWLREDAQGLT-----VGISDFAQAQLEEVVYVDMPAVGSAVEAGKEFGSVESMKSVSALFSPVSGVVEAVNTDLENKPDLVNSDCYGQGWILRLRREGDNAV-PLLSAEEYRTHLKSQA-","order":2},{"name":"tr|I3R784|I3R784_HALMT/1-126","start":1,"end":126,"id":"257642587","seq":"---MFEIPDDRKYLESHEWTTTEGDTVR-----IGISDFAQDELGDVVFVELPTEGDELDAGDEFGVVESIKAVSDLYAPISGTVTAVNEALFDAPELVNDDPYGDGWMFEMEPSDTDEFDDLLSPEEYREQTE----","order":3},{"name":"tr|A0A0C5WMF1|A0A0C5WMF1_9GAMM/1-131","start":1,"end":131,"id":"352621805","seq":"---MSYIPSELKFTNTHEWVRHEGDGIFT----VGITDHAQSLLGDMVFVDLPEIDAATEAGEDCAVAESVKAASDIYAPLSGFVIAINEDLEGSPELVNSDPYGDGWLFQLKVEDDGEFADLLDADSYSDLVDEDSD","order":4},{"name":"tr|D8MWG5|D8MWG5_ERWBE/1-128","start":1,"end":128,"id":"102609426","seq":"---MSNVPNELKYRDSHEWVRKEADGTYT----VGITEHAQELLGDMVFVDLPDVGNTYSQGDDCAVAESVKAASDIYAPISGEIIEVNSALDGEPELVNSAPYAEGWLFKIKASNESELDSLLDADAYKGSIDE---","order":5}],"appSettings":{"globalColorScheme":"Clustal","webStartUrl":"http://www.jalview.org/services/launchApp","application":"Jalview","showSeqFeatures":"false","version":"2.9.0b2","hiddenCols":"0-2;133-137"},"seqGroups":[],"alignAnnotation":[{"annotationSettings":{"scaleColLabel":false,"visible":true,"hasIcon":false,"belowAlignment":true,"showAllColLabels":false,"centreColLabels":false},"score":0,"graphType":0,"annotations":[{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"colour":"000000","description":"","displayCharacter":"B","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"B","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"B","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"B","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"B","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"B","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"B","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"B","value":0,"secondaryStructure":" "},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"colour":"000000","description":"","displayCharacter":"B","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"B","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"B","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"B","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"B","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"B","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"B","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"B","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"B","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"B","value":0,"secondaryStructure":" "},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"colour":"000000","description":"","displayCharacter":"B","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"B","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"B","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"B","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"B","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"B","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"B","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"B","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"B","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"B","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"B","value":0,"secondaryStructure":" "},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"}],"calcId":"","label":"Вертикальные блоки"}],"svid":"1.0","seqFeatures":[]}
tr|F6GDD7|F6GDD7_LACS5/1-113 tr|B8J2I4|B8J2I4_DESDA/1-130 tr|I3R784|I3R784_HALMT/1-126 tr|A0A0C5WMF1|A0A0C5WMF1_9GAMM/1-131 tr|D8MWG5|D8MWG5_ERWBE/1-128 Вертикальные блоки Conservation Quality Consensus
13 23 33 43 53 63 73 83 93 103 113 123 V E A I I R K S K F S V V Q E A L Q S V G V N F F T H W D V I S I D N E N Q D V L - - - - - - D L I S S E A N K E N S K K C Y L - - S I I V N D D F K A - - - - - - V T I K A I L E S A - - - - S T G A I G D G R I F - - - I S T I D E A Y K I R T K E K G K K T L S Y T L P E D R S Y T D E H I W L R E D A Q G L T - - - - - V G I S D F A Q A Q L E E V V Y V D M P A V G S A V E A G K E F G S V E S M K S V S A L F S P V S G V V E A V N T D L E N K P D L V N S D C Y G Q G W I L R L R R E G D N A V - P L L S A E E Y R T H L M F E I P D D R K Y L E S H E W T T T E G D T V R - - - - - I G I S D F A Q D E L G D V V F V E L P T E G D E L D A G D E F G V V E S I K A V S D L Y A P I S G T V T A V N E A L F D A P E L V N D D P Y G D G W M F E M E P S D T D E F D D L L S P E E Y R E Q T M S Y I P S E L K F T N T H E W V R H E G D G I F T - - - - V G I T D H A Q S L L G D M V F V D L P E I D A A T E A G E D C A V A E S V K A A S D I Y A P L S G F V I A I N E D L E G S P E L V N S D P Y G D G W L F Q L K V E D D G E F A D L L D A D S Y S D L V M S N V P N E L K Y R D S H E W V R K E A D G T Y T - - - - V G I T E H A Q E L L G D M V F V D L P D V G N T Y S Q G D D C A V A E S V K A A S D I Y A P I S G E I I E V N S A L D G E P E L V N S A P Y A E G W L F K I K A S N E S E L D S L L D A D A Y K G S I B B B B B B B B B B B B B B B B B B B B B B B B B B B B B 5 3 3 9 6 5 6 3 5 9 3 4 3 6 5 6 6 7 3 4 + 4 7 5 4 - - - - - 9 8 * 7 7 4 7 * 4 4 * 0 3 1 3 2 3 9 9 6 4 3 4 5 4 3 6 6 8 6 6 8 6 5 2 3 * 8 6 6 6 7 5 8 6 8 2 2 3 3 0 2 4 4 9 6 4 3 * 4 5 3 2 3 2 3 9 7 5 7 7 + 7 * 6 9 8 1 1 2 2 6 5 4 5 4 6 0 2 + 5 7 3 9 3 6 5 3 2 6 M S + I P + + + K Y T D S H E W + R + E G D G + F T H W D V V G I + D + A Q D L L G D + V F V D L P + V G A A + E A G + + C + V + E S + K A + S D + Y A P I S G + V I A V N E + L E G A P E L V N S D P Y G D G W + F + L K + S D D D E F D D L L + A E E Y + + + L
скрипт
Проект
JalView . То же в формате fasta .
Один блок из части последовательностей
Мы включили в блок все последовательности, кроме D8MWG5_ERWBE.
Символом H выделены участки, на которых в пределах группы все остатки с очень высокой вероятностью гомологичны
ввиду абсолютной консервативности колонок.
скрипт
Launch in Jalview {"seqs":[{"name":"tr|F6GDD7|F6GDD7_LACS5/1-113","start":1,"end":113,"id":"504844587","seq":"MKKVEAIIRKSKFSVVQEALQSVGVNFFTHWDVISIDNENQDVL------DLISSEANKENSKKCYL--SIIVNDDFKA------VTIKAILESA----STGAIGDGRIF---ISTIDEAYKIRTKEKGKKTLR----","order":1},{"name":"tr|B8J2I4|B8J2I4_DESDA/1-130","start":1,"end":130,"id":"1383750059","seq":"-MSSYTLPEDRSYTDEHIWLREDAQGLT-----VGISDFAQAQLEEVVYVDMPAVGSAVEAGKEFGSVESMKSVSALFSPVSGVVEAVNTDLENKPDLVNSDCYGQGWILRLRREGDNAV-PLLSAEEYRTHLKSQA-","order":2},{"name":"tr|I3R784|I3R784_HALMT/1-126","start":1,"end":126,"id":"1794487028","seq":"---MFEIPDDRKYLESHEWTTTEGDTVR-----IGISDFAQDELGDVVFVELPTEGDELDAGDEFGVVESIKAVSDLYAPISGTVTAVNEALFDAPELVNDDPYGDGWMFEMEPSDTDEFDDLLSPEEYREQTE----","order":3},{"name":"tr|A0A0C5WMF1|A0A0C5WMF1_9GAMM/1-131","start":1,"end":131,"id":"708753620","seq":"---MSYIPSELKFTNTHEWVRHEGDGIFT----VGITDHAQSLLGDMVFVDLPEIDAATEAGEDCAVAESVKAASDIYAPLSGFVIAINEDLEGSPELVNSDPYGDGWLFQLKVEDDGEFADLLDADSYSDLVDEDSD","order":4},{"name":"tr|D8MWG5|D8MWG5_ERWBE/1-128","start":1,"end":128,"id":"243542705","seq":"---MSNVPNELKYRDSHEWVRKEADGTYT----VGITEHAQELLGDMVFVDLPDVGNTYSQGDDCAVAESVKAASDIYAPISGEIIEVNSALDGEPELVNSAPYAEGWLFKIKASNESELDSLLDADAYKGSIDE---","order":5}],"appSettings":{"globalColorScheme":"Blosum62","webStartUrl":"http://www.jalview.org/services/launchApp","application":"Jalview","showSeqFeatures":"false","version":"2.9.0b2","hiddenCols":"0-2;28-32;133-137"},"seqGroups":[{"displayText":true,"groupName":"JGroup:160479216","startRes":0,"sequenceRefs":["1383750059","1794487028"],"endRes":137,"colourText":false,"showNonconserved":false,"colourScheme":"Blosum62","displayBoxes":true}],"alignAnnotation":[{"annotationSettings":{"scaleColLabel":false,"visible":true,"hasIcon":false,"belowAlignment":true,"showAllColLabels":false,"centreColLabels":false},"score":0,"graphType":0,"annotations":[{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"description":"","displayCharacter":"H","value":0,"secondaryStructure":" "},{"description":"","displayCharacter":"H","value":0,"secondaryStructure":" "},{"description":"","displayCharacter":"H","value":0,"secondaryStructure":" "},{"description":"","displayCharacter":"H","value":0,"secondaryStructure":" "},{"description":"","displayCharacter":"H","value":0,"secondaryStructure":" "},{"description":"","displayCharacter":"H","value":0,"secondaryStructure":" "},{"description":"","displayCharacter":"H","value":0,"secondaryStructure":" "},{"description":"","displayCharacter":"H","value":0,"secondaryStructure":" "},{"description":"","displayCharacter":"H","value":0,"secondaryStructure":" "},{"description":"","displayCharacter":"H","value":0,"secondaryStructure":" "},{"description":"","displayCharacter":"H","value":0,"secondaryStructure":" "},{"description":"","displayCharacter":"H","value":0,"secondaryStructure":" "},{"description":"","displayCharacter":"H","value":0,"secondaryStructure":" "},{"description":"","displayCharacter":"H","value":0,"secondaryStructure":" "},{"description":"","displayCharacter":"H","value":0,"secondaryStructure":" "},{"description":"","displayCharacter":"H","value":0,"secondaryStructure":" "},{"description":"","displayCharacter":"H","value":0,"secondaryStructure":" "},{"description":"","displayCharacter":"H","value":0,"secondaryStructure":" "},{"description":"","displayCharacter":"H","value":0,"secondaryStructure":" "},{"description":"","displayCharacter":"H","value":0,"secondaryStructure":" "},{"description":"","displayCharacter":"H","value":0,"secondaryStructure":" "},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"description":"","displayCharacter":"H","value":0,"secondaryStructure":" "},{"description":"","displayCharacter":"H","value":0,"secondaryStructure":" "},{"description":"","displayCharacter":"H","value":0,"secondaryStructure":" "},{"description":"","displayCharacter":"H","value":0,"secondaryStructure":" "},{"description":"","displayCharacter":"H","value":0,"secondaryStructure":" "},{"description":"","displayCharacter":"H","value":0,"secondaryStructure":" "},{"description":"","displayCharacter":"H","value":0,"secondaryStructure":" "},{"description":"","displayCharacter":"H","value":0,"secondaryStructure":" "},{"description":"","displayCharacter":"H","value":0,"secondaryStructure":" "},{"description":"","displayCharacter":"H","value":0,"secondaryStructure":" "},{"description":"","displayCharacter":"H","value":0,"secondaryStructure":" "},{"description":"","displayCharacter":"H","value":0,"secondaryStructure":" "},{"description":"","displayCharacter":"H","value":0,"secondaryStructure":" "},{"description":"","displayCharacter":"H","value":0,"secondaryStructure":" "},{"description":"","displayCharacter":"H","value":0,"secondaryStructure":" "},{"description":"","displayCharacter":"H","value":0,"secondaryStructure":" "},{"description":"","displayCharacter":"H","value":0,"secondaryStructure":" "},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"description":"","displayCharacter":"H","value":0,"secondaryStructure":" "},{"description":"","displayCharacter":"H","value":0,"secondaryStructure":" "},{"description":"","displayCharacter":"H","value":0,"secondaryStructure":" "},{"description":"","displayCharacter":"H","value":0,"secondaryStructure":" "},{"description":"","displayCharacter":"H","value":0,"secondaryStructure":" "},{"description":"","displayCharacter":"H","value":0,"secondaryStructure":" "},{"description":"","displayCharacter":"H","value":0,"secondaryStructure":" "},{"description":"","displayCharacter":"H","value":0,"secondaryStructure":" "},{"description":"","displayCharacter":"H","value":0,"secondaryStructure":" "},{"description":"","displayCharacter":"H","value":0,"secondaryStructure":" "},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"description":"","displayCharacter":"H","value":0,"secondaryStructure":" "},{"description":"","displayCharacter":"H","value":0,"secondaryStructure":" "},{"description":"","displayCharacter":"H","value":0,"secondaryStructure":" "},{"description":"","displayCharacter":"H","value":0,"secondaryStructure":" "},{"description":"","displayCharacter":"H","value":0,"secondaryStructure":" "},{"description":"","displayCharacter":"H","value":0,"secondaryStructure":" "},{"description":"","displayCharacter":"H","value":0,"secondaryStructure":" "},{"description":"","displayCharacter":"H","value":0,"secondaryStructure":" "},{"description":"","displayCharacter":"H","value":0,"secondaryStructure":" "},{"description":"","displayCharacter":"H","value":0,"secondaryStructure":" "},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"}],"calcId":"","label":"segment vertical"},{"annotationSettings":{"scaleColLabel":false,"visible":true,"hasIcon":false,"belowAlignment":true,"showAllColLabels":false,"centreColLabels":false},"score":0,"graphType":0,"annotations":[{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"colour":"000000","description":"","displayCharacter":"X","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"X","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"X","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"X","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"X","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"X","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"X","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"X","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"X","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"X","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"X","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"X","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"X","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"X","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"X","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"X","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"X","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"X","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"X","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"X","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"X","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"X","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"X","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"X","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"X","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"X","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"X","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"X","value":0,"secondaryStructure":" "},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"}],"calcId":"","label":"gap's"},{"annotationSettings":{"scaleColLabel":false,"visible":true,"hasIcon":false,"belowAlignment":true,"showAllColLabels":false,"centreColLabels":false},"score":0,"graphType":0,"annotations":[{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"colour":"000000","description":"","displayCharacter":"B","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"B","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"B","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"B","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"B","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"B","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"B","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"B","value":0,"secondaryStructure":" "},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"colour":"000000","description":"","displayCharacter":"B","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"B","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"B","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"B","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"B","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"B","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"B","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"B","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"B","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"B","value":0,"secondaryStructure":" "},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"colour":"000000","description":"","displayCharacter":"B","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"B","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"B","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"B","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"B","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"B","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"B","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"B","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"B","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"B","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"B","value":0,"secondaryStructure":" "},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"}],"calcId":"","label":"Вертикальные блоки"}],"svid":"1.0","seqFeatures":[]}
tr|F6GDD7|F6GDD7_LACS5/1-113 tr|B8J2I4|B8J2I4_DESDA/1-130 tr|I3R784|I3R784_HALMT/1-126 tr|A0A0C5WMF1|A0A0C5WMF1_9GAMM/1-131 tr|D8MWG5|D8MWG5_ERWBE/1-128 segment vertical gap's Вертикальные блоки Conservation Quality Consensus
13 23 38 48 58 68 78 88 98 108 118 128 V E A I I R K S K F S V V Q E A L Q S V G V N F F S Y T L P E D R S Y T D E H I W L R E D A Q G L T M F E I P D D R K Y L E S H E W T T T E G D T V R M S Y I P S E L K F T N T H E W V R H E G D G I F M S N V P N E L K Y R D S H E W V R K E A D G T Y I S I D N E N Q D V L - - - - - - D L I S S E A N K E N S K K C Y L - - S I I V N D D F K A - - - - - - V T I K A I L E S A - - - - S T G A I G D G R I F - - - I S T I D E A Y K I R T K E K G K K T L V G I S D F A Q A Q L E E V V Y V D M P A V G S A V E A G K E F G S V E S M K S V S A L F S P V S G V V E A V N T D L E N K P D L V N S D C Y G Q G W I L R L R R E G D N A V - P L L S A E E Y R T H L I G I S D F A Q D E L G D V V F V E L P T E G D E L D A G D E F G V V E S I K A V S D L Y A P I S G T V T A V N E A L F D A P E L V N D D P Y G D G W M F E M E P S D T D E F D D L L S P E E Y R E Q T V G I T D H A Q S L L G D M V F V D L P E I D A A T E A G E D C A V A E S V K A A S D I Y A P L S G F V I A I N E D L E G S P E L V N S D P Y G D G W L F Q L K V E D D G E F A D L L D A D S Y S D L V V G I T E H A Q E L L G D M V F V D L P D V G N T Y S Q G D D C A V A E S V K A A S D I Y A P I S G E I I E V N S A L D G E P E L V N S A P Y A E G W L F K I K A S N E S E L D S L L D A D A Y K G S I H H H H H H H H H H H H H H H H H H H H H H H H H H H H H H H H H H H H H H H H H H H H H H H H H H H H H H H H H H X X X X X X X X X X X X X X X X X X X X X X X X X X X X B B B B B B B B B B B B B B B B B B B B B B B B B B B B B 5 3 3 9 6 5 6 3 5 9 3 4 3 6 5 6 6 7 3 4 + 4 7 5 4 9 8 * 7 7 4 7 * 4 4 * 0 3 1 3 2 3 9 9 6 4 3 4 5 4 3 6 6 8 6 6 8 6 5 2 3 * 8 6 6 6 7 5 8 6 8 2 2 3 3 0 2 4 4 9 6 4 3 * 4 5 3 2 3 2 3 9 7 5 7 7 + 7 * 6 9 8 1 1 2 2 6 5 4 5 4 6 0 2 + 5 7 3 9 3 6 5 3 2 6 M S + I P + + + K Y T D S H E W + R + E G D G + F V G I + D + A Q D L L G D + V F V D L P + V G A A + E A G + + C + V + E S + K A + S D + Y A P I S G + V I A V N E + L E G A P E L V N S D P Y G D G W + F + L K + S D D D E F D D L L + A E E Y + + + L
Проект
JalView . То же в формате fasta .
Число и процент позиций с гэпами на самом длинном участке за пределами блоков
Самый длинный участок выравнивания, не входящий в состав блоков, - это блок с координатами 42-69.
Этот участок обозначен символом X.
Таблица 1. Гэпы в блоке 42-69
Позиции
Число
Процент
С гэпами
8
29%
Всего
28
100%
Число и процент консервативных позиций
Функционально абсолютно консервативными позициями считались позиции со следующими группами аминокислот:
KR
ST
LIVM
FYW
DN
EQ
P
G
A
H
С Параметры были подсчитаны для вертикального блока с координатами 131-138, в нем 8 колонок.
Таблица 2. Консервативные позиции в блоке 131-138
Позиции
Число
Процент
Абсолютно консервативные
2
25%
Абсолютно функционально консервативные
3
37.5%
Всего
8
100%
Консенсусная последовательность и LOGO одного блока
Launch in Jalview {"seqs":[{"name":"tr|F6GDD7|F6GDD7_LACS5/1-113","start":1,"end":113,"id":"113071330","seq":"MKKVEAIIRKSKFSVVQEALQSVGVNFFTHWDVISIDNENQDVL------DLISSEANKENSKKCYL--SIIVNDDFKA------VTIKAILESA----STGAIGDGRIF---ISTIDEAYKIRTKEKGKKTLR----","order":1},{"name":"tr|B8J2I4|B8J2I4_DESDA/1-130","start":1,"end":130,"id":"528671120","seq":"-MSSYTLPEDRSYTDEHIWLREDAQGLT-----VGISDFAQAQLEEVVYVDMPAVGSAVEAGKEFGSVESMKSVSALFSPVSGVVEAVNTDLENKPDLVNSDCYGQGWILRLRREGDNAV-PLLSAEEYRTHLKSQA-","order":2},{"name":"tr|I3R784|I3R784_HALMT/1-126","start":1,"end":126,"id":"1046694407","seq":"---MFEIPDDRKYLESHEWTTTEGDTVR-----IGISDFAQDELGDVVFVELPTEGDELDAGDEFGVVESIKAVSDLYAPISGTVTAVNEALFDAPELVNDDPYGDGWMFEMEPSDTDEFDDLLSPEEYREQTE----","order":3},{"name":"tr|A0A0C5WMF1|A0A0C5WMF1_9GAMM/1-131","start":1,"end":131,"id":"2023505632","seq":"---MSYIPSELKFTNTHEWVRHEGDGIFT----VGITDHAQSLLGDMVFVDLPEIDAATEAGEDCAVAESVKAASDIYAPLSGFVIAINEDLEGSPELVNSDPYGDGWLFQLKVEDDGEFADLLDADSYSDLVDEDSD","order":4},{"name":"tr|D8MWG5|D8MWG5_ERWBE/1-128","start":1,"end":128,"id":"350912341","seq":"---MSNVPNELKYRDSHEWVRKEADGTYT----VGITEHAQELLGDMVFVDLPDVGNTYSQGDDCAVAESVKAASDIYAPISGEIIEVNSALDGEPELVNSAPYAEGWLFKIKASNESELDSLLDADAYKGSIDE---","order":5}],"appSettings":{"globalColorScheme":"Blosum62","webStartUrl":"http://www.jalview.org/services/launchApp","application":"Jalview","showSeqFeatures":"false","version":"2.9.0b2","hiddenCols":"0-32;41-133;134-137"},"seqGroups":[],"alignAnnotation":[{"annotationSettings":{"scaleColLabel":false,"visible":true,"hasIcon":false,"belowAlignment":true,"showAllColLabels":false,"centreColLabels":false},"score":0,"graphType":0,"annotations":[{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"colour":"000000","description":"","displayCharacter":"X","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"X","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"X","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"X","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"X","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"X","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"X","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"X","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"X","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"X","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"X","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"X","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"X","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"X","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"X","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"X","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"X","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"X","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"X","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"X","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"X","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"X","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"X","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"X","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"X","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"X","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"X","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"X","value":0,"secondaryStructure":" "},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"}],"calcId":"","label":"гэпы"},{"annotationSettings":{"scaleColLabel":false,"visible":true,"hasIcon":false,"belowAlignment":true,"showAllColLabels":false,"centreColLabels":false},"score":0,"graphType":0,"annotations":[{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"colour":"000000","description":"","displayCharacter":"B","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"B","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"B","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"B","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"B","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"B","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"B","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"B","value":0,"secondaryStructure":" "},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"colour":"000000","description":"","displayCharacter":"B","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"B","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"B","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"B","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"B","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"B","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"B","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"B","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"B","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"B","value":0,"secondaryStructure":" "},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"colour":"000000","description":"","displayCharacter":"B","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"B","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"B","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"B","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"B","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"B","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"B","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"B","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"B","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"B","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"B","value":0,"secondaryStructure":" "},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"}],"calcId":"","label":"Вертикальные блоки"}],"svid":"1.0","seqFeatures":[]}
tr|F6GDD7|F6GDD7_LACS5/1-113 tr|B8J2I4|B8J2I4_DESDA/1-130 tr|I3R784|I3R784_HALMT/1-126 tr|A0A0C5WMF1|A0A0C5WMF1_9GAMM/1-131 tr|D8MWG5|D8MWG5_ERWBE/1-128 гэпы Вертикальные блоки Conservation Quality Consensus
I S I D N E N Q V G I S D F A Q I G I S D F A Q V G I T D H A Q V G I T E H A Q B B B B B B B B 9 8 * 7 7 4 7 * V G I + D + A Q V I G S I S T D D E N F H E A N Q
В проекте
JalView отдельно записан блок 131-138.
Тот же блок в формате fasta .
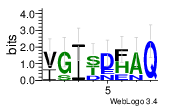
Консенсусная последовательность высчитывается исходя из встречаемости остатков (нуклеотидных или аминокислотных).
Consensus/34-134 Percentage Identity Consensus
Чтобы сохранить консенсусную последовательность и LOGO блока 131-138,
мы воспользовались программой JalView и сервисом http://weblogo.threeplusone.com/ ,
который принимает на вход совокупность выровненных последовательностей и
автоматически выдает искомое изображение в указанном разрешении.
По умолчанию аминокислотные последовательности окрашиваются по гидрофобности.
Гидрофобные аминокислоты выделяются черным, гидрофильные синим, а нейтральные зеленым.
Паттерн для блока 131-138: [IV]-[SG]-I-[DST]-[NDE]-[EFH]-[NA]-Q
Выравнивание с негомологичной последовательностью
Для данного задания была использована последовательность белка с идентификатором
Uniprot BLAN1_KLEPN, очевидно не гомологичную пяти последовательностям, исследованным ранее.
Было обнаружено некоторое количество "консервативных" колонок, но тем не менее качество выравнивания невысоко.
Launch in Jalview
{"seqs":[{"name":"tr|F6GDD7|F6GDD7_LACS5/4-112","start":4,"end":112,"id":"957988829","seq":"VEAI--IRKSKFSVVQEALQSVGVNFFTHWDVISID-NENQDVLDLIS----------------------------------------------------------------------------------SEANKENSKKCYLSIIVNDDFKAVTIKAILESA----STG-------------------------------AIGDGRIF---ISTIDEAYKIRTKEKGKKTL","order":1},{"name":"sp|C7C422|BLAN1_KLEPN/3-233","start":3,"end":233,"id":"171447044","seq":"LPNIMHPVAKLSTALAAALMLSGCMPGEIRPTIGQQMETGDQRFGDLVFRQLAPNVWQHTSYLDMPGFGAVASNGLIVRDGGRVLVVDTAWTDDQTAQILNWIKQEINLPVALAVVTHAHQDKMGGMDALHAAGIATYANALSNQLAPQEGMVAAQHSLTFAANGWVEPATAPNFGPLKVFYPGPGHTSDNITVGIDGTDIAFGGCLIKDSKAKSLGNLGDA-DTEHYAASA","order":2},{"name":"tr|B8J2I4|B8J2I4_DESDA/3-126","start":3,"end":126,"id":"431210359","seq":"SYTL--PEDRSYTDEHIWLREDAQGLT-----VGIS-DFAQAQLEEVV-------------YVDMPAVGSAVEAG-------------------------------------------------------KEFGSVESMKSVSALFSPVSGVVEAVNTDLENKPDLVNSD-------------------------------CYGQGWILRLR-REGDNAVPLLSAEEYRTHL","order":3},{"name":"tr|I3R784|I3R784_HALMT/1-125","start":1,"end":125,"id":"1033951948","seq":"MFEI--PDDRKYLESHEWTTTEGDTVR-----IGIS-DFAQDELGDVV-------------FVELPTEGDELDAG-------------------------------------------------------DEFGVVESIKAVSDLYAPISGTVTAVNEALFDAPELVNDD-------------------------------PYGDGWMFEMEPSDTDEFDDLLSPEEYREQT","order":4},{"name":"tr|A0A0C5WMF1|A0A0C5WMF1_9GAMM/1-126","start":1,"end":126,"id":"1236495858","seq":"MSYI--PSELKFTNTHEWVRHEGDGIFT----VGIT-DHAQSLLGDMV-------------FVDLPEIDAATEAG-------------------------------------------------------EDCAVAESVKAASDIYAPLSGFVIAINEDLEGSPELVNSD-------------------------------PYGDGWLFQLKVEDDGEFADLLDADSYSDLV","order":5},{"name":"tr|D8MWG5|D8MWG5_ERWBE/1-126","start":1,"end":126,"id":"56223731","seq":"MSNV--PNELKYRDSHEWVRKEADGTYT----VGIT-EHAQELLGDMV-------------FVDLPDVGNTYSQG-------------------------------------------------------DDCAVAESVKAASDIYAPISGEIIEVNSALDGEPELVNSA-------------------------------PYAEGWLFKIKASNESELDSLLDADAYKGSI","order":6}],"appSettings":{"globalColorScheme":"Blosum62","webStartUrl":"http://www.jalview.org/services/launchApp","application":"Jalview","showSeqFeatures":"false","version":"2.9.0b2"},"seqGroups":[],"alignAnnotation":[],"svid":"1.0","seqFeatures":[]}
tr|F6GDD7|F6GDD7_LACS5/4-112 sp|C7C422|BLAN1_KLEPN/3-233 tr|B8J2I4|B8J2I4_DESDA/3-126 tr|I3R784|I3R784_HALMT/1-125 tr|A0A0C5WMF1|A0A0C5WMF1_9GAMM/1-126 tr|D8MWG5|D8MWG5_ERWBE/1-126 Conservation Quality Consensus
10 20 30 40 50 60 70 80 90 100 110 120 130 140 150 160 170 180 190 200 210 220 230 V E A I - - I R K S K F S V V Q E A L Q S V G V N F F T H W D V I S I D - N E N Q D V L D L I S - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - S E A N K E N S K K C Y L S I I V N D D F K A V T I K A I L E S A - - - - S T G - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - A I G D G R I F - - - I S T I D E A Y K I R T K E K G K K T L L P N I M H P V A K L S T A L A A A L M L S G C M P G E I R P T I G Q Q M E T G D Q R F G D L V F R Q L A P N V W Q H T S Y L D M P G F G A V A S N G L I V R D G G R V L V V D T A W T D D Q T A Q I L N W I K Q E I N L P V A L A V V T H A H Q D K M G G M D A L H A A G I A T Y A N A L S N Q L A P Q E G M V A A Q H S L T F A A N G W V E P A T A P N F G P L K V F Y P G P G H T S D N I T V G I D G T D I A F G G C L I K D S K A K S L G N L G D A - D T E H Y A A S A S Y T L - - P E D R S Y T D E H I W L R E D A Q G L T - - - - - V G I S - D F A Q A Q L E E V V - - - - - - - - - - - - - Y V D M P A V G S A V E A G - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - K E F G S V E S M K S V S A L F S P V S G V V E A V N T D L E N K P D L V N S D - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - C Y G Q G W I L R L R - R E G D N A V P L L S A E E Y R T H L M F E I - - P D D R K Y L E S H E W T T T E G D T V R - - - - - I G I S - D F A Q D E L G D V V - - - - - - - - - - - - - F V E L P T E G D E L D A G - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - D E F G V V E S I K A V S D L Y A P I S G T V T A V N E A L F D A P E L V N D D - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - P Y G D G W M F E M E P S D T D E F D D L L S P E E Y R E Q T M S Y I - - P S E L K F T N T H E W V R H E G D G I F T - - - - V G I T - D H A Q S L L G D M V - - - - - - - - - - - - - F V D L P E I D A A T E A G - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - E D C A V A E S V K A A S D I Y A P L S G F V I A I N E D L E G S P E L V N S D - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - P Y G D G W L F Q L K V E D D G E F A D L L D A D S Y S D L V M S N V - - P N E L K Y R D S H E W V R K E A D G T Y T - - - - V G I T - E H A Q E L L G D M V - - - - - - - - - - - - - F V D L P D V G N T Y S Q G - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - D D C A V A E S V K A A S D I Y A P I S G E I I E V N S A L D G E P E L V N S A - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - P Y A E G W L F K I K A S N E S E L D S L L D A D A Y K G S I 5 2 3 9 - - 6 2 3 3 3 5 3 3 3 3 3 6 6 5 1 3 + 4 6 4 3 - - - - - 9 8 7 6 - 7 3 7 7 4 3 8 4 4 8 6 - - - - - - - - - - - - - 2 2 3 1 2 0 0 1 1 0 0 1 0 3 - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - 3 4 7 7 3 3 7 6 4 6 7 5 5 5 7 7 6 8 4 6 7 2 7 3 4 6 5 4 3 6 4 5 3 1 0 1 3 6 5 5 - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - 7 7 + 4 9 4 9 5 1 0 2 0 4 6 3 5 7 6 2 2 7 0 7 3 9 2 6 4 3 2 5 M S N I M H P + + + K Y T D S H E W L R + E G D G + F T + + + + + G I + M D + A Q D L L G D + V F R Q L A P N V W Q H T S F V D L P + V G A A + + A G L I V R D G G R V L V V D T A W T D D Q T A Q I L N W I K Q E I N L P V A L A V V T H A H Q D K M G G M D A L D E + G V A E S V K A + S D I Y A P I S G + V I A V N + + L E G A P E L V N S D T A P N F G P L K V F Y P G P G H T S D N I T V G I D G T D I P Y G D G W I F + L K A S D + D E + D D L L D A E E Y + + S L
Проект
JalView .
Таблица 3. Консервативные позиции при выравнивании с заведомо негомологичной последовательностью
Позиции
Число
Процент
Абсолютно консервативные
10
4.3%
Абсолютно функционально консервативные
38
16.3%
Всего
232
100%
Выравнивание заведомо неродственных белков
Для данного задания были использованы последовательности белков с идентификаторами
Uniprot ISCS_ECOLI, C8W8H7_ATOPD, Q8AAN6_BACTN, B4EAZ8_BURCJ, NANE_CLOPE и PHNS_DESVM.
Launch in Jalview {"seqs":[{"name":"UniProt/Swiss-Prot|P0A6B7|P39171|P76581|P76992|Q8XA86|ISCS_ECOLI/1-394","start":1,"end":394,"id":"676895792","seq":"MKLPIYLDYSATTP------------------------------VDPR-------VAEKM----MQFMTMDGTF------------GNPASRSHRFGWQAEEAVDIAR--------------------------------NQIAD---LVGA------------------------------------------------DPREIV--------------FT-------------------------------------SGATESDNLAIKGAANFYQKKGKHIITSKTEHKAVLDTCRQLEREGFEVTYLAPQRNGIIDLKEL---EAAMRDDTILVSI------------------------------------------------------------------------------------------------------------------------------------------------MHVNNEIGVVQDIAAIGEMCRARGIIYHVDATQSVGKLP-IDLSQLKVDLM-----------------------------------SFSGHKIYGPKGIGALYVRRKPRVRIEAQMHGGGH-------------ERGMRSGTLPVHQIV---GMGEAYRIAKEEMATEMERLRGLRNRLWNGIKDIEEVYLNGDLEHGAPNILNVSFNYVEGESLIMALKDLAVSSGSACTSASLEPSYV------------LRALGLNDELAHSSIRFSLGRFTT---------------------------------------------------------EEEIDYTIELVRKSIGR-LRD---LSPLWEMYKQGV----------","order":1},{"name":"tr|C8W8H7|C8W8H7_ATOPD/43-857","start":43,"end":857,"id":"892723461","seq":"MAVPIGLFAAFTVAHIAIRFLAPAADPAILPIVFVLSGIGITMVTRLSPNLAVNQTIWLFISVVVMIVVLAIIRNLDALADYKYSIGILGVILLLLPIVIGQDRYGSRLWISFGPFTFQPGEIAKIAITLFLAFYLALNREALSVSMRSVGPFRIPRFKMLLPLFVMWGISLIVVIFERDLGSALLFFVFFVIMLYVATGRASYVFVSVA-LLAIGGVILYHFFSHVQTRVNIWLDPFKDPSGDGFQIVQSLYSIADGGLAGTGIDKGMPT---------LIPVVESDFIFSAIAEEMGLFGGAAIITLFLLLTVRGLATAARAKSDSSAFAAAGLTSVLAFQTFLIIAGVTKLMPLTGVTLPFMSQGGSSLLSSFIIVALLLRAGDEGTGRETELEPSKKVLDSQRLEISSGGAHASSVLHGSHIRGGFDLQSEESGVLGRVALGKRLTNLVTVFTLFFTVLLGNLTFLMVIDAPRLQALPTNNHTIAKSAYVQRGAIITSDGVTLAESVKQDDGTYVRNYPHDGMASHTVGYISTQYGT----AGIESSMNETLTGHADHSDWRSALYSMAGINTTGSSVVLTINSQMQAVAEAALQGYSGSIVVMDPSTGAVLAKASSPSYTHAELGTII---ESGTGSQLVDRTTQALYSPGSSFKTVTLAAGIDTHKTTLDTTYSAPGTME----------------------IGGGTIHNYANEDMGTIPLREA--FA-----RSSNTALAQLGVALGADNLVSYARAFGYGTALGQDFSTTPSLMPNPAEMTTWELAWASCGLPVGEHASPAGPQTTVMQNAVIAAAIANGGVVMNPYIVDRV-LSPEGAVVSTTS-PKSLGQAVS","order":2},{"name":"tr|Q8AAN6|Q8AAN6_BACTN/24-427","start":24,"end":427,"id":"524594980","seq":"QRLKKTINIAANSPYYKEVFSKNGITGDSIQSLDDIRKIPFTTKSDMRANYPFGLVAGDMKRDGVRIHSSSGTT------------GNPTVIVH-----SQHDLDSWA--------------------------------NLVARCLYMVGI------------------------------------------------RKTDVFQNSSGYGMFTGGLGFQ-------------------------------------YGAERLGCLTVPAAAG----------NSKRQIKFISD---------FKTTALHAIPSYAIRLAEVFQEEGIDPRETTLKTL------------------------------------------------------------------------------------------------------------------------------------------------V---------------------------------IGAEPHTDEQRRKIERMLNVKAY----------------------------NSFGMTEMNGPG------------VAFECQEQNGMHFWEDCYLVEIIDPETGE-----PVPE-------GEIGELVLTTLDREMMPLIRYRTR------DLTRI-LPGKCPCGRTHL---RIDRIKGRS-----DDMFIIKGVNIFPMQVEKILVQFPELGSNYLITLETVNNQDEMI---VEVELSDLST---------------------------------------------------------DNYIE--LEKIRRDIIRQLKDEILVTPKVKLVKKGSLPQSEGKAVR","order":3},{"name":"tr|B4EAZ8|B4EAZ8_BURCJ/1-235","start":1,"end":235,"id":"1003586384","seq":"MTAPLE--------------------------------------------------------------------------------GQVAIVTG-----------GAR----------------GIG-------------RGIALTLAGAGA---------------------NILLADLLDDALD-----------ATAREVRALGRRA--------------------------------------------------AIAKVDVTQAA---------QVDAMVAQ----ALAD----------------------------------------------------------------------------------------------------------------------------------LGGLDILVNCAGVIS-------------------------------------------IHPVAE---------LTERDWDFVMNV-------------------------NAKGT---FLGCRA--------------------ALAHLKAQGRGRIINVAS-----------------------------IAGK--------------------------------------------------------------------------------------------------EGFPNLAHYSASKFAVVGFTNALAKELARDGVTVNAI-----------------------------------------CPGIV----------RTYMWDRLSDEWKTDGESVEQSWQRHQLTLIPQGR---AQT-PEDMGRLA-","order":4},{"name":"sp|Q8XNZ3|NANE_CLOPE/21-193","start":21,"end":193,"id":"1459892486","seq":"LHSSFIM-------------------------------------------------------------------------------GRMAI--------------AAK--------------------------------QGGAAAIRAQGVNDINEIKEVTKLPIIG-------IIKRNYDDSEIYIT--------PTMKEVDELLKTD-------------------------------------------------CEMIALDATKRKRPNGENVKDLVDAIHAK-GRLAMAD--------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------------ISTLEE--------------------------------------------------------GIEA-------------------------------------------------------------------------------------------------------------------------------------------------------------------------EKLG---------FDCVSTTLSGYTPYSKQSNSVDFELLEELVKTVKIPVI---------------------------CEGRI-------------------------------------------------NT-PEELKKAL-","order":5},{"name":"sp|P21853|PHNS_DESVM/1-299","start":1,"end":299,"id":"1302585130","seq":"MKISIGL-------------------------------------------------------------------------------GKEGV----------EERLAER----------------GVSRRDFLKFC-----TAIAVTM-GMGP-----------------------AFAPEVARALM-----------GPRRPSVVYLHNAECTGCSESVLRAFEPYIDTLI---LDTL---SLDYHETI----------MAAAG-DAAEAA---------LEQAVNSPHGFIAVVE-------------------GGIPTAA------------------------------------------------------------------------------------------------------------------NGIYGKVA----------------------------------------NHTMLD---------ICSRILPKAQAV-------------------------IAYGTCATFGGVQA----------------------AKPNPTGAK---GVNDALKHLGVKAIN------------------IAGCPPNPYNLVGTIVYYLKNKAAPEL-----DSLNRPTMFF---------------------------------------------------GQTVH----EQCPRLPHFDAGEFA-----PSFESEEARKG----------------------------------------WCLYELGCKGPV------------TMNN------------------------CPKIKFNQTNW-PVDAGHPC-","order":6}],"appSettings":{"globalColorScheme":"Blosum62","webStartUrl":"http://www.jalview.org/services/launchApp","application":"Jalview","showSeqFeatures":"false","version":"2.9.0b2","hiddenCols":"0-85;296-852;853-862"},"seqGroups":[],"alignAnnotation":[{"annotationSettings":{"scaleColLabel":false,"visible":true,"hasIcon":false,"belowAlignment":true,"showAllColLabels":false,"centreColLabels":false},"score":0,"graphType":0,"annotations":[{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"colour":"000000","description":"","displayCharacter":"A","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"A","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"A","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"A","value":0,"secondaryStructure":" "},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"colour":"000000","description":"","displayCharacter":"A","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"A","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"A","value":0,"secondaryStructure":" "},{"colour":"000000","description":"","displayCharacter":"A","value":0,"secondaryStructure":" "},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"},{"value":0,"secondaryStructure":"\u0000"}],"calcId":"","label":"blocks"}],"svid":"1.0","seqFeatures":[]}
UniProt/Swiss-Prot|P0A6B7|P39171|P76581|P76992|Q8XA86|ISCS_ECOLI/1-394 tr|C8W8H7|C8W8H7_ATOPD/43-857 tr|Q8AAN6|Q8AAN6_BACTN/24-427 tr|B4EAZ8|B4EAZ8_BURCJ/1-235 sp|Q8XNZ3|NANE_CLOPE/21-193 sp|P21853|PHNS_DESVM/1-299 blocks Conservation Quality Consensus
96 106 116 126 136 146 156 166 176 186 196 206 216 226 236 246 256 266 276 286 G N P A S R S H R F G W Q A E E A V D I A R - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - N Q I A D - - - L V G A - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - D P R E I V - - - - - - - - - - - - - - F T - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - S G A T E S D N L A I K G A A N F Y Q K K G K H I I T S K T E H K A V L D G I L G V I L L L L P I V I G Q D R Y G S R L W I S F G P F T F Q P G E I A K I A I T L F L A F Y L A L N R E A L S V S M R S V G P F R I P R F K M L L P L F V M W G I S L I V V I F E R D L G S A L L F F V F F V I M L Y V A T G R A S Y V F V S V A - L L A I G G V I L Y H F F S H V Q T R V N I W L D P F K D P S G D G F Q I V Q S L Y S I A D G G L A G T G I D K G M P T - - - - - - - - - L I P V V E S D F I F S A I A E G N P T V I V H - - - - - S Q H D L D S W A - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - N L V A R C L Y M V G I - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - R K T D V F Q N S S G Y G M F T G G L G F Q - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - Y G A E R L G C L T V P A A A G - - - - - - - - - - N S K R Q I K F I S D G Q V A I V T G - - - - - - - - - - - G A R - - - - - - - - - - - - - - - - G I G - - - - - - - - - - - - - R G I A L T L A G A G A - - - - - - - - - - - - - - - - - - - - - N I L L A D L L D D A L D - - - - - - - - - - - A T A R E V R A L G R R A - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - A I A K V D V T Q A A - - - - - - - - - Q V D A M V A Q - - - - A L A D G R M A I - - - - - - - - - - - - - - A A K - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - Q G G A A A I R A Q G V N D I N E I K E V T K L P I I G - - - - - - - I I K R N Y D D S E I Y I T - - - - - - - - P T M K E V D E L L K T D - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - C E M I A L D A T K R K R P N G E N V K D L V D A I H A K - G R L A M A D G K E G V - - - - - - - - - - E E R L A E R - - - - - - - - - - - - - - - - G V S R R D F L K F C - - - - - T A I A V T M - G M G P - - - - - - - - - - - - - - - - - - - - - - - A F A P E V A R A L M - - - - - - - - - - - G P R R P S V V Y L H N A E C T G C S E S V L R A F E P Y I D T L I - - - L D T L - - - S L D Y H E T I - - - - - - - - - - M A A A G - D A A E A A - - - - - - - - - L E Q A V N S P H G F I A V V E A A A A A A A A * 4 3 7 5 - - - - - - - - - - - - - - 5 3 4 - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - 6 5 7 8 2 1 1 - 5 5 * 5 - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - 6 2 3 2 3 6 0 1 0 1 - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - - 0 4 7 3 3 0 5 3 5 3 2 3 - - - - - - - - - 0 0 0 1 1 2 8 3 - 0 0 0 7 8 5 9 G N P A V I + H + + + + + + + E D R D + A R L W I S F G P F T F Q P G E I A G I + + + + F L + F + L A L N R N + I A V T + R G V G + + + I + + + K + + + + L + + + + G I S L I + + I F A R + L D D A L + + + + F F V I M L Y V A T + R + + D V + L + + A + + + + + + G + + L F + F + + + + + T + + N I W L D + + K D P S + D + + + + + Q S L Y S I A D G G + A A + G L D A + + A + + A A G + + + K + L V D A + N S K + G F K A + A D
Проект JalView .
Таблица 4. Консервативные позиции при выравнивании заведомо негомологичных последовательностей
Позиции
Число
Процент
Абсолютно консервативные
2
0.6%
Абсолютно функционально консервативные
6
2%
Всего
295
100%