Эволюционные домены 1
Выбор домена
Для дальнейшней работы выбран домен RGS. Регуляторы каскада G белков (RGS) - активирют ГТФазу для альфа-субъединицы гетеромерного G-белка, тем самым инактивируя G-белок.
Pfam AC |
Pfam ID |
Описание |
Доменные архитектуры |
PF00615 |
RGS |
Регулятор каскада G-белков |
139 архитектур ,для которых доступно 4150 последовательностей. |
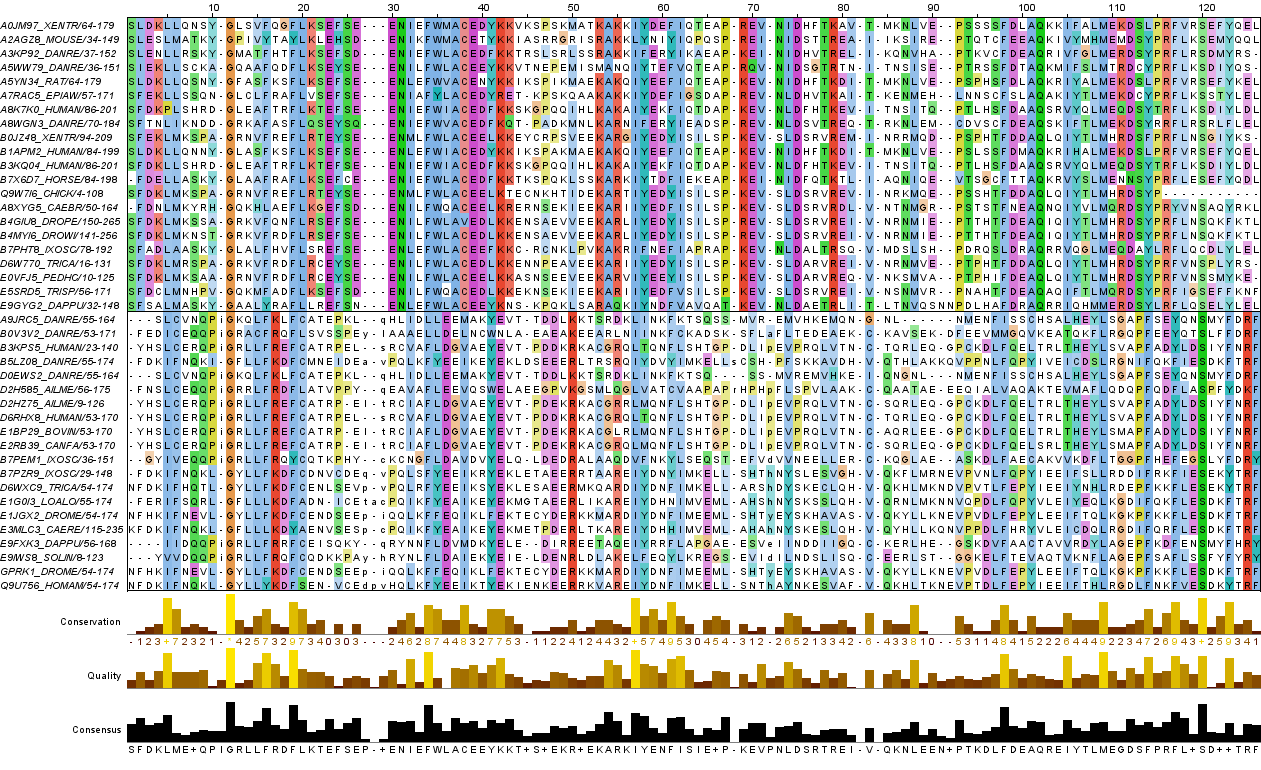
Загруженное с помощью JalView выравнивание из Pfam (File > Fetch Sequences по идентификатору PF00615) было раскрашено по консервативности (ClustalX и By conservation с порогом консервативности 10%).К последовательности ARBK1_BOVIN была добавлена 3D структура
1YM7 .
Выравнивание было сохранено как проект JalView в файле rgs.jar
Выбор доменных архитектур
С использованием скрипта swisspfam-to-xls.py и файла, содержащего информацию об архитектуре всех последовательностей UniProt (/srv/databases/pfam/swisspfam-2011.gz), была получена таблица с информацией об архитектуре последовательностей, содержащих домен RGS, на основе которой в Excel была составлена сводная таблица (лист PivotTable).
python swisspfam-to-xls.py -p PF00615 -i /srv/databases/pfam/swisspfam-2011.gz -z -o swisspfam_to_xls_out.txt
Затем в таблицу были добавлены колонки с информацией о таксономической принадлежности (лист taxonomy). Для этого по идентификаторам отобранных последовательностей в UniProt были получены AC (ID Mapping), по которым были получены файлы в формате UniProt (Retrieve), которые были использованы для работы скрипта uniprot-to-taxonomy.py. Также был добавлен столбец, содержащий информацию о длине доменов RGS и Pkinase (лист length).
python uniprot-to-taxonomy.py -i retrieved_seq_ids.txt -o uniprot_to_taxonomy_out.txt
Для дальнейшего изучения эволюции доменных архитектур, включающих домен RGS, были выбраны архитектуры:
Архитектура |
Число представителей |
|
RGS |
 |
1737 последовательностей |
RGS+Pkinase |
 |
369 последовательностей |
Описание домена Pkinase
AC |
PF00069 |
ID |
Pkinase |
Функция |
Протеинкиназы - группа ферментов, которая перемещает фосфатную группу на белки, сам процесс называется фосфорилированием. |
Число разных доменных архитектур с этим доменом |
4512 |
Число последовательностей |
114309 |
Число видов |
7677 |
Выбор таксона и представителей архитектур
В Metazoa были выбраны два подтаксона - филумы Chordata и Ecdysozoa(линяющие)
Представители выбраны при работе с таблицей (лист PivotTable со сводной таблицей, а весь список выбранных - лист selected_seqs)
Для каждой архитектуры было отобрано по 28 последовательности (по 14 из каждого таксона). Чтобы оставить в выравнивании нужные последовательности из двух групп (был использован скрипт filter-alignment.py).
python filter-alignment.py -i rgs.fa -m selected_ids.txt -o filter_alignment_out.faПолученное выравнивание
было загружено
в JalView и отредактировано (удалены пустые колонки). Затем в нём были выделены группы согласно архитектуре,
в каждой из них была выполнена раскраска последовательностей ClustalX, Conservation (порог на консервативность 10%). После удаления некоторых последовательностей (15), удаления пустых колонок и несодержательных C- и N- концевых участков
в конечном файле выравнивания JalView содержится 41 последовательность.
Эволюционные домены 2
Построение филогенетического дерева выборки последовательностей домена RGS
Зашифровка доменных архитектур и сравниваемых таксонов в названиях последовательностей:
| Архитектура | 1 | Архитектура №1 (RGS) |
| 2 | Архитектура №2 (RGS + Pkinase) | |
| Таксон | C | Chordata |
| E | Ecdysozoa |
выравнивание в fasta с модифицированными названиями последовательностей
Все деревья были графически построены в ITOL.
Строим филогетеническое дерево выбранных последовательностей, используя известные программы. Для всех деревьев ветви Chordata окрашены зеленым, ветви Ecdysozoa - фиолетовым. Листья раскрашены по архитектурам - красным цветом обозначена первая архитектура, синим - вторая.
Дерево, построенное UPMGA в JalView (скобочная формула )
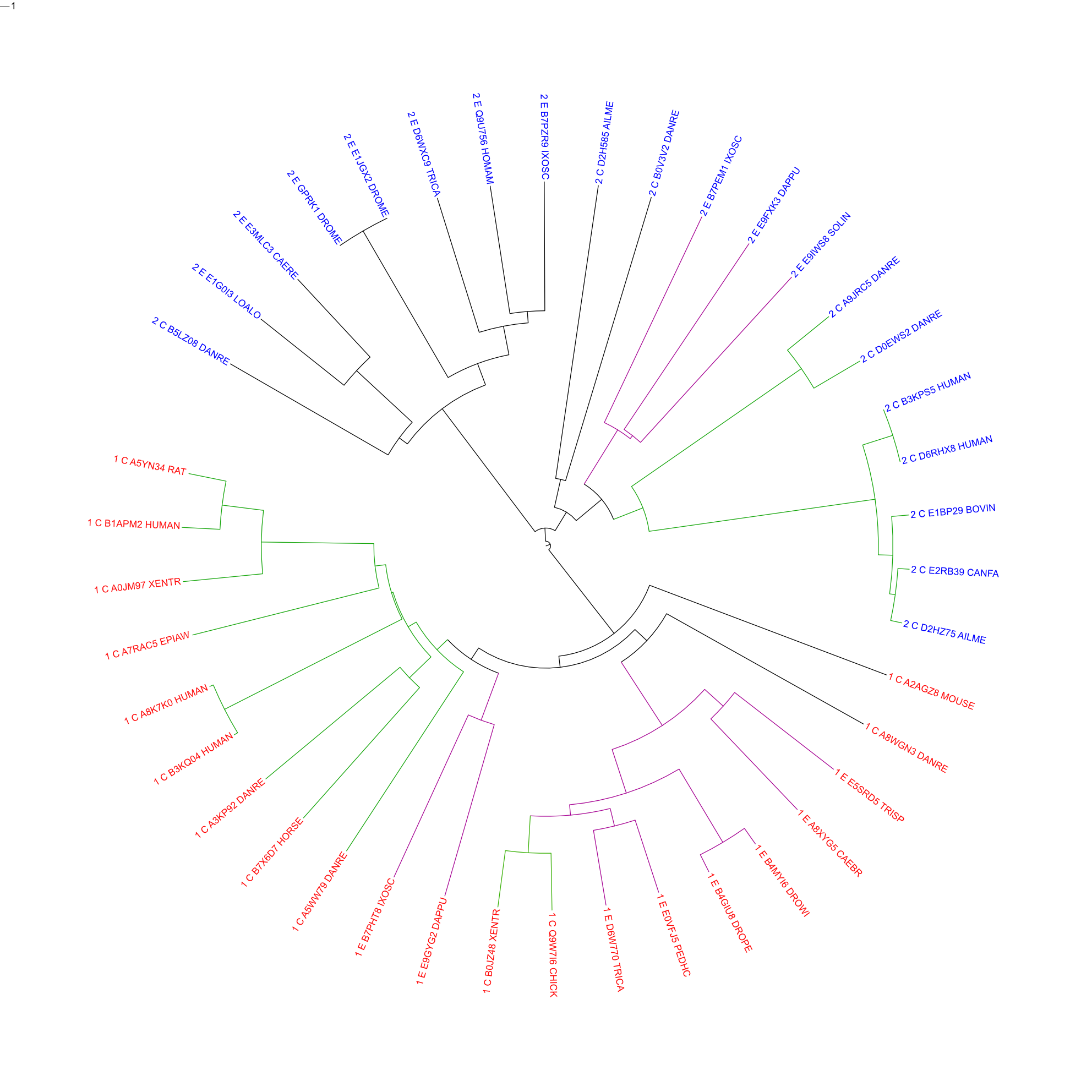
Дерево, построенное методом maximal parsimony fprotpars (скобочная формула ;изображение )
Укоренить дерево программой PHYLIP не удалось
Дерево, построенное с помощью NJ в JalView (скобочная формула )
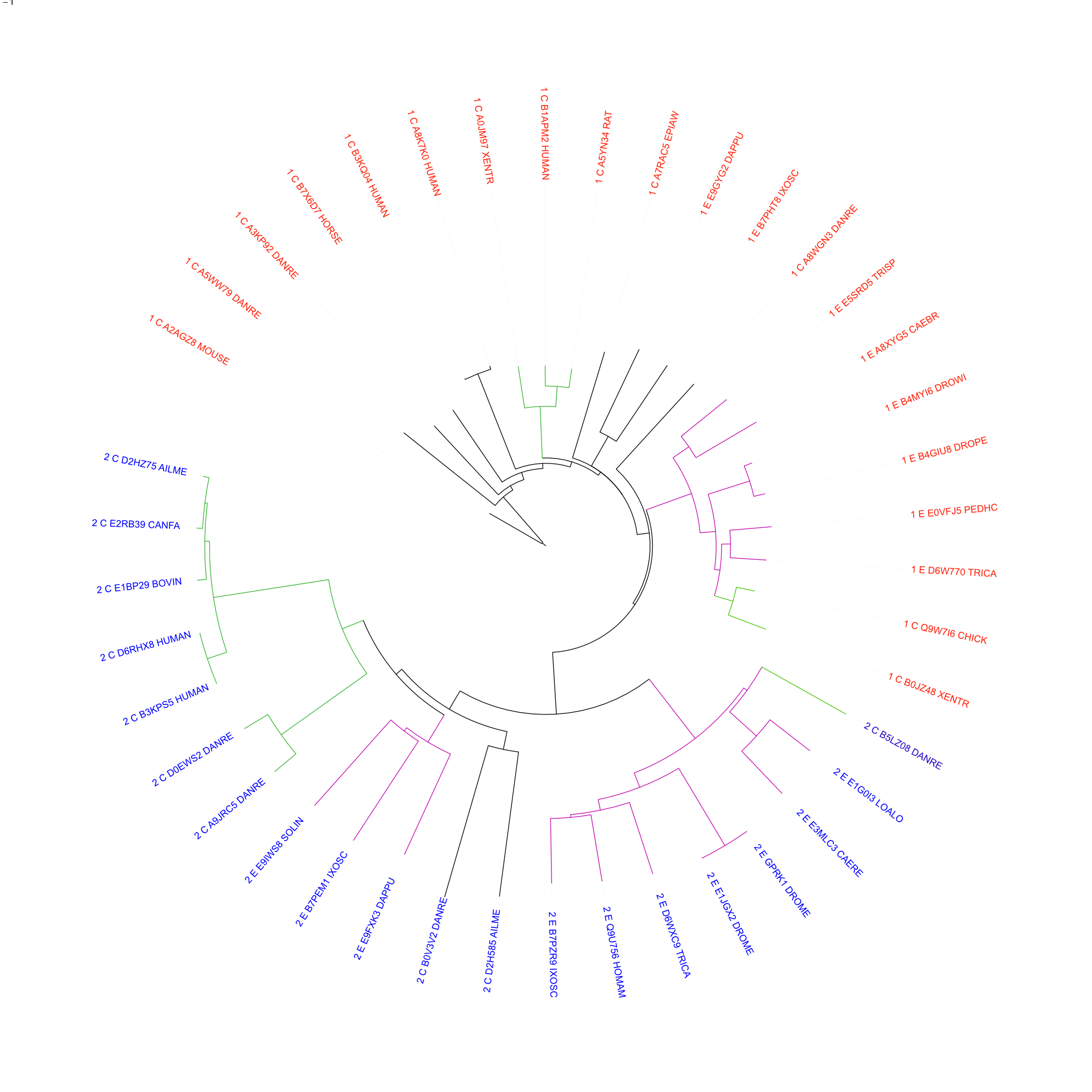
Последнее дерево неукоренено, поэтому укореним его программой PHYLIP ( скобочная формула )
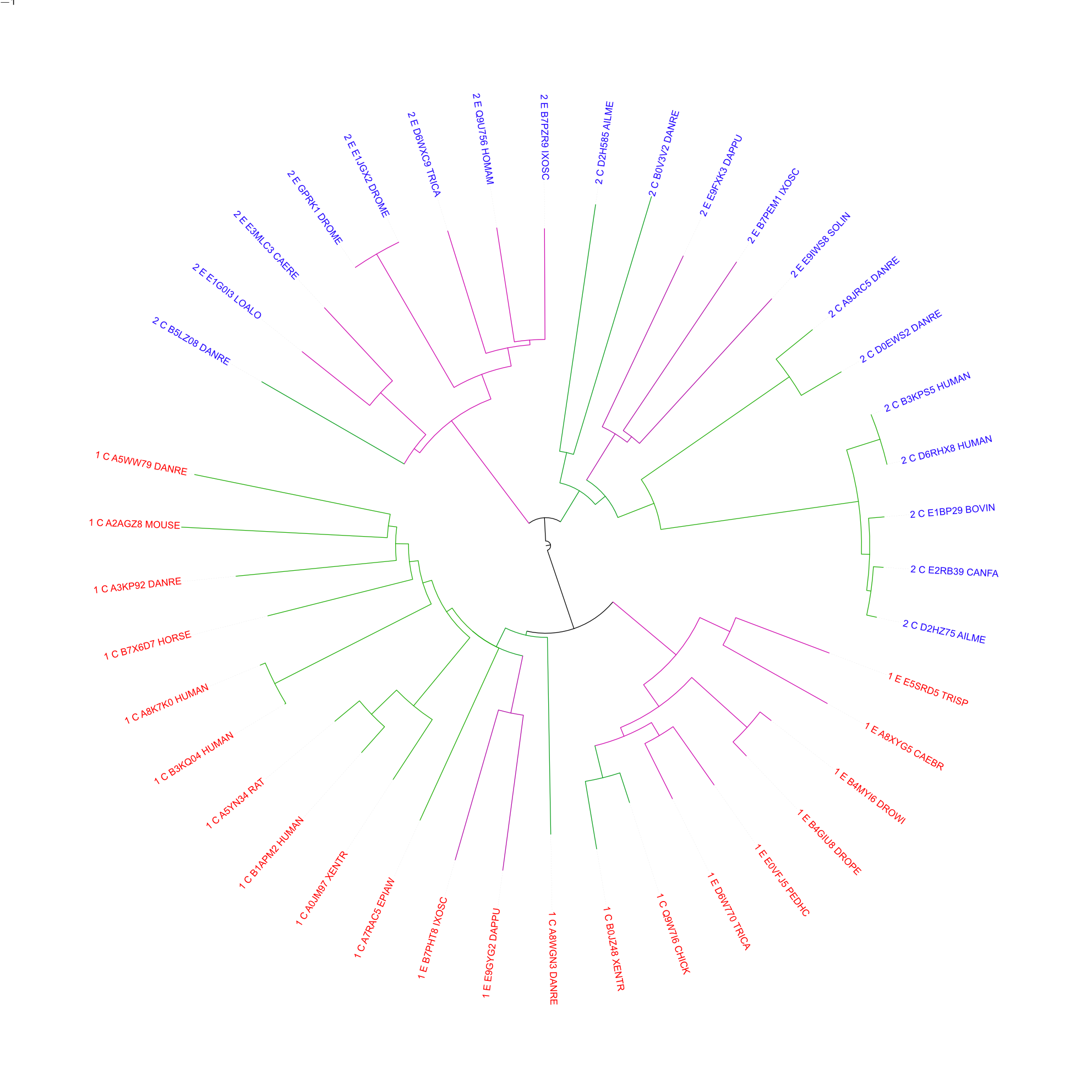
Укоренение программой PHYLIP происходит в то же место, в которое укореняет UPMGA - место разделения архитектур. Если деревья правильные, то домены эволюционировали независимо. Наиболее правдоподобным считаю дерево, построенное с помощью NJ в Jalview и укорененное программой PHYLIP. На нем достаточно хорошо видно разделение в каждой архитектуре на хордовых и линяющих (разделение не совсем чистое, но достаточно очевидное).