Анализ результатов поиска по профилю
Разделение выравнивания на две группы
По данным филогенетических деревьев решила разделить последовательности по принадлежности к разным доменным архитектурам - RGS архитектура и RGS+Pkinase. Далее они будут называться просто архитектуры 1 и 2 соответственно.
Построение профиля, отличающего последовательности домена из первой и второй архитектуры
Выравнивания, используемые далее для создания профиля: 1.msf (все последовательности, содержащие первую архитектуру) и 2.msf (все последовательности, содержащие вторую архитектуру).
seqret 1.msf msf::1_1.msf
noreturn -infile 1_1.msf -outfile 1_2.msf
pfw 1_2.msf > 1_weighted.msf
pfmake 1_weighted.msf /usr/share/pftools23/blosum62.cmp > 1.prf
Те же самые операции были повторены для файла 2.msf
Далее был создан файл со всеми последовательностями в формате fasta
Поиск по профилю в исходных последовательностях
Далее, проведем cам поиск по профилю в исходных последовательностях. Порог веса поставим маленьким (-C 0.0), чтобы все последовательности оказались в выдаче.
pfsearch –C 0.0 –f 1.prf all.fa | sort -nr > 1_scores.txt
pfsearch –C 0.0 –f 2.prf all.fa | sort -nr > 2_scores.txt
Результаты поиска при помощи полученных профилей по исходным последовательностям. По данным из файлов 1_scores.txt и 2_scores.txt Таблица Excel (листы RGS, RGS+Pkinase).
Построение ROC-кривой по полученным данным и анализ результатов поиска
Отметили в таблице последовательности, которые действительно принадлежат профилям. Заметила, что
среди результатов поиска некоторые последовательности встречаются несколько раз (по профилю находятся разные участки последовательности)
при этом второе вхождение последовательности в результаты имеет вес значительно меньше, чем первое.
Для последующего анализа все последующие вхождения не учитывались (отмечены цветом в Excel файле).
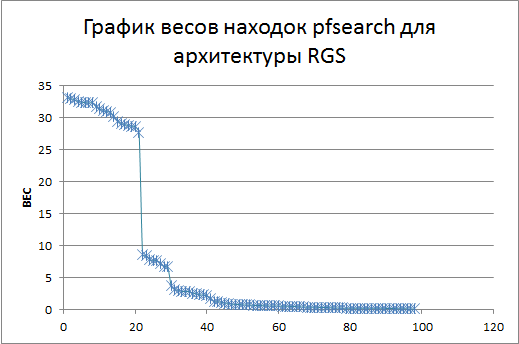
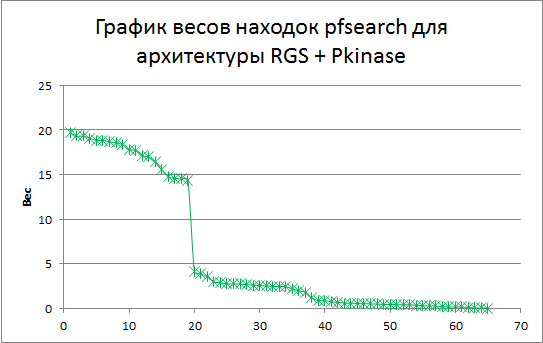
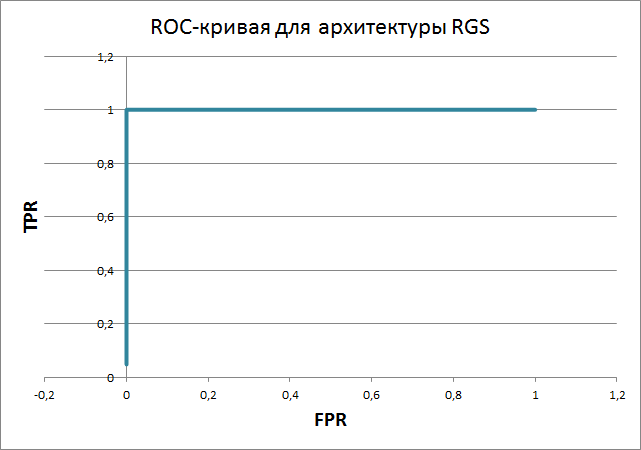
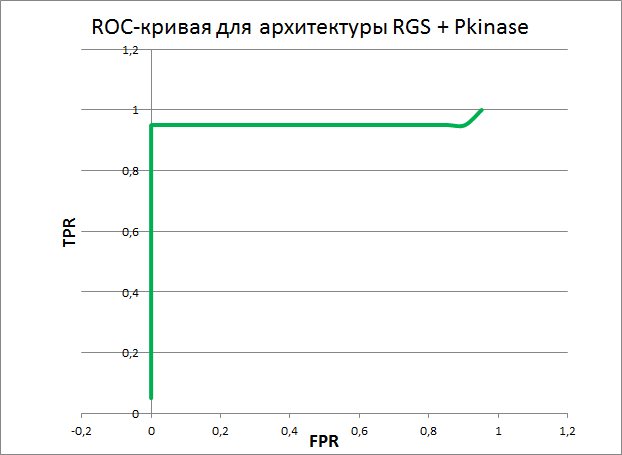
По полученным данным, сохранённым в Excel файле, были построены ROC-кривые и графики весов находок pfsearch для каждой из групп последовательностей:
ROC-кривые:
По графикам для нормализованного веса можно установить порог - 27 для первой архитектуры (RGS) и 14 для второй архитектуры (RGS+Pkinase)
Ошибки первого и второго рода для этих порогов для первой архитектуры равны нулю, а для второй архитектуры ошибка первого рода равна 1(5%), а второго - нулю. В принципе, по этим профилям можно различать последовательности RGS домена в двух архитектурах - RGS и RGS+Pkinase. 5% ошибка первого рода вызвана последовательностью E2RB39_CANFA, которая имеет очень маленький вес и очень маленькую длину в профиле. Также эта последовательность имеет маленький вес и длину в "true negative" в профиле для первой архитектуры. Как мне кажется, тут что-то не то с последовательностью (может, какой-нибудь фрагмент, в который наш домен почти не попал).
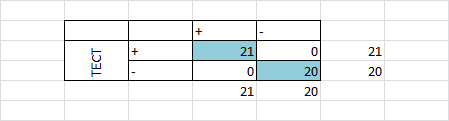
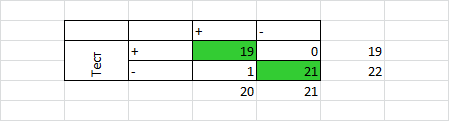
Таблица Excel