
Контрольная работа №2
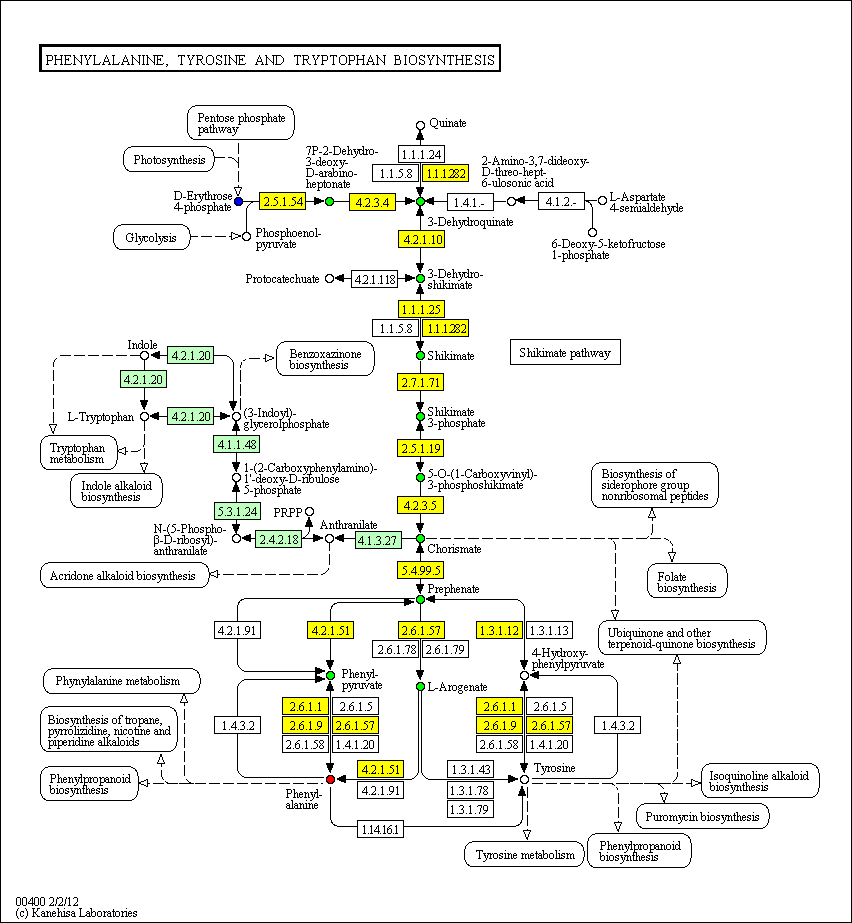
Нужно было найти путь от пентозофосфатного пути до фенилаланина. Самый
короткий путь, который я нашел, был от эритрозо-4-фосфата. Ниже
приведена картинка, на которой выделены желтым ферменты, которые
участвуют в этом пути.
Список генов E.coli приведен в этом файле, во
втором столбце направление гена,
в третьем начало оперона, в четвертом
начало первого гена в опероне.
У некоторых оперонов было 2 начала, в одному случае я записал их
отдельно(т.к. слишком далеко друго от друга располагаись)
Я скачал геном E.coli из базы данных геномов ncbi, и использую тот
файл с генами и свой скрипт на питоне, вытащил
нужные последовательности.
В задании велелось посмотреть -200:0 апстрим, но т.к. сайты регуляции
могут находиться и до начала оперона и после, я решил попробовать
взять разные последовательности, например -200:0 от начала первого
гена и 150:100 от начала оперона
На МЕМЕ все находки разные:
- -200:0 апстрим оперона
- -200:0 апстрим первого гена
- -150:100 участок от начала оперона
Так как большинство из тех, кого я
спросил, брали -200:0 апстрим от начала первого гена в опероне, я тоже
решил рассмотреть подробнее этот вариант. К тому же на Гиббс-Сэмплере
находки были только в двух случаях: при
recursive sampler и апстриме гена и
при motif sampler и -200:0 апстриме
оперона
Мотив из Гиббс-Сэмплера частично совпал с мотивом №2 из MEME, но MEME
нашел этот мотив только в 2 последовательностях, а гиббс почти во всех
MEME:

Gibbs-Sampler:

Далее я взял 2 бактерий: Pseudomonas aeruginosa и Vibrio cholerae
Скачал их геномы, вытащил апстримы ортологов гена aroL(следовательно и
aroK). И опять сделал
поиск мотивов.
В гиббс-сэмплере ничего не нашел в апстриме первого гена оперона, в
МЕМЕ нашел новый мотив.




© Almukhametov Azat






