| Главная | Семестры | Проекты | Обо мне |
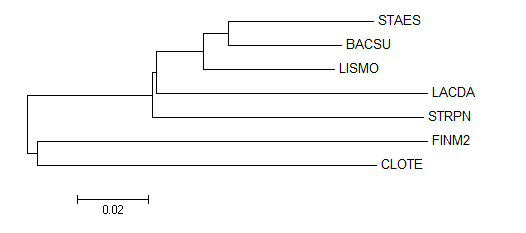
1. Построение дерева по нуклеотидным последовательностям
Задача: Построить филогенетическое дерево тех же бактерий, что в предыдущих заданиях, используя последовательности РНК малой субъединицы рибосомы (16S rRNA).
| Название | Мнемоника | AC записи EMBL | Начало | Конец | Последовательность |
| Bacillus subtilis | BACSU | AL009126 | 9810 | 11364 | + |
| Clostridium tetani | CLOTE | AE015927 | 8715 | 10223 | - |
| Finegoldia magna | FINM2 | AP008971 | 197837 | 199361 | + |
| Lactobacillus delbrueckii | LACDA | CR954253 | 45160 | 46720 | + |
| Listeria monocytogenes | LISMO | AL591974 | 37466 | 39020 | + |
| Staphylococcus epidermidis | STAES | AE015929 | 1598006 | 1599559 | - |
| Streptococcus pneumoniae | STRPN | AE005672 | 15450 | 16784 | + |
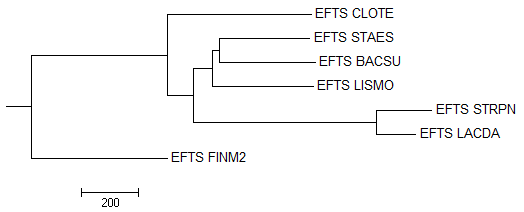

2. Построение и анализ дерева, содержащего паралоги
Достоверные гомологи белка CLPX_BACSU ищем с помощью программы blastp,
воспользовавшись файлом на диске Р proteo.fasta.
На выходе получили файл.
Теперь выбираем белки нужных нам бактерий, их почему-то получилось маловато
(файл).
Создали лист файл, чтобы получить последовательности и воспользовались
командой seqret, после выровняли последовательности с помощью команды muscle (файл).
На выходе получили выравнивание, которое открыли через программу MEGA и
реконструировали дерево методом "Minimum-Evolution" (картинка для
"Bootstrap consensus tree"):
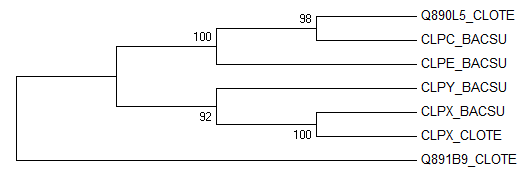
Паралоги:
CLPC_BACSU & CLPE_BACSU;
CLPY_BACSU & CLPX_BACSU.
Ортологи:
CLPX_BACSU & CLPX_CLOTE;
Q890L5_CLOTE & CLPC_BACSU.