|
|
Анализ качества и очистка чтений
Для анализа качества чтений мне был дан один из файлов с чтениями генома Arabidopsis thaliana. Для этого я использовала
программу FasqQC, установленую на сервере kodomo. Ссылка на результат
тут.
Для jчистки чтений я использовала все тот же файл и программу Trimmomatic. Все файлы с примерами адаптеров я поместила в
один (adapters.fa). Cтрока запроса:
java -jar /usr/share/java/trimmomatic.jar SE -phred33 Ath_tae_CTTGTA_L003_R1_003.fastq result.fastq
ILLUMINACLIP:adapters.fasta:2:7:7 TRAILING:20 MINLEN:50
Затем снова с помощью FasqQC я получила
отчет.
Приведем несколько результатов очистки. Сравним Per base sequence quality. Хорошо видно, что Trimmomatic удалил
хвостовые нуклеотиды с качеством ниже 28, среднее значение качества нуклеотидов выросло. Это хорошо заметно для концевых ридов.
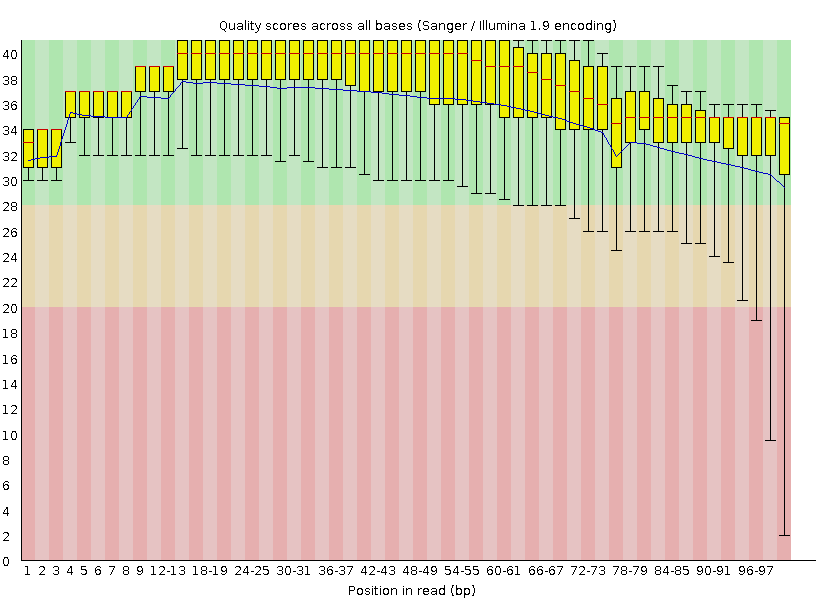
Рис 1. Per base sequence quality до очистки.
|
|
.png)
Рис 2. Per base sequence quality после очистки.
|
Еще одно отличие, бросающееся в глаза - это графики Sequence length distribution. На самом деле тут отличия почти нет.
Сначала длины всех ридов были 101, а после очистке появили (по причинам описанным выше) и более короткие, однако по второму графику
видно, что доля их мала.
.png)
Рис 1. Sequence length distribution до очистки.
|
|
.png)
Рис 2. Sequence length distribution после очистки.
|
|

.png)
.png)
.png)