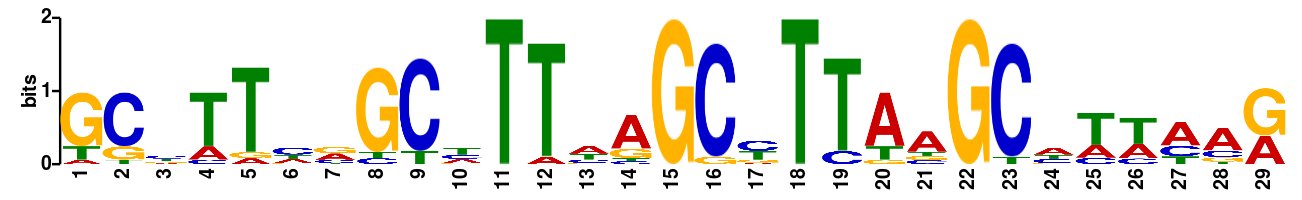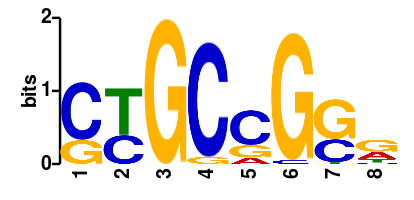|
|
Поиск сигналов. Последовательности Шайна-Дальгарно
Задание 1.
Я нашла данные по геному моей бактерии (Cytophaga hutchinsonii, штамм ATCC 33406) с помощью FTP-сервиса NCBI. Из предложенных
файлов нужны с расширением .fna (полный геном) и с
расширением .gff (хромосомная таблица).
Дальше мне нужно было создать файл с участками [-35;0] для каждого гена. Для этого я написала два скрипта.
Первый выбирает номера нужных участков из хромосомной таблице
в новый файл. Затем
с помощью команды:
seqret @NC_008255_num_list seqlist.fasta
я получила файл с нужными последовательностями, которые я
переименовала с помощью второго скрипта в порядковые номера.
(файл.) Всего получилось 3785 последовательностей.
Так как программа MEME не принимает на вход больше 1000 последовательностей, я взяла первую тысячу генов. Последовательность
Шайна-Дальгарно короткая и сигнал не очень сильный. Сначала я запустила без ограничений, нашлась длинная последовательность, в
которой ничего похожего на консенсус Шайна-Дальгарно, что меня удивило, так как этот мотив занимает почти всю длиу участков. Лого
последовательности на рис 1.
(Результат работы MEME.)
Поэтому я запустила MEME еще раз с ограничением по длине 6-10 нуклеотидов в сигнале.
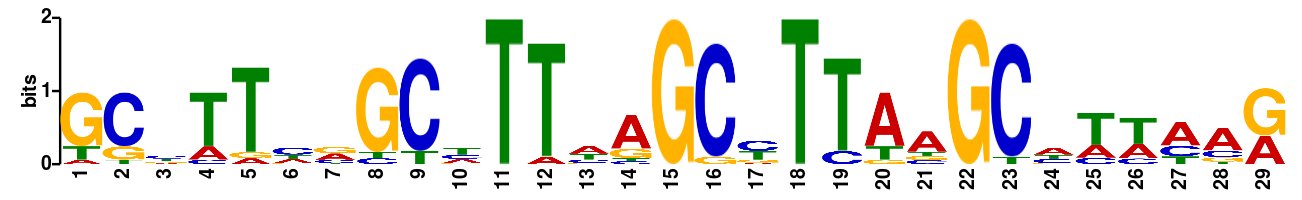
Рис 1. Лого мотива, найденного программой MEME без ограничений.
Программа MEME нашла совершенно иной сигнал длинной в 8 нуклеотидов (рис 2).
Результат работы программы.
Видно, что это GC-богатый участок, что часто несет сигнальную функцию, но на консенсус Шайна-Дальгарно все еще не похож.
Однако в статье сказано, что у бактерий группы, в которую входит
и моя, наблюдается замена последовательности ШД на GC-богатый участок, который у меня и нашелся.
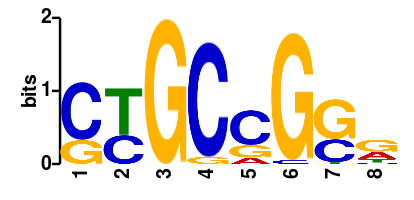
Рис 2. Лого мотива, найденного программой MEME с ограничением в 6-10 нуклеотидов.
Найденный мотив и файл со всеми последовательностями был подан на вход программе MAST. В результате было найдено 52
последовательности с достоверным совпадением с найденным мотивом (для первого мотива). Результат работы программы MAST
тут. А для второго мотива -
17. Результат работы программы MAST
тут.
|