Укоренение в среднюю точку
С помощью программы retree было укоренено в среднюю точку дерево, построенное
по алгоритму neighbor-joining на предыдущем
занятии
Укоренение в среднюю точку нельзя сделать
с деревьями, построенными методом максимальной экономии, так как этот метод не оценивает
длины ветвей. А с деревом, построенным алгоритмом UPGMA не имеет смысла это делать,
так как программа выдает уже укорененное дерево
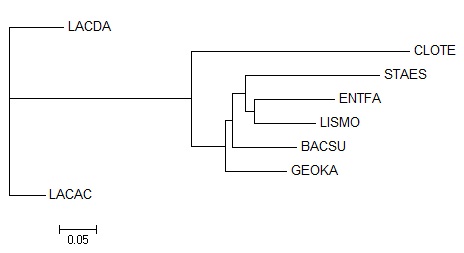 Дерево, полученное
алгоритмом neighbor-joining
Дерево, полученное
алгоритмом neighbor-joining
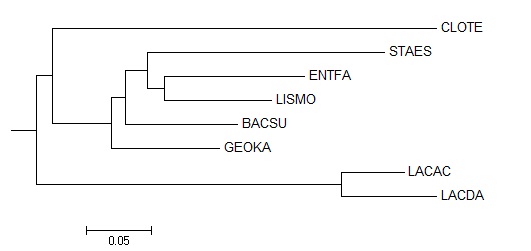 Укорененное дерево
Укорененное дерево
Укоренение произошло в ветвь {LACAC, LACDA} vs {CLOTE, STAES, ENTFA, LISMO, BACSU, GEOKA}; Это не соответствует укоренению в "правильном дереве" (см. предыдущее занятие)
Использование внешней группы
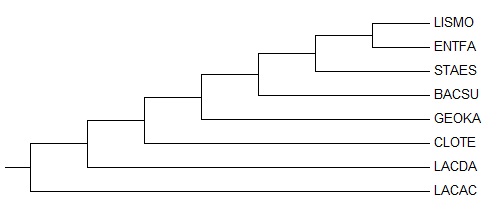 Неукорененное дерево, полученное программой fprotpars
Неукорененное дерево, полученное программой fprotpars
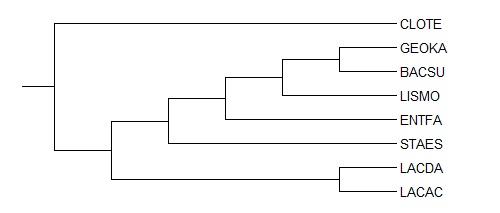 Укорененное дерево
Укорененное дерево
Укоренение произошло в тривиальную ветвь {CLOTE} против всего остального. "Правильное" дерево так же укоренено в эту ветвь.
Бутстрэп
+---------------STAES
+--71.6-|
| | +-------LISMO
+--43.2-| +--45.6-|
| | +-------ENTFA
| |
+-------| +-----------------------BACSU
| |
| | +-------LACDA
| | +--100.0-|
| +----------85.0-| +-------LACAC
| |
| +---------------CLOTE
|
+---------------------------------------GEOKA
Species in order:
1. GEOKA
2. BACSU
3. LISMO
4. ENTFA
5. STAES
6. CLOTE
7. LACDA
8. LACAC
Sets included in the consensus tree (Ветви, включенные в консенсусное дерево)
Set (species in order) How many times out of 100.00
......** 100.00
.....*** 85.00
..***... 71.60
..**.... 45.63
.****... 43.20
Sets NOT included in consensus tree: (Ветви, невключенные в консенсусное дерево)
Set (species in order) How many times out of 100.00
...**... 27.17
..****** 26.07
.*...*** 14.40
..***.** 14.00
.****.** 13.00
...*..** 9.00
...**.** 7.00
..*.*... 6.83
.***.*** 6.53
..**..** 6.50
....*.** 6.00
....**** 5.53
...***** 4.67
...*.*** 3.33
..**.*** 1.33
.**..*** 1.20
.***.... 1.00
.**..... 1.00
Дерево, полученное данным образом полностью совпадает с деревом, полученным программой fprotpars на предыдущем занятии, и, точно так же, имеет только одну общую ветвь с "правильным" деревом:
{LACDA, LACAC} vs {CLOTE, LISMO, ENTFA, STAES, BACSU, GEOKA};
Причем, эта ветвь есть в каждом из 100 деревьев, поданных на вход программе fconsense, что говорит о высокой консервативности белка LEPA у бактерий Lacda и Lacac.
Среди ветвей, не получивших большинства, есть общие ветви:
{BACSU, GEOKA} vs {LISMO, STAES, LACDA, LACAC, ENTFA, CLOTE}; Поддержка - 26.07
{LACDA, LACAC, ENTFA} vs {BACSU, GEOKA, LISMO, STAES, CLOTE}; Поддержка - 9.00
{BACSU, GEOKA, LISMO, STAES} vs { LACDA, LACAC, ENTFA, CLOTE}; Поддержка - 3.33
То есть, все остальные ветви правильного дерева не получили большинства, однако, на каких-то стадях программы появлялись. Это может говорить об отличающихся путях эволюции белка LEPA и моего "набора" бактерий.
