Докинг низкомолекулярных лигандов в структуру белка
Цель данного занятия ознакомится с возможностями докинга
низкомолекулярного лиганда в структуру белка.
Мой объект- лизоцим, структуру которого я построила на прошлом
занятии.
4-ая модель.
- Со страницы структуры 1lmp была скачана SMILES структура NAG.
- Далее, с помощью obgen была построена 3D структура сахара в формате pdb.
obgen nag.smi > mol-файл babel .. из mol-файла в pdb-файл
- Были созданы файлы nag.pdbqt и
04.pdbqt, где второй файл - это 4-ая модель с прошлого занятия,
у которого удалена молекула лиганда. Эти файлы были созданы с помощью команд:
prepare_ligand4.py -l nag.pdb prepare_receptor4.py -r 04.pdb
- После этого был создан файл с параметрами докинга vina.cfg,
в котором указывается область структуры белка, в котором идет поиск места связывания.
Это место было задано как куб с неким центром. Центр был определен между атомами,
использовавшимися в прошлом занятии для построения комлекса белка с лигандоим с помощью
команды pseudoatom :
pseudoatom pseudo, (/04///*32/OE2 or /04///*102/O or /04///*60/OH)
- Проведение докинга:
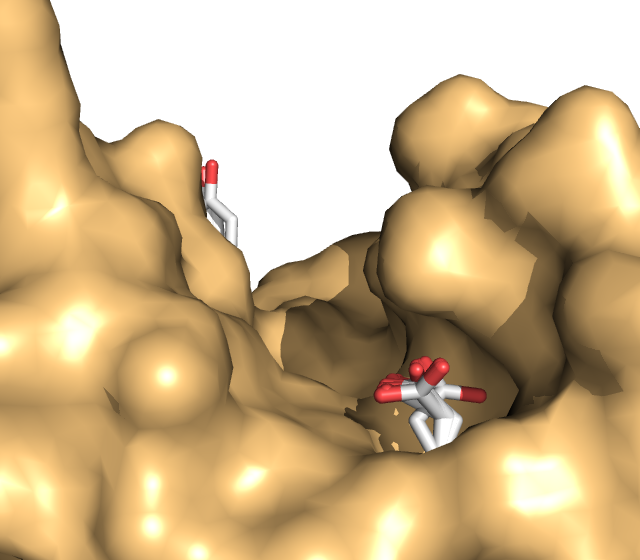
vina --config vina.cfg --receptor 04.pdbqt --ligand nag.pdbqt --out nag_04.pdbqt --log nag_04.logПолученные файлы: nag_04.log, nag_04.pdbqt. - Три лучших расположения:
1 -6.2 kcal/mol 2 -5.8 kcal/mol 3 -5.5 kcal/mol
Геометрическая разница между 2-ым и 1-ым: 2.147, между 3-им и 1-ым: 1.738.png)
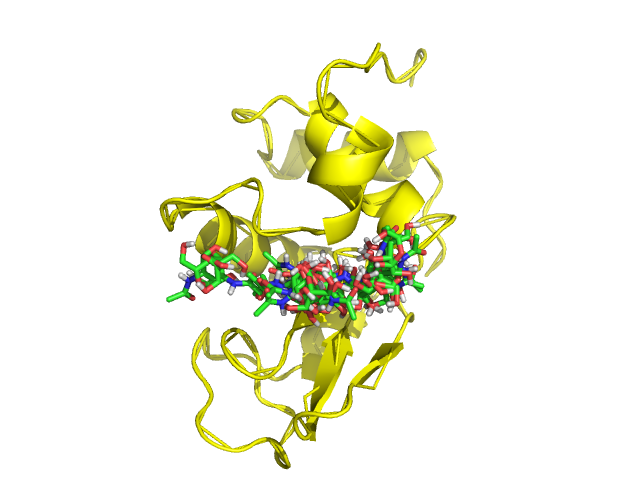
Видно, что лиганд в различные моменты времени занимает позиции внутри места связывания. - Затем я провела докинг, расматривая подвижность некоторых боковых радикалов белка.
Разбила белок на подвижную и неподвижную части:
python /usr/share/pyshared/AutoDockTools/Utilities24/prepare_flexreceptor4.py -r 04.pdbqt -s GLU32_TYR60_ALA102
Для подвижной части я выбрала те же аминокислоты, кторые использовала при позиционировании лиганда на прошлом занятии. Проведение докинга:vina --config vina.cfg --receptor 04_rigid.pdbqt --flex 04_flex.pdbqt --ligand nag.pdbqt --out nag_04_2.pdbqt --log nag_04_2.log
- Три лучших расположения:
1 -5.9 kcal/mol 2 -5.4 kcal/mol 3 -5.3 kcal/mol
Геометрическая разница между лучшими изображениями: 2-ым и 1-ым: 1.267, 3-им и 1-ым: 1.991.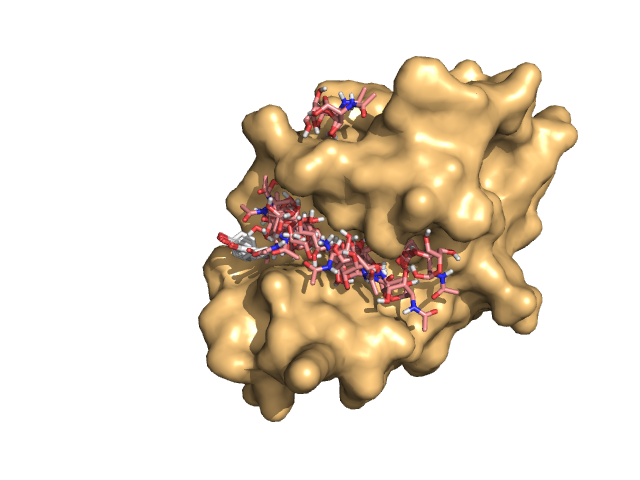
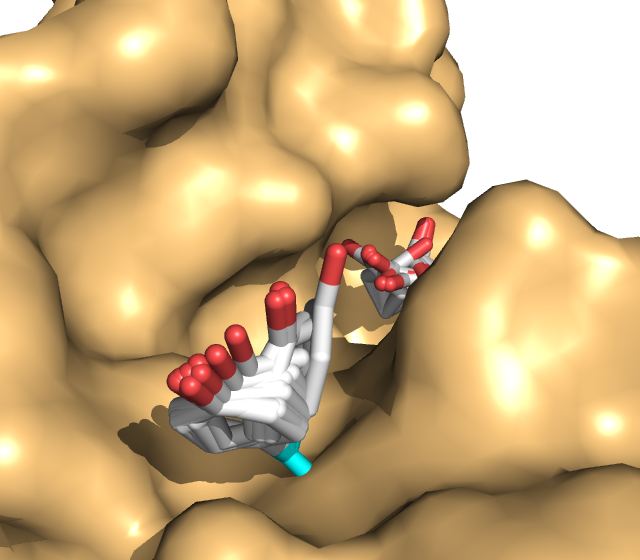
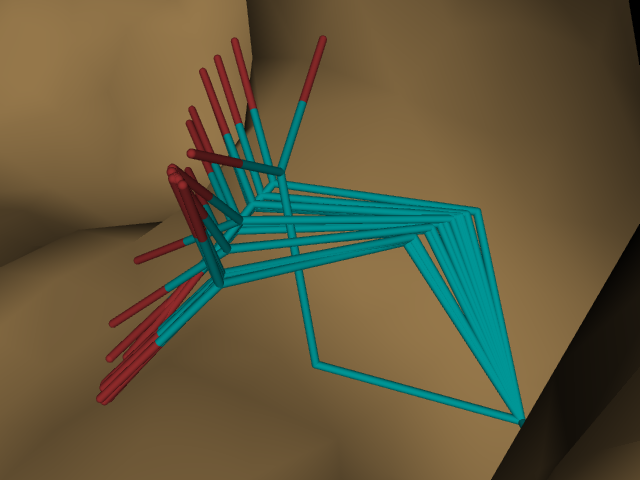
 Интересно, что на паре слайдов сахар "выпригивает" из активного центра. Возможно,
это связано с тем, что остаток перехоит границы своего "куба".
Не показана аминокислота аланин, возможно, из-за того, что не изменяет своего положения
во время докинга. Для тирозина и глутамина виден широкий диапазон вращения.
Интересно, что на паре слайдов сахар "выпригивает" из активного центра. Возможно,
это связано с тем, что остаток перехоит границы своего "куба".
Не показана аминокислота аланин, возможно, из-за того, что не изменяет своего положения
во время докинга. Для тирозина и глутамина виден широкий диапазон вращения.
- Визуально 6 модель кажется наиболее близкой по расположению к тому, что
получилось при моделировании. Энергия этого расположения - -5.3 kcal/mol
-
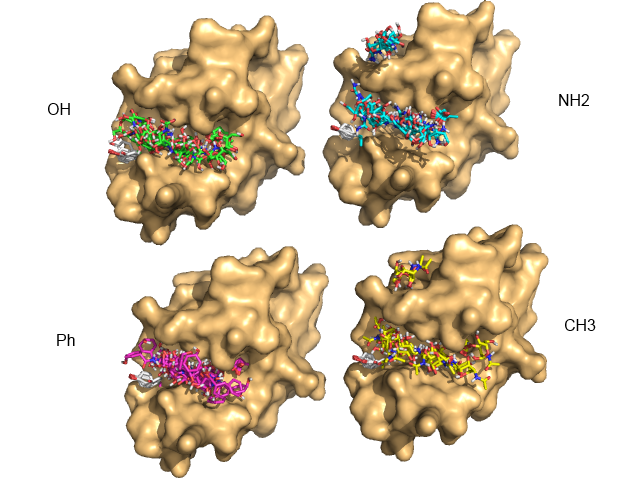
NAG содержит в себе СH3C(=O)NH группу. Было создано 4 лиганда, у которых заменен
метильный радикал, и проведен обыкновенный докинг:
Заметно, что при увеличении радикала энергия связи падает.Радикал CH3 OH NH2 H Ph Значение энергий трех лучших положений (kcal/mol) 1 -6.2 -6.0 -6.4 -5.7 -7.3 2 -5.8 -5.5 -5.7 -5.5 -6.7 3 -5.5 -5.4 -5.6 -5.3 -6.1 -
Далее был проведен докинг с подвижными радикалами для новых 3 лигандов (кроме Н).
Вот, что получилось:
- Подвижность GLU

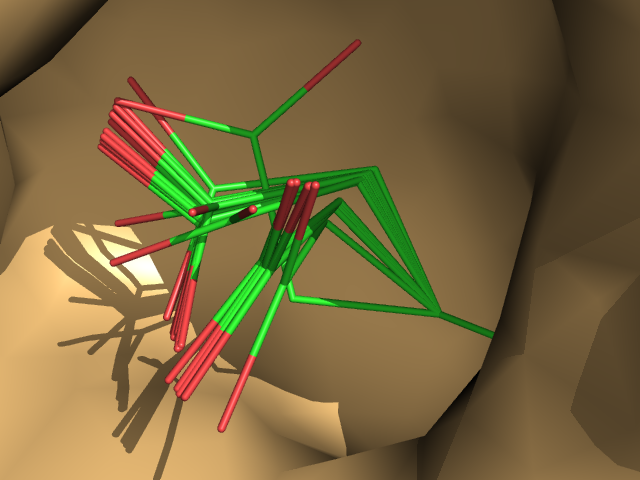
OH NH2 Ph - Подвижность TYR

OH NH2 Ph
- Подвижность GLU
- Общая картинка